
简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
生信碱移来自微软研究院的研究者在不进行任何微调的前提下对Geneformer和scGPT进行了系统性评估,发现这些单细胞大语言模型在零样本情况下的性能表现甚至不如简单方法。基于目前积累的大量单细胞数据,多项单细胞大语言模型被陆续提出。大部分研究者希望借助如scGPT和Geneformer这类预训练大模型,实现细胞类型注释、基因表达预测等多种分析的“自动化”。另外,因为这些大模型都在跨物种/细胞类型

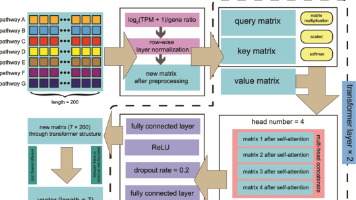
第四步,将多头连接后的矩阵依次通过全连接层、ReLU 层、随机 dropout 层、另一全连接层与另一随机 dropout 层,最后再进行层归一化。a.维恩图基于以 cGAS-STING 为中心的通路中按 AUC 选取的特征,展示用于预测抗 PD-1/PD-L1 应答结局的特征筛选结果。作者先在 TCGA-LIHC 中计算了 cGAS–STING 通路的 ssGSEA 分数,随后与 50 个 ha
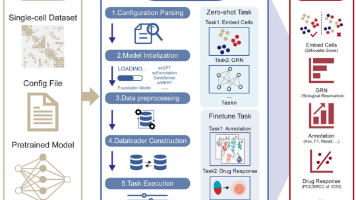
单细胞 RNA 测序(scRNA-seq)通过实现高分辨率转录组分析,彻底改变了传统分子生物学。已经开发了几种基础模型来分析大规模的单细胞测序数据,如 scBERT, Geneformer, scGPT 和 scFoundation。然而,这些模型不仅在架构设计和预训练策略上表现出一定差异,而且数据集大小和参数数量也有所不同。
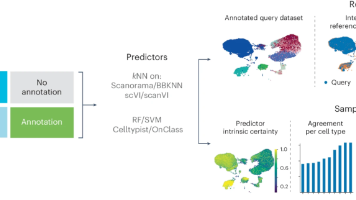
细胞类型注释是单细胞RNA测序(scRNA-seq)数据分析中的关键步骤。目前注释方法的金标准依赖于人工专家,需要手动将每个细胞簇中高表达的基因与文献中的经典细胞类型标记基因进行比对。尽管如此,这一流程及其耗时,而且需要专业的生物知识。随着测序成本的下降,当数据集规模扩大到数百万个来自不同组织的细胞,手动注释的方法已变得难以实现。
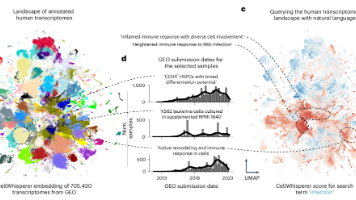
以6个数据集约10万细胞的人胚scRNA-seq为对象,作者把Carnegie阶段的文本描述变成查询,计算每日龄的平均分数,得到与受精后时间吻合的阶段性轨迹;②对圈选细胞可直接在聊天框请求“描述这些细胞”,系统基于选区的平均嵌入生成包含细胞类型、组织/发育信息与代表基因的文本报告;简单来讲,CellWhisperer 通过把转录组信号和自然语言文本放到同一个嵌入空间,再配一个能看得懂嵌入的聊天模型
调用硅基流动deepseekapi

GeneGPT的核心思想是通过大语言模型(如GPT-3.5)对基因信息进行嵌入,从而生成能够反映基因功能和细胞特征的嵌入向量(其他方法则是需要使用大规模的单细胞数据进行预训练,GeneGPT 属于是触类旁通、借花献佛了)。这些基因嵌入向量能够帮助我们在单细胞转录组学研究中更好地表示基因和细胞的生物学信息,被进一步用于细胞级别的嵌入表示。具体来说,基因嵌入通过提取NCBI基因数据库中的文本摘要,并利

TCGA 数据库基因表达数据的下载:最大的公开肿瘤数据库,可用于各种深度学习项目
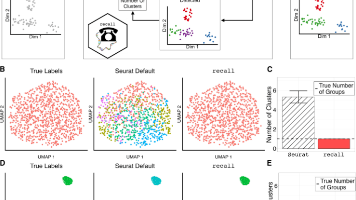
生信碱移Recall 通过人工生成随机的噪声假基因判断细胞聚类簇是否拆得过细,如果过细就自动往回合并或者调整分辨率,从而得到优化的聚类。单细胞 RNA 测序(scRNA-seq)能够分析包含数千至数百万个单细胞转录组图谱的数据集。主流分析流程 seurat 与 scanpy 一般包括以下三个大步骤:预处理(质控、归一化、选 HVGs、PCA)无监督聚类(在 PCA 空间构建的邻接图上 执行Louv
该研究通过搭建单细胞整合评估平台(scIB),在13个整合任务(含2个模拟任务、5个 scRNA-seq 任务和6个 scATAC-seq 任务)上。为检验方法的准确性、可用性和可扩展性,研究设计了14个性能指标从批次效应移除与生物学变异保持两大维度进行量化评估;同时,考虑了多种输出格式(嵌入、矫正矩阵、集成图等)及不同的预处理策略(含/不含缩放与高变基因筛选)。











