
简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
概述了几种主要的生物信息学数据库,包括核酸序列、蛋白质序列及其结构和专用生物路径数据库。文章从一级核酸数据库开始,详细介绍了GenBank、Ensemble和JCVI等数据库,这些数据库提供了广泛的原核和真核生物的遗传信息。然后是蛋白质数据库,从UniProtKB的基本序列信息到PDB的三维结构信息,以及如Pfam、Cath和SCOP2等二级蛋白质数据库的深入分析。最后,探讨了KEGG和OMIM等

来自Manolis Kellis教授(MIT计算生物学主任)的课《人工智能与机器学习》。本节课主要介绍了network和graph的知识。主要内容有网络和图的基础知识(网络类型、相关算法)、网络性质(motifs性质、中心性等)、特征向量、SVD奇异值分解、PCA、Sparse PCA、t-SNE等。我觉得讲的很好!短短一个半小时讲的比我大一学的线性代数有意思多了,很有启发,尽管我的线性代数很烂但

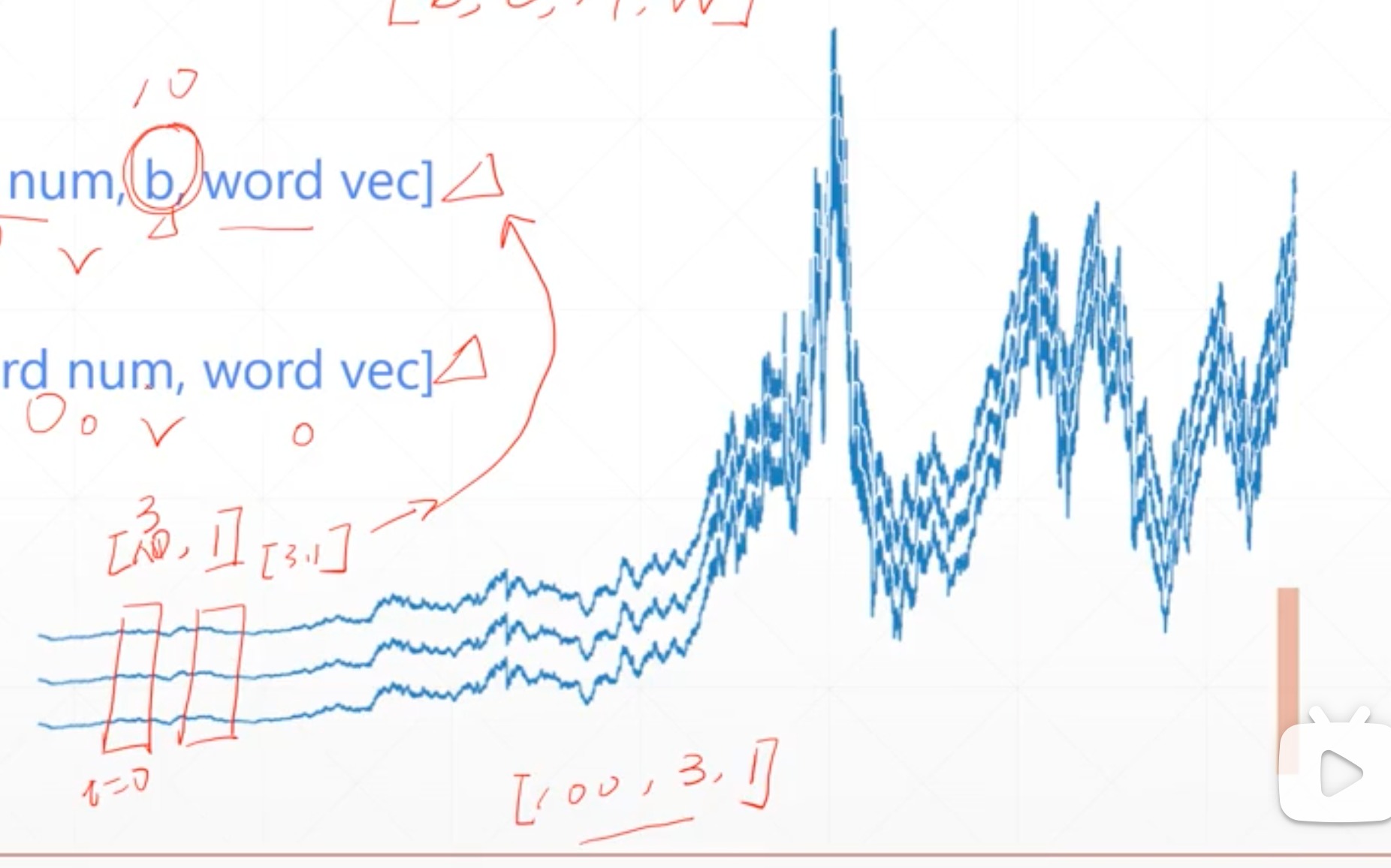
b站课程笔记 循环神经网络部分,理论讲的还挺通俗易懂的,虽然有时候把自己讲晕了,包括RNN和LSTM
数学建模清风课程笔记 topsis和用熵权法改进
第一节 点符号的制作方法1 ArcGIS的样式管理器使用2 制作一些点符号,如一个图中间一个字3 FontCreator使用4 修改己有的符号库第二节 线面符号制作1 简单线符号2 制图线符号和单位换算3 混列线符号4 标记线符号5 面符号制作6 查看已有符号库第三节 专题图1 单一专题2 唯一值专题3 数量渲染专题4 图表专题5 符号库在专题图中应用6 Mxd保存和打包7 复杂、高级专题和图层覆










