简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
生信分析全面向时代迈进,传统服务器已经越来越难以满足现在的数据分析需求。科研越来越快、数据越来越大,但算力却常常跟不上。数信院正式推出目前生信圈顶级配置:数信院 X3 服务器。
摘要: 生物信息学分析离不开Linux服务器,主要原因有三:一是高通量测序数据量庞大(单个样本可达几十GB),笔记本存储和算力不足;二是服务器配置(大内存、多核CPU、TB级存储)能高效完成比对、组装等计算密集型任务;三是主流生信工具(如BWA、GATK)多在Linux环境优化,命令行脚本更适合批量处理海量数据。服务器通过SSH远程连接,结合多用户管理功能,成为团队协作的高效计算平台。掌握Linu

AI工具正在改变生物信息学工作方式,三大主流模型各有优势:Claude擅长全流程分析脚本生成,GPT-5-Codex注重操作安全性控制,Gemini3在多模态绘图方面表现突出。测试显示,Claude对生物信息学理解最深,能直接生成可执行的Snakemake脚本;GPT-5-Codex提供安全模式选择;Gemini3在文献图表复现方面表现出色。这些工具使零代码完成生信分析成为可能,显著降低了分析门槛
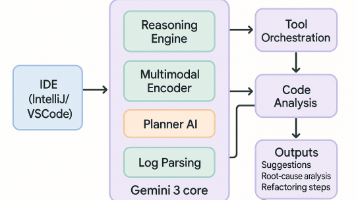
Google最新大模型Gemini3正在革新科研绘图流程,让SCI级论文配图实现"一键生成"。该模型具备多模态理解能力,可同时处理文本、图像和实验数据,支持Python/R/MATLAB等语言的代码自动生成与调试。在生物信息学领域,Gemini3能快速完成RNA-seq、ChIP-seq等分析流程,自动产出火山图、热图等专业图表,显著提升科研效率。部署在服务器上时,还能直接处理
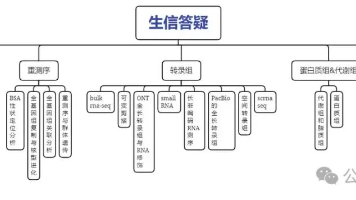
数信院提供专业生物信息学答疑服务,帮助科研人员解决代码报错、算法选择、数据分析等难题。服务特色包括:1)硕博团队支持,涵盖多组学领域技术指导;2)个性化解决方案,优化分析流程;3)快速响应机制。购买共享/独享服务器可获赠答疑服务,实现算力资源与技术指导的无缝衔接,助力科研突破。
【生信专属云服务上线】提供高算力集群,预装Seurat等工具,支持单细胞分析等复杂场景,实现百万级数据秒加载。采用银行级加密和多节点备份保障数据安全,弹性算力可将3天任务缩短至4小时,显著提升科研效率。官网现已开放,助力生信研究加速突破。

数信云创作助手迎来重磅升级,推出多项创新功能:新增GPT4O、Claude3.5等顶尖AI模型;上线学术辅助工具包括思维导图生成、文献分析和图像识别;引入DALL·E-3文生图模型;优化用户界面支持历史对话检索和算力监控。特别推出GPTCelltype工具,基于GPT4实现单细胞RNA分群自动注释。访问新网址http://sxygptcloud.com:8080/即刻体验全面提升的智能创作服务。

Google最新大模型Gemini3正在革新科研绘图流程,让SCI级论文配图实现"一键生成"。该模型具备多模态理解能力,可同时处理文本、图像和实验数据,支持Python/R/MATLAB等语言的代码自动生成与调试。在生物信息学领域,Gemini3能快速完成RNA-seq、ChIP-seq等分析流程,自动产出火山图、热图等专业图表,显著提升科研效率。部署在服务器上时,还能直接处理

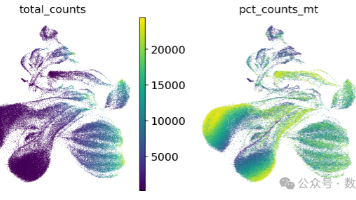
FindNeighbors 函数用于计算数据集中每个细胞的 k 个最近邻居,并可选构建共享近邻图(SNN),通过计算每个细胞和其 k 个最近邻居之间的邻域重叠(Jaccard 指数)来实现。作为科研创新的一部分,我们还为用户提供免费的AI技术服务,帮助您在数据分析、预测建模等方面提升研究效率,为您的科研成果加速赋能。4.海量实用脚本资源:即拿即用,效率翻倍。计算资源:如果计算资源有限,RPCAIn
摘要:本文介绍了单细胞RNA-seq数据分析中的细胞聚类方法。通过UMAP降维可视化评估数据质量后,重点讨论了基于KNN图和Leiden算法的聚类流程。文章比较了不同分辨率参数(0.25、0.5、1.0)对聚类结果的影响,指出较高分辨率会产生更多精细簇群。实验采用scanpy工具包实现,包含邻居图构建、UMAP可视化和多分辨率Leiden聚类等步骤,最终通过UMAP图展示不同参数下的聚类效果。该方