组学数据的真正价值?
过去几十年里,大量的高通量生物数据(其中组学数据占了很大部分)已在公共数据库中公开。数据以各种格式存在,涵盖了多种物种和研究领域,提供了对复杂生物系统的洞察;托管数据的公共数据库成了多功能资源。随着数据科学和人工智能在生物学中的应用不断推进,数据的潜在更大价值在于2次利用。重点评估了2次数据使用中的挑战,特别是针对公共数据库中可获取的人类胚胎肾细胞系的组学数据。出现的问题是不同领域的2次数据使用者
摘要
过去几十年里,大量的高通量生物数据(其中组学数据占了很大部分)已在公共数据库中公开。数据以各种格式存在,涵盖了多种物种和研究领域,提供了对复杂生物系统的洞察;托管数据的公共数据库成了多功能资源。随着数据科学和人工智能在生物学中的应用不断推进,数据的潜在更大价值在于2次利用。重点评估了2次数据使用中的挑战,特别是针对公共数据库中可获取的人类胚胎肾细胞系的组学数据。出现的问题是不同领域的2次数据使用者面临的障碍,涉及接受任何物种类型数据存储的平台和数据库。生物学中数据驱动研究的不断发展促使重新评估开放获取数据的管理和提交程序,以确保挑战不会阻碍数据利用而产生的新研究机会。本文旨在引起对数据报告中普遍存在的问题的关注,并鼓励数据所有者精心策划提交的数据,以最大化其不仅在当前研究中的影响,也在长期的数据集遗产中的影响。
材料和方法
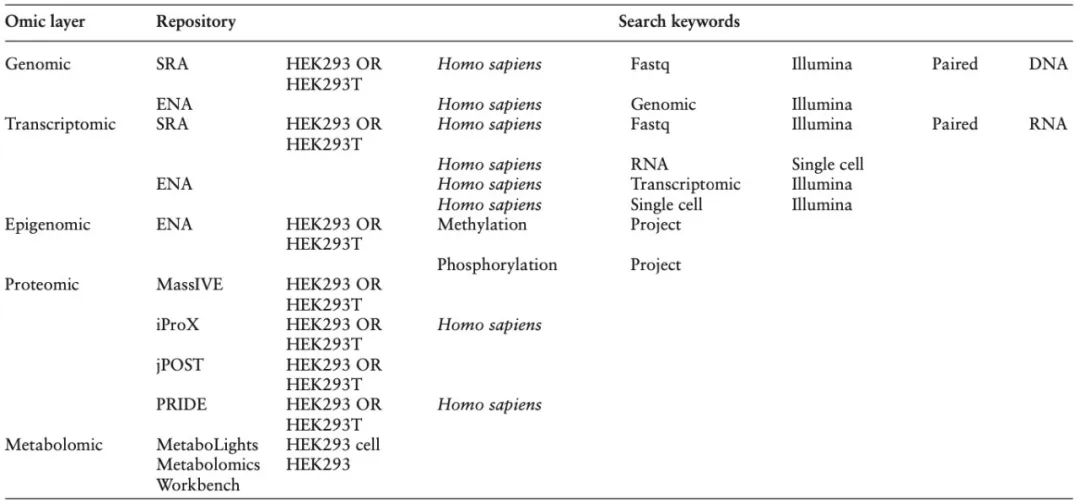
表1 按组学层次在每个数据库中的搜索关键词细分
结果和讨论
表2 数据库概览
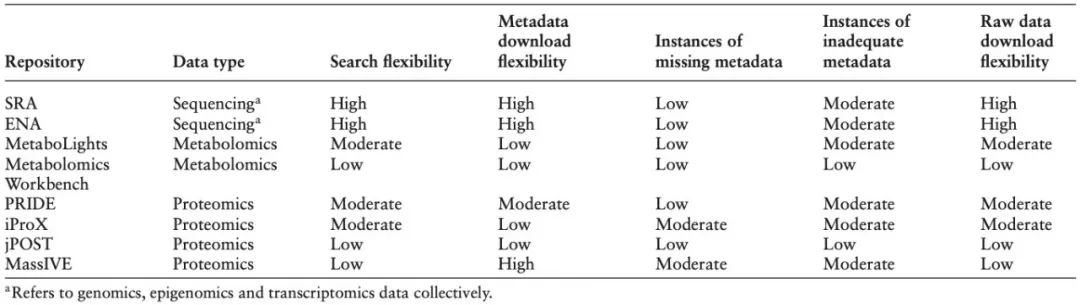
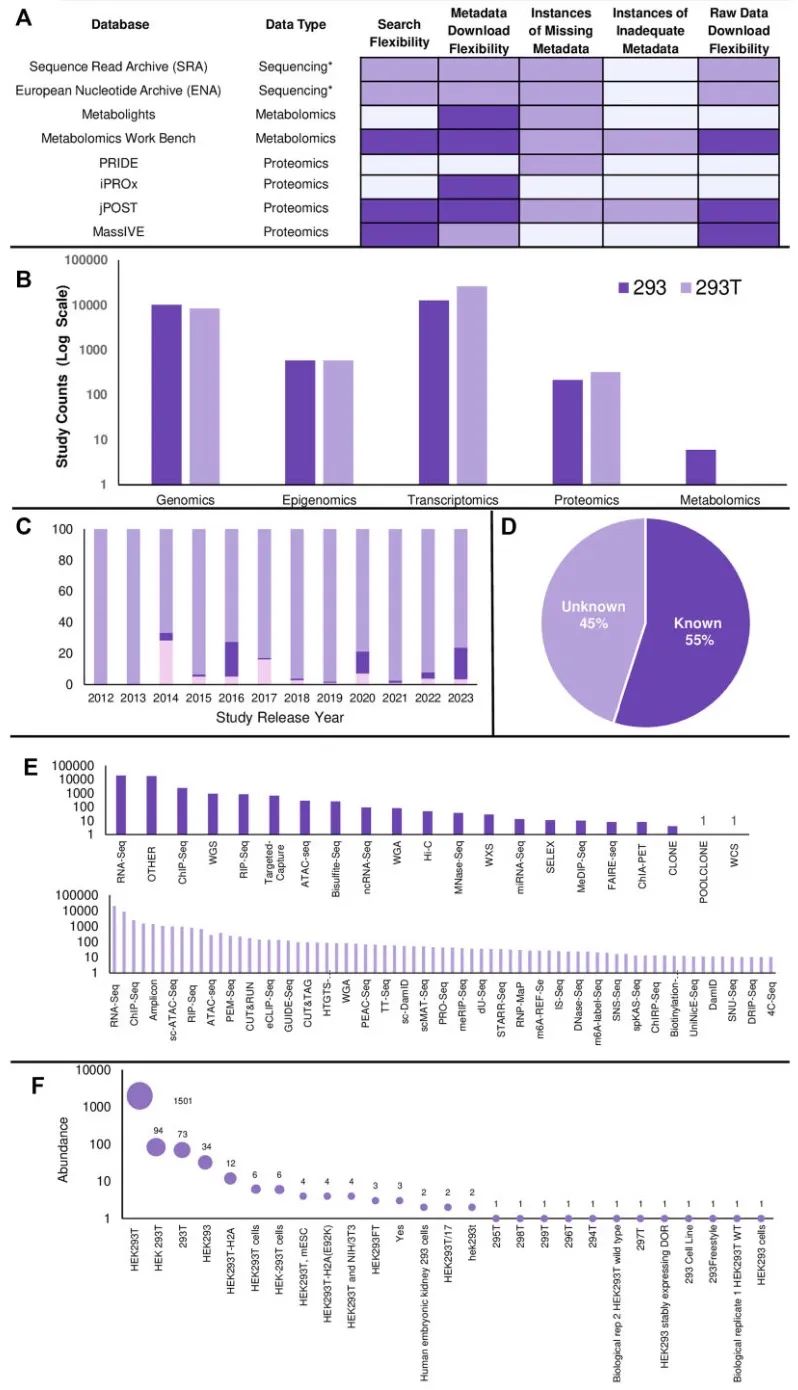
图1 数据管理过程的评估。(A) 所有数据库中观察到的问题概览。浅紫表示高需求领域,淡紫表示中等需求领域,突出需要改进的方面,深紫表示低需求领域。上标*指的是基因组学、表观基因组学和转录组学的总称。(B) 每种细胞类型可获得的不同数据类型的相对丰度。y轴是以log10尺度表示的。(C) 在SRA上随时间推移的样本性别报告情况。浅紫表示缺失数据。从2014年左右开始,样本性别的选项开始出现,平均而言,识别HEK293细胞系为女性的准确性最高,用粉红表示。深紫表示被标记为“不适用”或“未知”的实例,提供了有价值但不完美的见解。深紫表示研究人员在向SRA提交数据时经常遗漏指定样本性别的反复模式。自2022年以来,研究人员在数据描述方面有所改进,提高准确性仍然至关重要。(D) 从PRIDE数据库获得的蛋白质组学研究量化方法的见解。从PRIDE获取的所有研究中,100%的研究在其研究页面的属性表中包含量化方法的值。几乎一半的研究缺乏信息量丰富的量化方法;45%被标记为未知,突显了蛋白质组学元数据中的重要缺口。大多数数据集在出版物级别上可找到缺失的信息。(E) 测序数据的文库策略选择。深紫条形图根据存储库中报告的元数据显示文库策略类型的分布,其中模糊报告为“其他”促使需要手动整理。手动整理后可描绘出更全面的文库策略阵列,如浅紫条形图所示;为清晰起见,发生次数<10次的文库策略已被移除。(F) ENA上HEK293T细胞系元数据描述的频率和差异。每个气泡代表特定的细胞系描述,其大小表示在描述中出现的频率。
参考
Nucleic Acids Res. 2024 Oct 17:gkae901. doi: 10.1093/nar/gkae901
注:AI辅助创作,如有错误欢迎指出。内容仅供参考,不构成任何建议。
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容








所有评论(0)