
简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
到这里还没有结束,我非常看好iTALK种的LRPlot函数,不仅可视化了细胞之间的关系,还展示了具体的受配体,这样的图形一目了然。它有两个优点,第一是可以比较差异受配体,第二是可视化比较好,我们可以借用借用它的函数,让自己的结果更加完美!效果还是很好的,用在论文中也是挺吸引人的,对于结果的解读也是更加清晰。当然了,我们也提到,iTALK可以做组间差异比较,可能是由于我的数据随意构建吧,没有差异,这
今天接着单细胞文章的内容:从Cell学单细胞转录组分析(一):开端!!!跟着Cell学单细胞转录组分析(二):单细胞转录组测序文件的读入及Seurat对象构建跟着Cell学单细胞转录组分析(三):单细胞转录组数据质控(QC)及合并去除批次效应跟着Cell学单细胞转录组分析(四):单细胞转录组测序UMAP降维聚类跟着Cell学单细胞转录组分析(五):单细胞转录组marker基因鉴定及细胞群注释前面几
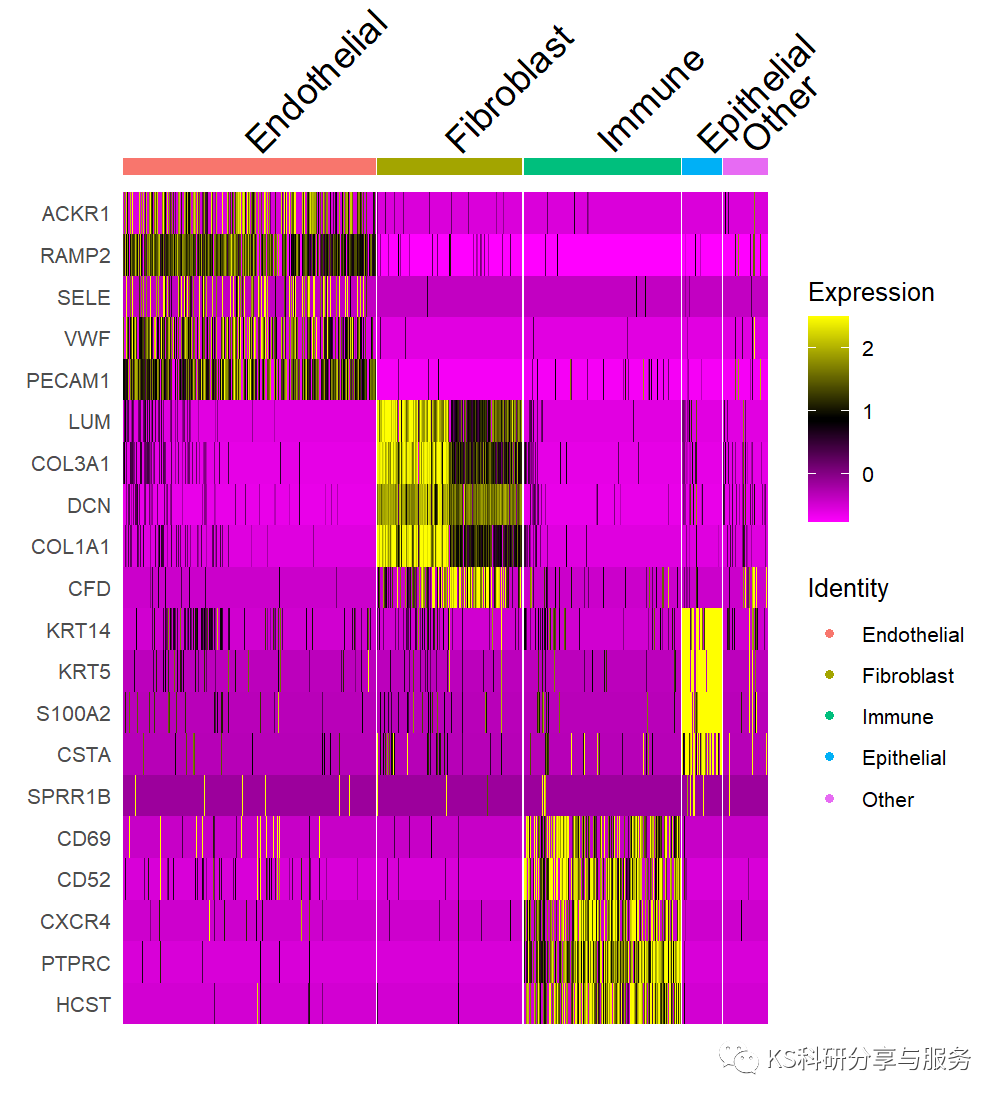
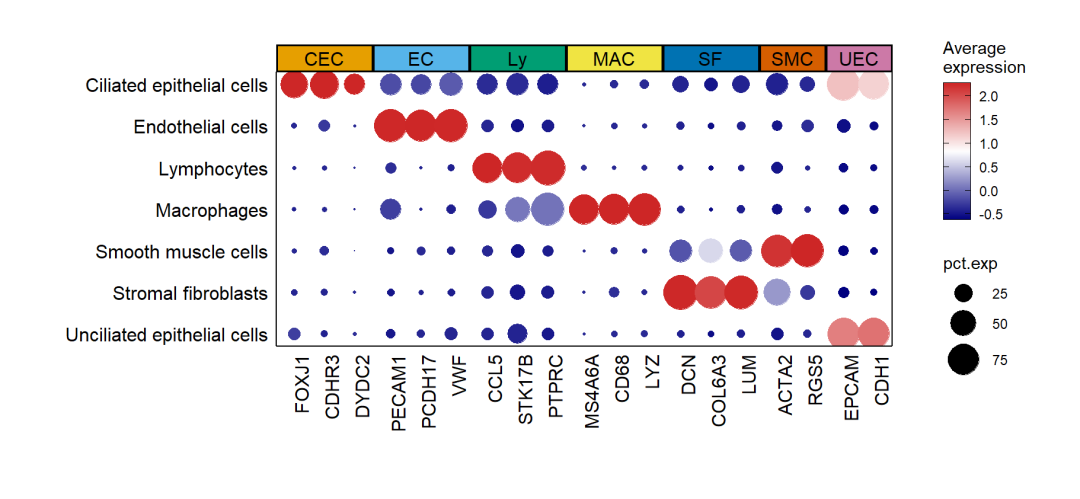
热图不再过多介绍了,参考之前的内容(热图系列大全)。单细胞基因可视化中热图也是比较受欢迎的,在分析完每群的marker基因之后,可以挑选显著的gene用seurat自带函数DoHeatmap可视化。当然也可以选任意自己想展示的基因进行可视化。首选选择基因,将其转化为列表,然后比对到原数据。markers <- c("ACKR1","RAMP2","SELE","VWF","PECAM1","
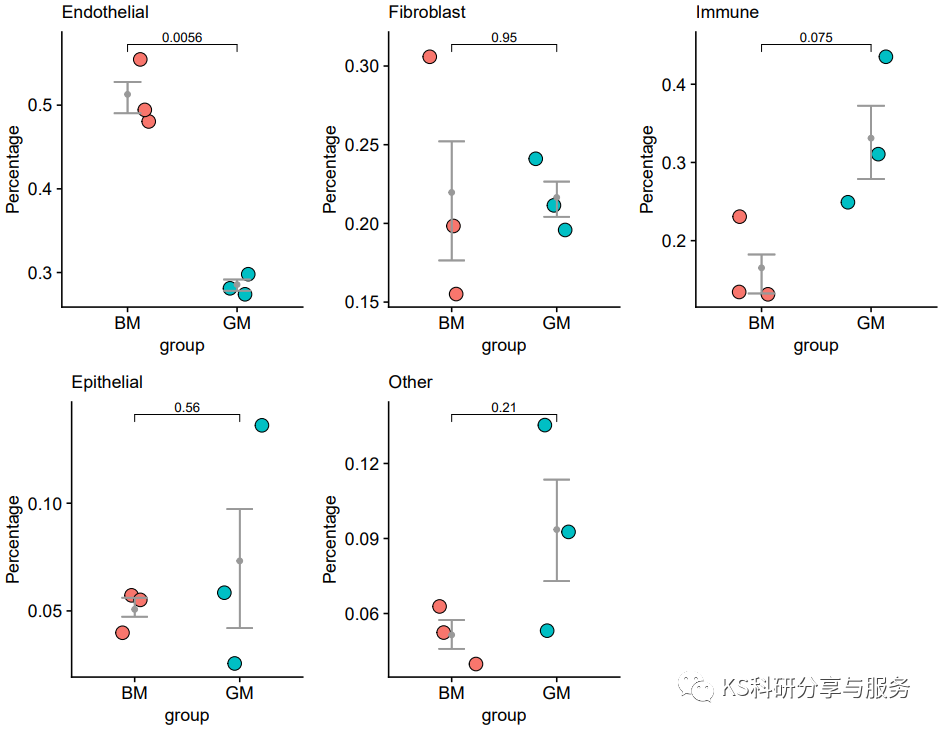
今天我们来复现一下一篇《cell》文章中的图标,图如下:(Liver Immune Profiling Reveals Pathogenesis and Therapeutics for Biliary Atresia)我们重点复现左边部分,右侧的作图在单细胞系列提到过:跟着Cell学单细胞转录组分析(十一):单细胞基因评分|AUCell评分,这里的复现不仅适用于单细胞,其他的数据也是可以做这样的

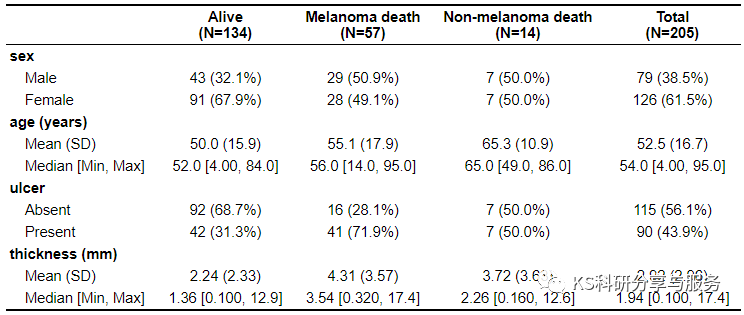
描述统计是统计学重要的一部分内容,尤其是在医学中的应用更广。很多医学、或者做统计的SCI文章,开头就是统计的内容,一般是由三线表的方式呈现的。之前小编也只会用excel统计然后自己制作表格,但这样效率很慢。这里我们介绍一个R包---table1,可以非常简单快速的完成统计工作,并制出三线表!安装包和示例数据install.packages("table1")install.packages("bo
,很多小伙伴购买函数,在使用过程却出现问题,主要的原因是我们在帖子里写的不够清楚,小伙伴也没有理解代码的意思,鉴于一个一个解释太费时间,我们决定写函数的帖子一律录制视频解说(视频在B站,搜索:KS科研分享与服务),方便大家使用。接下来看看bulk的效果,对于bulk表达数据,bulk基因表达量那一列列名要修改命名成exp,对于非单细胞的数据, 首先要进行设置的一个参数就是single_type=F
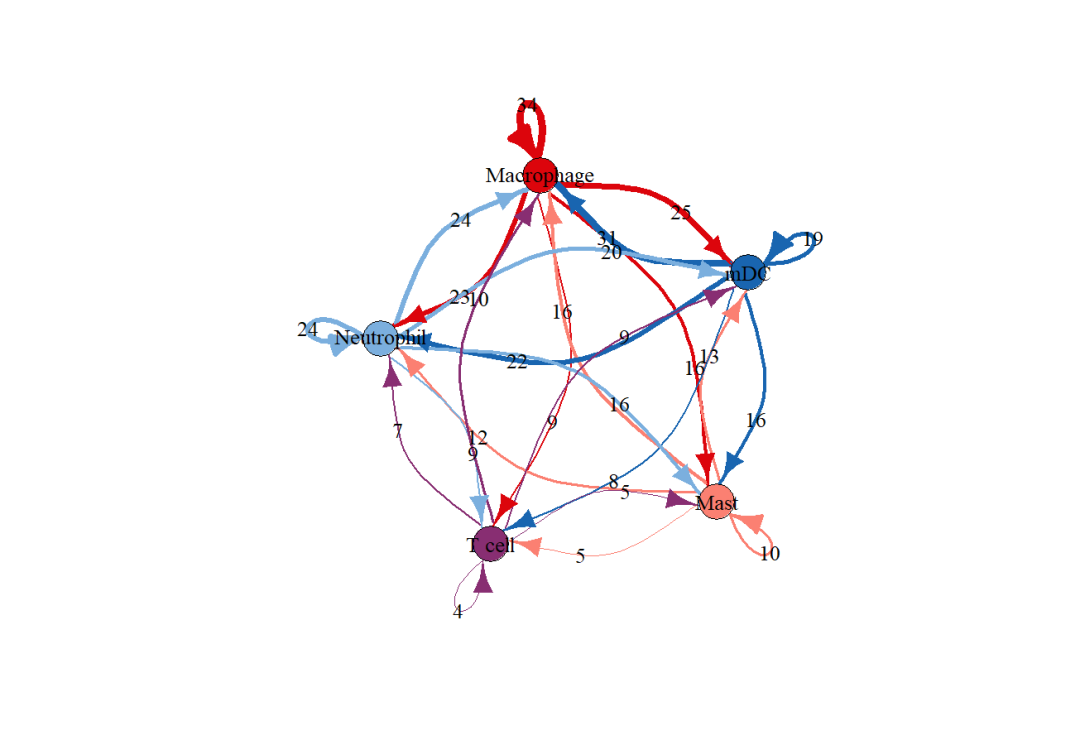
总之,这个图还是很有用的,一个图展示了多个信息,但是凑图这个路被堵死了[图片上传中...(image-152128-1687142947490-4)]然后计算细胞比例,添加上每个细胞群中心位置,用于添加饼图。也可以添加上细胞群的数量,后面做一个相对化处理,用来表示饼图大小,这个图就会更加生动。在这样就完成了,感兴趣的小伙伴可以在自己文章里面展示起来了。本来是一个简简单单的小破图, 可是需求这个东西

更多内容请关注个人公众号---KS科研分享与服务---接上节(跟着Cell学单细胞转录组分析(三):单细胞转录组数据质控(QC)及合并去除批次效应)。数据合并之后,就需要跑标准的Seurat分析流程了。在《cell》文章中,作者还计算了细胞周期评分,因为我们收集到的细胞可能处于不同的分裂时期,所以看周期是很有必要的,尤其是针对具体的研究目的。在示例数据中,可以看到,各个样品细胞周期基本一致。s.g

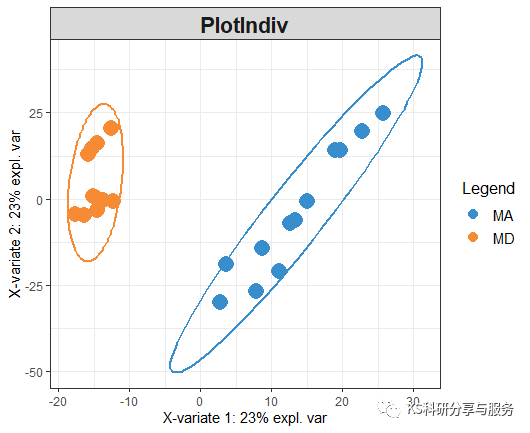
最近有小伙伴问道PLS-DA(Partial least squares Discriminant Analysis,偏最小二乘判别分析),一种组学分析中常用的多变量分析方法,具体的原理就不多说了,主要也怕数学的内容说错了误导人,我们就看看R怎么分析和可视化即可,详细的原理可翻阅《多元统计学》偏最小二乘判别分析部分学习。其实,如果不做代谢组学或者看这方面的文章,一般学点组学分析的人还真不容易看到P
关于单细胞转录组转录因子的分析我们之前在单细胞系列讲过R语言版本的,参考:跟着Cell学单细胞转录组分析(十二):转录组因子分析,但是R语言分析起来速度非常慢,如果你动辄上万的单细胞可能要运行好几周,这显然不现实。pySCENIC则很好的解决了这个问题,分析速度很快。











