[TBME 2023]Patient Clustering for Vital Organ Failure Using ICD Code With Graph Attention
计算机-人工智能-预训练ICD聚类

目录
2.5.2. Model Training and Assessment
2.5.3. Validation of the Deep Clustering Pipeline on MNIST
2.6.1. Testing Deep Clustering Model on MNIST
2.6.2. OF Patients Deep Clustering on MIMIC-III
2.6.3. Comparison with DCN and Med-BERT
1. 心得
(1)failure?me!
(2)怪费解的
(3)聚类类别比较少,聚的大类
2. 论文逐段精读
2.1. Abstract
①作者是无监督的。咦
respiratory adj.呼吸的 comorbidity n.共病;并存疾病;共病性;共病现象;共病症
2.2. Introduction
①作者还是从现存医学问题写到了他们用无监督。但类别很多的情况下无监督真的好吗?有罕见类的时候又能真的分出来吗?
2.3. Related Work
①学习嵌入,匹配患者记录矩阵和ICD嵌入矩阵的概率
②ICD ontology?对ICD树使用图神经网络
③纯嵌入聚类
2.4. Data
①数据集:MIMIC-III
②编码:ICD-9
③作者挑的和衰竭有关的24个,包含:428 (HF)、518.81(急性 RF)、518.83(慢性 RF)、518.84(急性和慢性 RF)、518.51(创伤和手术后的急性 RF)、518.53(创伤和手术后的急性和慢性 RF)、770.84(新生儿 RF)、584(急性 KF)、 669.3(分娩后急性 KF)和 586(肾功能衰竭)等
④统计数据:

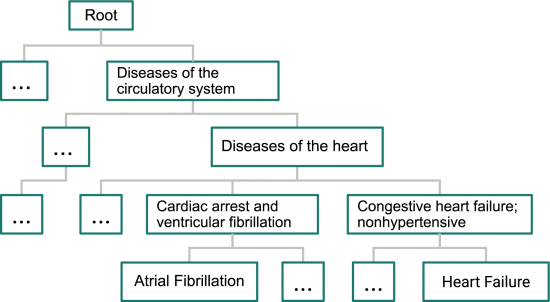
⑤在临床分类软件(CCS)中,使用多级 CCS 来构建本体树:
2.5. Methods
2.5.1. Analysis Pipeline
①框架示意图:
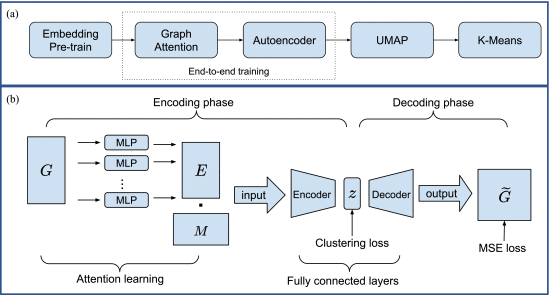
②为每个ICD叶节点建立嵌入矩阵。
都是由祖先和自身的线性加权构成:
其中是一个两层的MLP

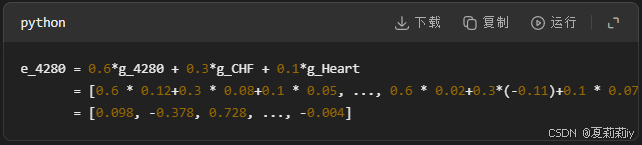
③使用,其中
是患者数量,
是ICD代码总数:
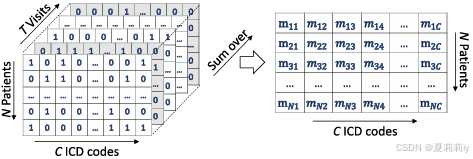
是右边压缩的那一个。作者一开始的第三维
是说复诊次数,每次可能疾病代码不一样了。空的没来复诊的用0填充
④使用GloVe来初始化,
是目标ICD编码的祖先
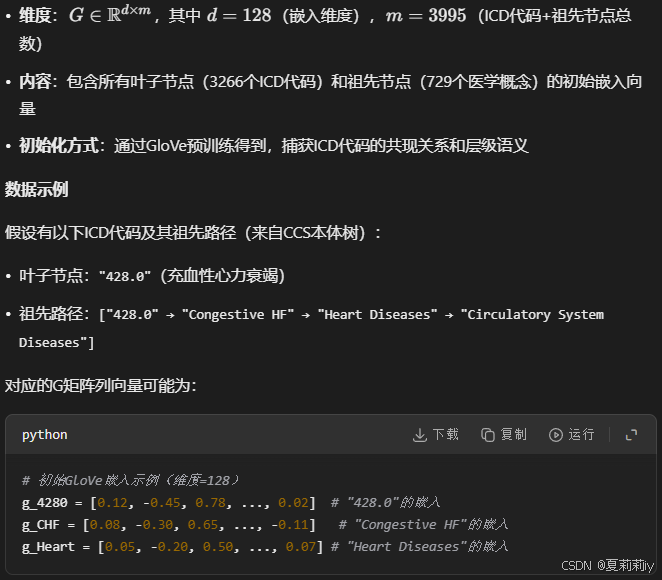
⑤重建损失:
其中是聚类数
2.5.2. Model Training and Assessment
①预训练:使用GloVe单独预训练和AE预训练;联合训练
2.5.3. Validation of the Deep Clustering Pipeline on MNIST
①在MNIST数据集上测试深度聚类模型?这好吗?感觉是完全不一样得分东西诶
②评估聚类效果:NMI互信息
2.6. Results
2.6.1. Testing Deep Clustering Model on MNIST
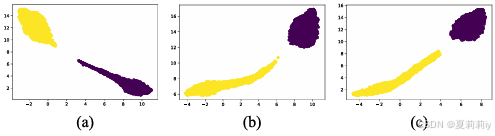
①在MNIST数据集上聚类的混淆矩阵:
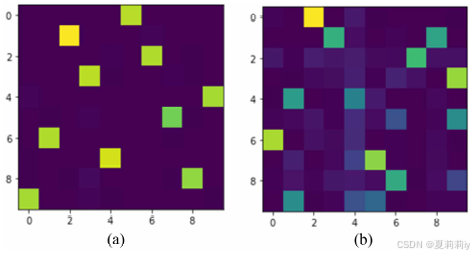
其中(a)由UMAP但是(b)没有
2.6.2. OF Patients Deep Clustering on MIMIC-III
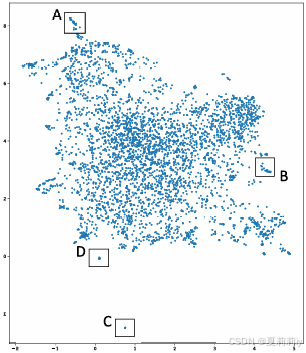
(1)Interpretation of ICD Pre-Train
①使用UMAP和GloVe的聚类分布:
(2)AE Pre-Train
①加上AE的聚类:
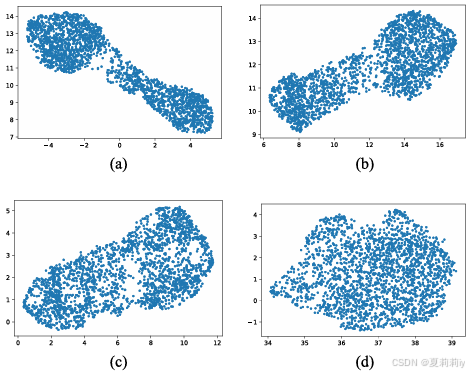
四个图具有不同的隐藏层
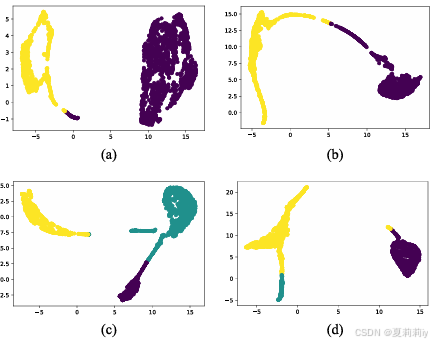
(3)Clustering Results From the Join-Train Stage
①联合训练阶段的聚类:
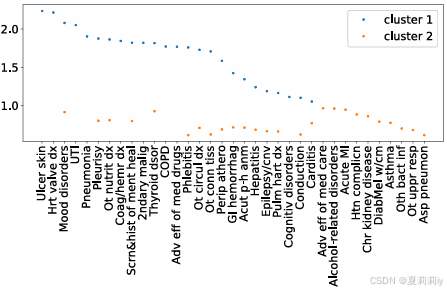
②疾病出现频率:
2.6.3. Comparison with DCN and Med-BERT
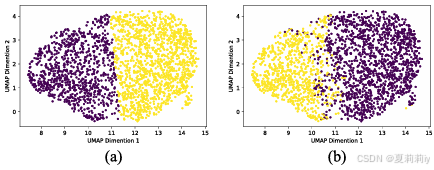
①对于不同聚类数下的聚类分布:
②Med-BERT下的聚类:
2.7. Discussions
①emm
2.8. Conclusion
~
更多推荐
 已为社区贡献11条内容
已为社区贡献11条内容






所有评论(0)