简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
基于Shiny开发的微生物组数据分析工具,旨在简化OTU/ASV_taxa表格的处理流程。该工具支持多种文件格式,通过调用QIIME1自动生成微生物分类学汇总结果,并优化了错误处理机制,提供实时日志反馈。用户无需命令行操作即可完成从原始数据到出版级统计表的全流程分析,显著提升效率。文章还对比了不同AI工具在Shiny应用开发中的表现,特别指出国产deepseek在功能实现和界面设计上的优势。工具已

美国退伍军人事务部的百万退伍军人计划(MVP)建立了全球最大的退伍军人基因组数据库,与能源部合作开发了CIPHER知识共享平台。该研究对63万退伍军人进行全基因组关联分析,发现3.8万个独立变异位点,其中2069个在非欧裔群体中发现,凸显遗传研究多样性的重要性。平台提供了PheWeb、gwPheWAS、GeoPheno等多个在线工具,支持表型基因组数据的可视化分析,包括疾病流行地理分布、药物靶点发
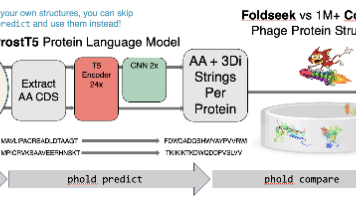
pharokka是一款专为噬菌体基因组设计的快速注释工具,最新版本为1.7.0(2024年6月更新)。它使用PHANOTATE进行基因预测,通过MMseqs2比对PHROGs等数据库进行功能注释,并支持tRNA、CRISPR检测等功能。phold是其补充工具,采用深度学习模型ProstT5进行结构注释,适合特征较少的噬菌体。两者可联合使用:先用pharokka完成基础注释,再用phold进行结构增
最近deepseek的大火,让大家掀起新一波的本地部署运行大模型的热潮,特别是deepseek有蒸馏的小参数量版本,电脑上就相当方便了,直接ollama+open-webui这种类似的组合就可以轻松地实现,只要硬件,如显存,RAM足够,参数量合适,速度还可以接受。本地部署的意义在于,一是可以数据不上网,让一些私密的数据有所保障,二是可以实现一些在线限制的功能。在手机上运行的意义,其实更多可能是玩玩

最近尝试入门点深度学习的内容,首先来个框架试试嘛,tensorflow2.0的GPU要求已经是nvida算力3.5以上的设备,当然,如果你能过G F W,tesla据说可以白嫖。想用自己的设备咋办呢,只有单纯CPU上了,那这速度就无语了,慢上几十倍,毕竟再差的GPU的核心数,也是cpu的几十倍,所以使用opencl,苹果metal等的框架是个不错的选择,在知乎上发现了一个答主推荐plaidml这个
STREAMS指南为环境和非人类宿主微生物组研究提供标准化报告框架,包含67项核心条目。该指南在STORMS基础上扩展,涵盖从研究设计到数据分析的全流程,特别针对环境研究的特殊性提供操作建议。通过248名研究人员的共识,STREAMS制定了机器可读的DMP工具模板和8个应用案例,推动微生物组数据的FAIR化。作为持续更新的"活文档",该指南为研究者提供规范化投稿准备,同时为审稿
发现手上的GT-740M,已经太老,算力3.0,早被pytorch抛弃,想要学习下深度学习,基于没好卡,刚好时间还有点,花了一天的时间编译了一下,发现显存是个大难题,可以把数据或模型搞小点吧!主要参考了这两个,基本上是个体力活,只按按照版本来的话是不会报错的,当然,也是挺花时间的,需要6个小时以上,i3-3120M,双核2.5G Hz的CPU。
最近尝试入门点深度学习的内容,首先来个框架试试嘛,tensorflow2.0的GPU要求已经是nvida算力3.5以上的设备,当然,如果你能过G F W,tesla据说可以白嫖。想用自己的设备咋办呢,只有单纯CPU上了,那这速度就无语了,慢上几十倍,毕竟再差的GPU的核心数,也是cpu的几十倍,所以使用opencl,苹果metal等的框架是个不错的选择,在知乎上发现了一个答主推荐plaidml这个
平台集成了数十个插件和数百种方法,支持从扩增子到宏基因组的多种数据类型处理,并且可视化功能强大,交互性良好,适合科研展示与结果解读。目前平台已推出多个“分布版”,例如扩增子分析套件、MOSHPIT 宏基因组工具包、致病基因组检测模块,以及面向开发者的最小框架版,覆盖了从基础研究到临床应用的广泛场景。对于希望深入参与的研究者,无论是否具备编程背景,都有明确的参与路径和贡献指南。QIIME 2 202
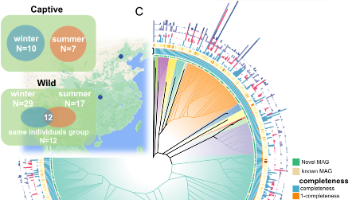
摘要(150字) 本研究通过宏基因组与宏转录组分析,揭示了金丝猴肠道微生物组响应季节性饮食变化的适应机制。研究发现野生金丝猴肠道微生物在夏季富集植物次生代谢功能,冬季则转向地衣多糖降解和能量平衡相关菌群(如毛螺菌科),而圈养个体微生物组无显著季节性波动。研究重建了578个宏基因组,其中76.5%为新菌种,凸显了金丝猴肠道微生物的高度新颖性。结果表明肠道微生物通过功能可塑性帮助宿主适应环境变化,为濒