[MIA 2021]Segmentation of cellular patterns in confocal images of melanocytic lesions in vivo via a
计算机-人工智能-多尺度软硬损失RCM皮肤组织分割

论文代码: https://github.com/kkose/MED-Net
目录
2.3.1. Semantic segmentation network architecture
2.4.1. Scenario-1: patient-wise cross-validation experiment
2.4.2. Scenario-2: clinic-wise cross-validation
2.5. Discussion and conclusions
1. 心得
(1)考古ing
(2)牛哇牛哇,虽然很早期了但也很值得看,适合新手入门
2. 论文逐段精读
2.1. Abstract
①作者旨在模拟人类的从粗到细的探查方式,设计了Multiscale Encoder-Decoder Network (MED-Net)
2.2. Introduction
①RCM很难人眼识别,因此开发深度学习是更高效的
②分割类别:四种细胞形态模式(纹理结构)以及伪影和非病变背景的两个“额外”类别
③挑战:数据集中只有 58% 的像素被我们的专家标记,但MED-Net能够对“部分标记”数据进行训练
2.3. Materials and methods
①马赛克数量:117 个有关黑色素细胞皮肤病变
②马赛克尺寸:从 7000 × 8000 像素到 12000 × 12000 像素不等,对应于 14 到 36 平方毫米之间的区域
③具体类别:环形、网状、嵌套型和非特异性(细胞形态),伪影和非病变背景:
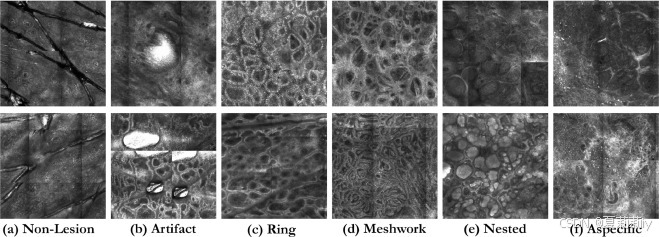
④标注者:两位专家使用开源软件包 Seg3D进行标记
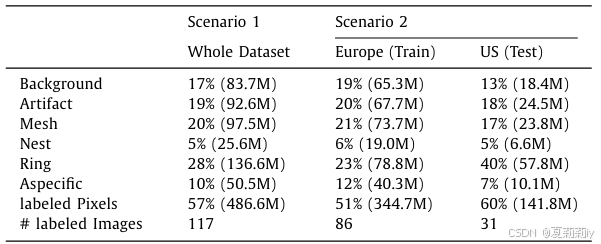
⑤标注量:
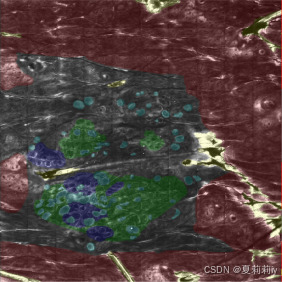
⑥标注样本:
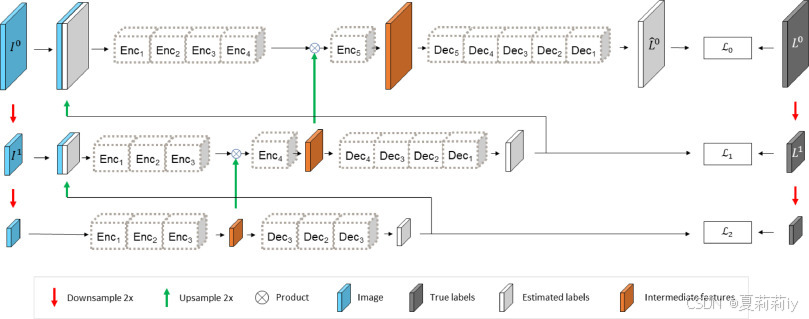
2.3.1. Semantic segmentation network architecture
①网络架构:
2.3.2. Loss function
①标准骰子相似系数损失:
这样的损失忽略了真阴性的检测而只在意真阳性。作者设计了考虑真阴性的软骰子损失:
其中GT 和预测
都是
,前两个维度是图片,最后一个维度是类别独热编码。对于属于
类的某个像素点,
。其中第一项是希望两个像素点在同个独热编码上都为1,第二项鼓励两个像素点在同个独热编码上都为0
②正则化损失函数:
这个损失是确保像素类别变化平缓
③总损失:
其中超参数
2.3.3. Implementation details
①在将马赛克输入 MED-Net 之前,将它们下采样了 4(根据专家说,2 μm/px 分辨率下也可以识别),以256 × 256 像素(0.5 毫米× 0.5 毫米)的块处理马赛克。虽然补丁是256 × 256 像素,但步幅只有32,这样一个像素最多会被决策八次。作者没有将这八次的预测直接取平均,而是为每个Patch分配一个二维高斯权重掩模。作者认为,一个Patch越靠近中心的位置,其预测结果越可靠。越靠近边缘,可靠性越差。
②Epoch: 200
③学习率:0.01,权重衰减为1e-8
④批量大小:48
⑤所有卷积层都使用 He Normal 初始化进行初始化
⑥通过空间采样实现了数据增强。为了涵盖可以从马赛克中提取的所有可能的补丁,在每个纪元之前,以滑动窗口方式提取 512 × 512 像素的补丁,重叠率为 50%。然后,在训练的每个时期,在较大斑块内的随机位置提取了 256 × 256 像素斑块。其他的数据增强有:

⑦推理输出形状:256 × 256 × 6
2.4. Results
①场景1中使用五折交叉验证,场景2按诊所分,欧洲诊所的用于培训和验证,美国的用于测试。
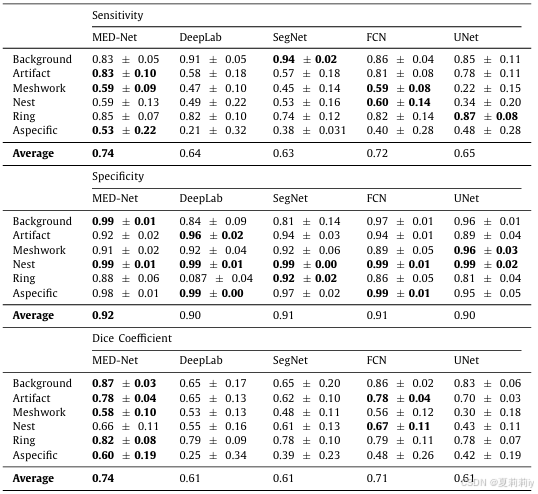
2.4.1. Scenario-1: patient-wise cross-validation experiment
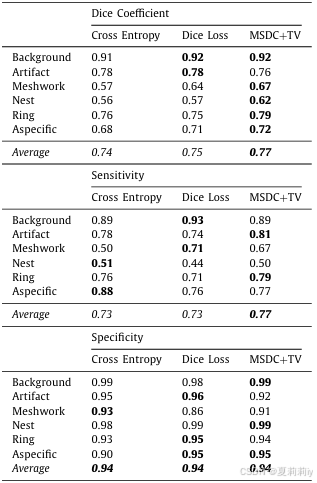
①对比表:
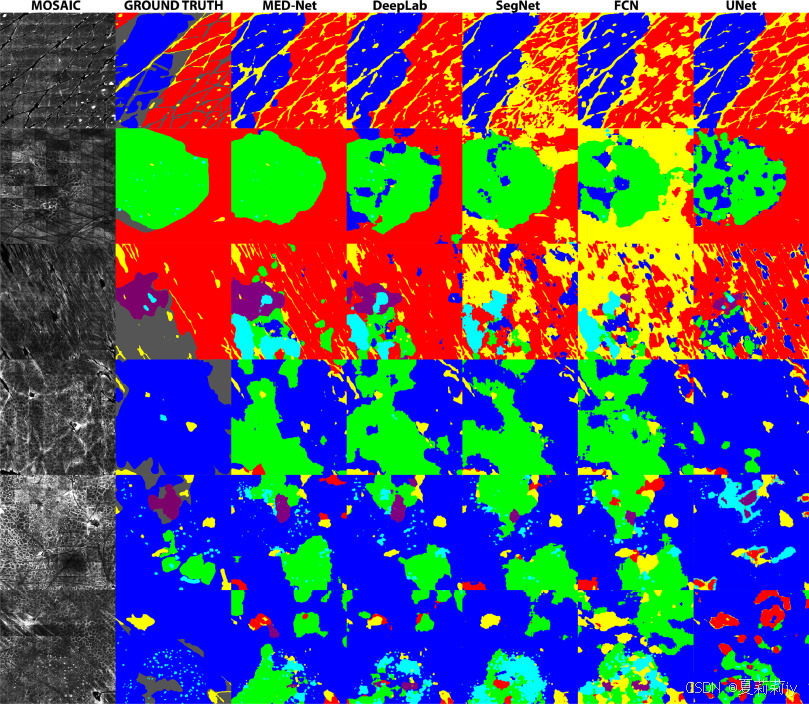
②分割示例:
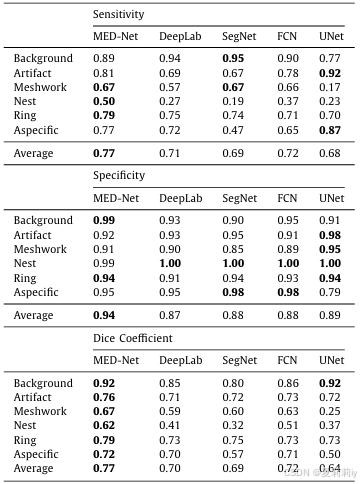
2.4.2. Scenario-2: clinic-wise cross-validation
①结果:
2.4.3. Ablation studies
①层数消融:
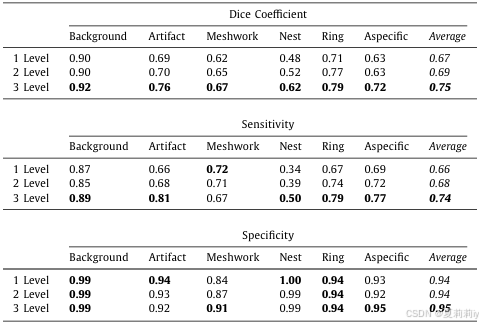
②损失消融:
2.5. Discussion and conclusions
①标注困难
②当前版本的 MED-Net 已经集成到我们研究所的两台 RCM 设备中,与制造商密切合作,并很快在诊所进行实时测试以进行性能分析(怎么集成进去的!!)
更多推荐
 已为社区贡献11条内容
已为社区贡献11条内容






所有评论(0)