医学图像处理——读取和解读NII文件
NII文件的存储格式网上有很多资料,在此只做一点简单的描述。nii是一种文件格式,它存储的是在空间中占有一定体积的小方块的物理位置和该位置对应的像素值。这个小方块我们也称之为体素(voxel)。存储的形式是一个三维数组(3D array),具有三个索引ijk。它存储的物理空间(3D volume)解析出来是整整齐齐的长方体。但是表示一个体积仅仅靠数组是不够的,需要打一些补丁。我们通过苏格拉底式地提
一 预备知识
NII文件的存储格式网上有很多资料,在此只做一点简单的描述。nii是一种文件格式,它存储的是在空间中占有一定体积的小方块的物理位置和该位置对应的像素值。这个小方块我们也称之为体素(voxel)。存储的形式是一个三维数组(3D array),具有三个索引 i , j , k i, j, k i,j,k。它存储的物理空间(3D volume)解析出来是整整齐齐的长方体。但是表示一个体积仅仅靠数组是不够的,需要打一些补丁。我们通过苏格拉底式地提问慢慢还原出nii文件的一些重要参数设置。
1. 数组只有索引,怎么表征每个体素具体的物理位置信息?
但是,仅仅有三维数组是无法表征物理位置的,因此还需要关注另外一个参数,zooms,也就是这个小方块的长宽高对应的单位长度,他们具有一定的比例关系,它所代表的具体物理单位是可以人为赋予的,只需保证人为定义之后能够保持后续图像处理程序所使用的单位一致即可,比如可以是毫米mm,也可以是厘米cm,甚至是米m。可以理解为做三维重建时的spacing参数。有了zooms,我们可以结合数组的shape计算出整个nii文件存储的3D volume在空间中的大小。例如对应一个索引为 ( i , j , k ) (i, j, k) (i,j,k)的voxel,它相对于这个数组坐标系原点的物理位置为 ( i × z o o m s [ 0 ] , j × z o o m s [ 1 ] , k × z o o m s [ 2 ] i\times zooms[0], j\times zooms[1], k\times zooms[2] i×zooms[0],j×zooms[1],k×zooms[2])。
2. 怎么知道一个物理空间中的三维体积是按照什么方向存储到数组里的?
这个问题再啰嗦一点表达就是,如果我对一个长方体用索引数据的方法(如
i
m
g
[
0
,
:
,
:
]
img[0,:,:]
img[0,:,:])进行切片处理,要怎么知道得到的图片是按照哪个方向且出来的。 索引的坐标原点在这个长方体的那个角落?这在医学上是一个比较重要的问题,因为当我们说左右的时候,从病人的背部表示的左右和从病人的正面表示的左右是不同的。在医学的早起,甚至有出现过做手术本来要切除身体左侧病变的部分脏器,因为左右没有表达好切成了右边的脏器的悲剧,所以后来才会先在病理部位用笔标识好后才做手术的惯例。扯回原话题,在医学上有把人体看做是一个长方体,定义了冠状面,矢状面等标准面,把人体的坐标系方向表达为前后(胸部Anterior,背部Posterior),左右(左手Left,右手Right),上下(头部或颅部Superior / Cranial,脚部或尾部Inferior / Caudal)。
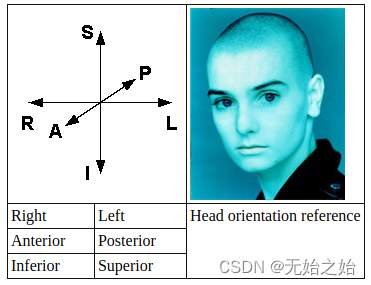
这也是nii的定义,定义了所存储的数组对应的方向是指向那个部位的,如 P, I, R 表示的是数组中第一个索引
i
i
i 对应的
x
x
x 轴正方向是从人体的前胸部指向人体后背部的,
j
j
j 对应的
y
y
y 轴正方向从人体的头颅指向脚部,
k
k
k 对应的
z
z
z 轴正方向从人体的左手指向右手。有一点需要注意的是,这几个字母表达的是
T
o
To
To,即指向那个部位为正方向,而不是从哪个方向开始。详细的定义可以看这个链接 Orientation and Voxel-Order Terminology: RAS, LAS, LPI, RPI, XYZ and All That: http://www.grahamwideman.com/gw/brain/orientation/orientterms.htm. 或者这篇博客:https://blog.csdn.net/zhangjipinggom/article/details/118523633
3. 如何表示像素在物理空间中的坐标?
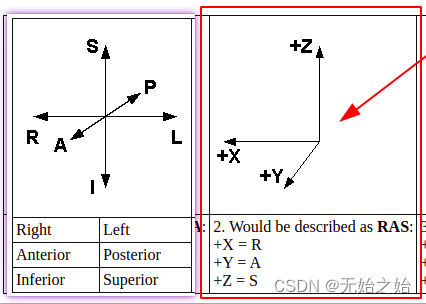
解决问题1之后,我们仅仅只是知道了体积之间,各个体素之间的物理位置关系。如果我们用同一台设备分多次扫描了用一个物体的不同部位然后再把他们整到一起,我们就需要知道每一次扫描得到的volume相对于该设备参考坐标系(reference frame)下的相对位姿。这时候nii就需要定义一个齐次变换矩阵(affine matrix)对体素进行坐标变换了。我们可以把问题1得到的各个体素的位置看作是解剖坐标系,这个affine matrix就是把解剖坐标系下的坐标变换到参考坐标系下的一个变换。其中,affine matrix的方向对应的是问题2中定义的方向,以
R
,
A
,
S
R, A, S
R,A,S 方向分别对应
x
,
y
,
z
x, y, z
x,y,z 轴的正方向(即右手系),注意上图中定义的正方向是以病人为中心定义的,所以我们从外部看的时候定义R在左边,L在右边。而affine matrix中定义的位置为相对于参考系定义的位置。
二 示例代码和解析
为了进一步理解nii文件存储的信息,我们提供了下面的示例代码。nii文件则是从医学影像顶会MICCAI的2020挑战赛中的Spine CT中获取,nii文件可以在这个github https://github.com/anjany/verse 的util/sample文件夹中。
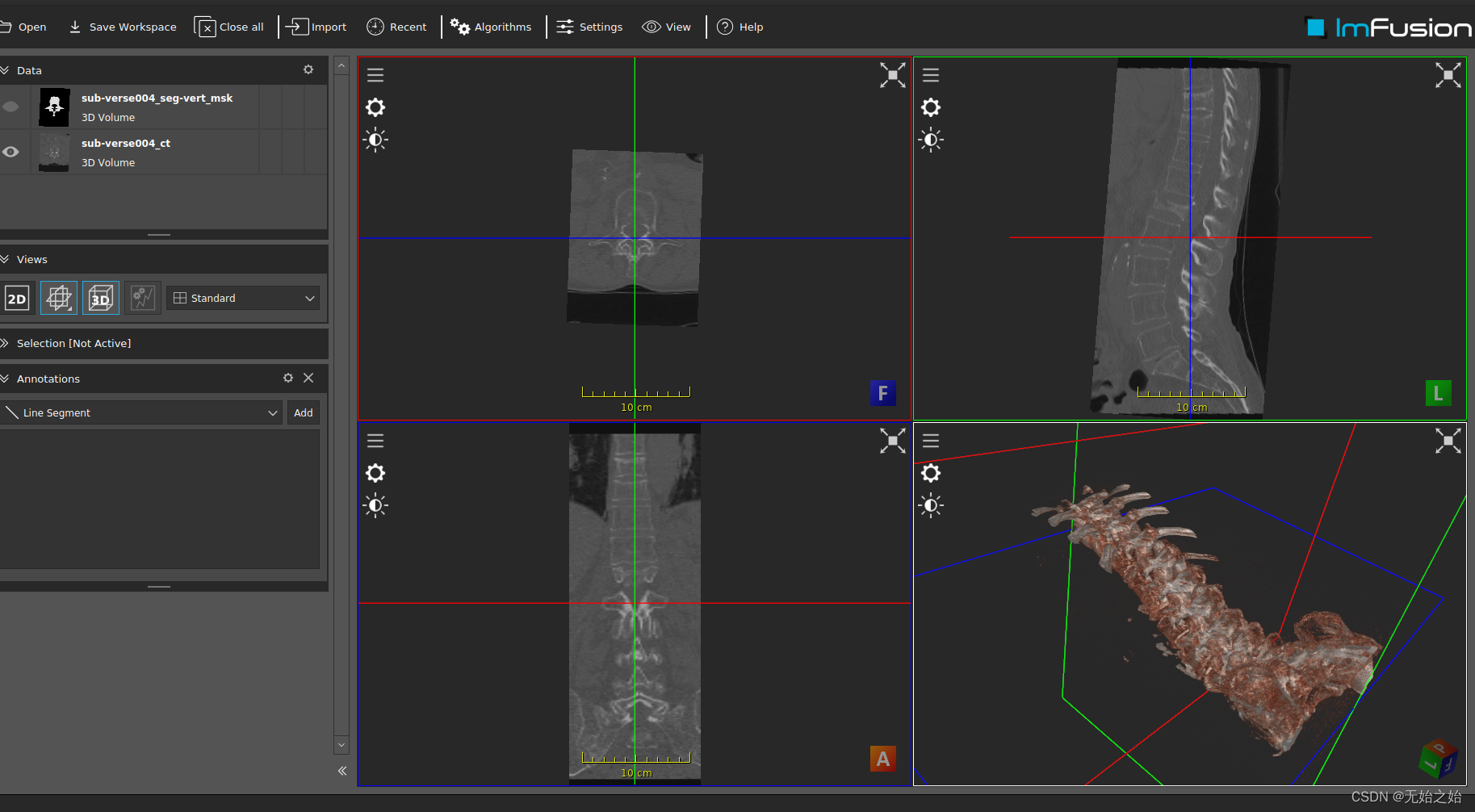
0. 看看nii文件长什么样子
将 sub-verse004_ct.nii.gz 文件拖拽到可视化软件中,如3D slicer (怎么搞自己网上搜),或者imfusion文件,可以看到如下图片:
1. 读取nii文件
import os
import numpy as np
import nibabel as nib
import nibabel.orientations as nio
import matplotlib.pyplot as plt
from data_utilities import *
directory = 'xxxxx'
img_nib = nib.load(os.path.join(directory,'sub-verse005_ct.nii.gz'))
2. 获取数组并查看nii的相关参数
# array info
img_data = img_nib.get_fdata()
print('img shape: ', img_nib.shape)
print('data shape: ', img_data.shape)
print('data type: ', type(img_data))
# volume info
zooms = img_nib.header.get_zooms() # similar to the spacing settings in the 3D compounding
print('zooms of the voxel: ', zooms)
axs_code = nio.ornt2axcodes(nio.io_orientation(img_nib.affine))
print('img orientation code: {}'.format(axs_code))
# global info
print('affine matrix: ', img_nib.affine)
3. 查看切片进一步理解方向表示
def show_slices(slices):
fig, axes = plt.subplots(1, len(slices))
for i, slice in enumerate(slices):
axes[i].imshow(slice, cmap='gray')
slice_0 = img_data[img_shape[0]//2, :, :]
slice_1 = img_data[:, img_shape[1]//2, :]
slice_2 = img_data[:, :, img_shape[2]//2]
show_slices([slice_0, slice_1, slice_2])
plt.suptitle("Center slices for body image")
plt.show()
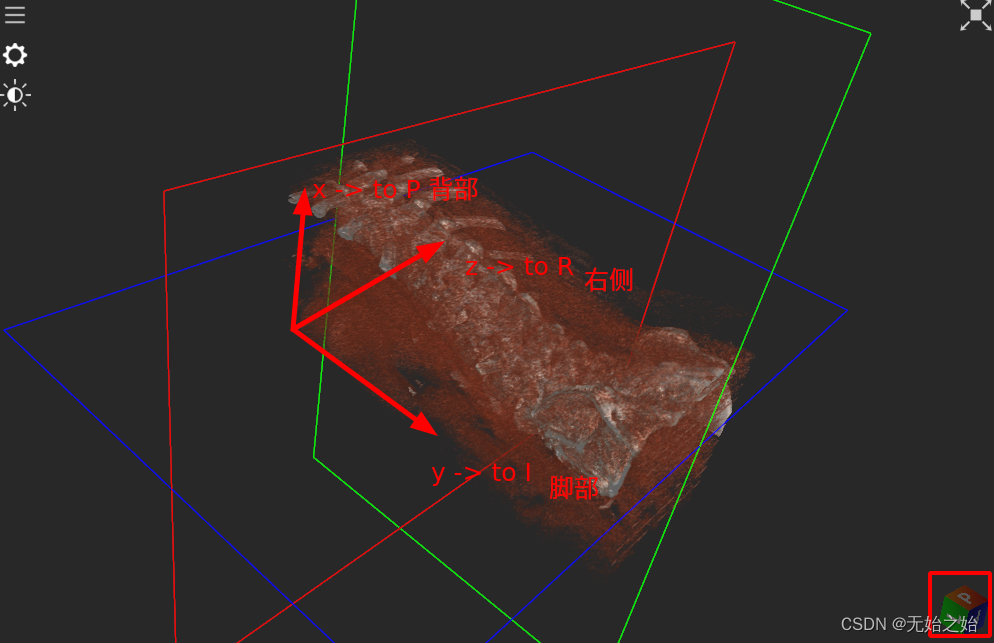
根据步骤2输出的结果:img orientation code: (‘P’, ‘I’, ‘R’)。我们可以判断出切片的
x
x
x轴(第一个索引, 图像的行)和
y
y
y(第二个索引, 图像的列)。坐标系的表达如下面几张图所示:

切片中的坐标系的定义如下:
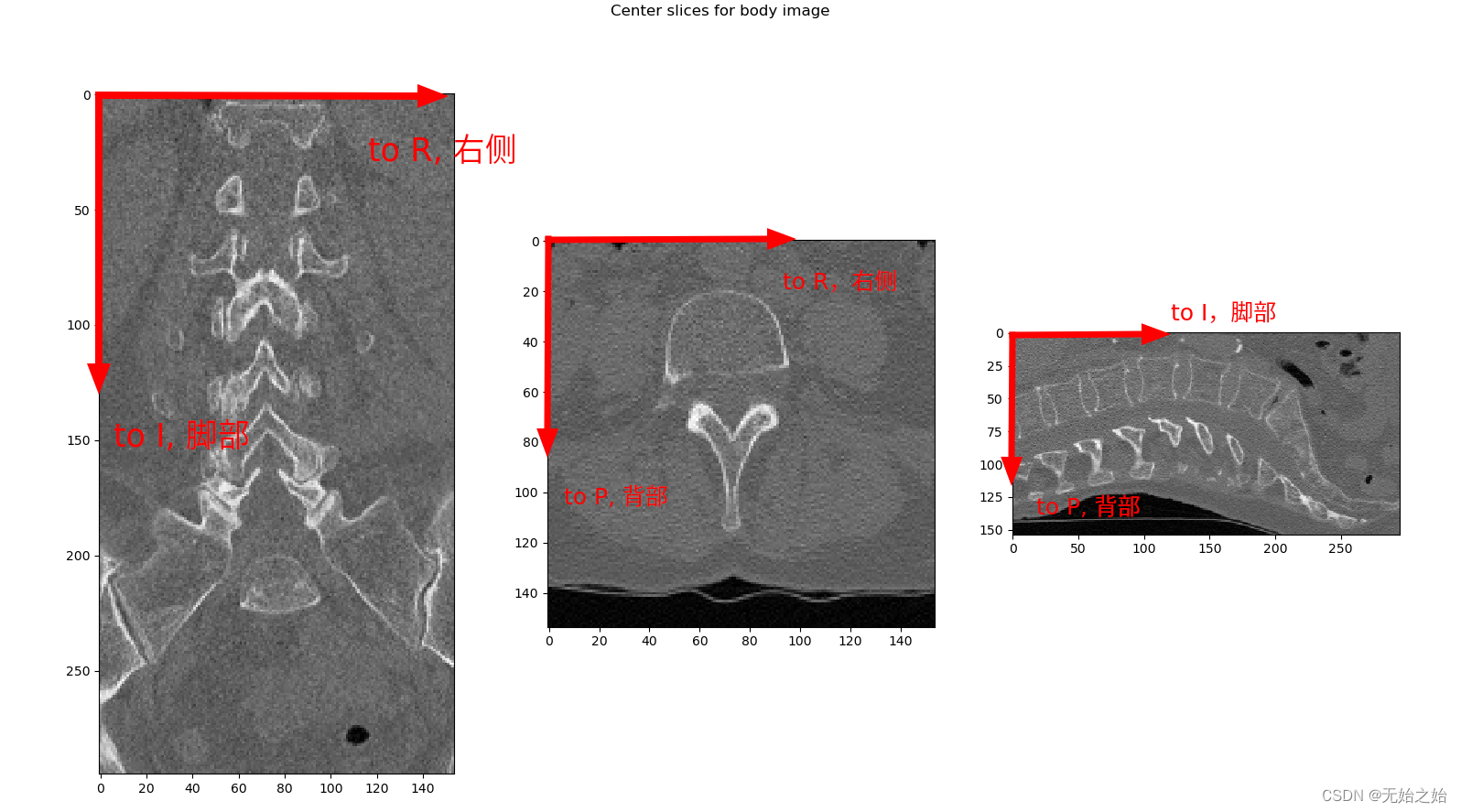
以上,
2023年7月12号
Dianye
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容










所有评论(0)