特征选择(模型输入参数的分析选择)方法汇总
特征选择是模式识别和机器学习领域的重要研究课题之一。为了提高准确率, 人们往往最大限度地提取特征信息。然而, 过大的特征向量维数不仅导致计算成本的增加, 复杂运算也随之带来分类识别率的下降。因此, 通过合适的特征选择算法, 去除无关特征和冗余特征, 获得有助于分类的最优特征子集, 对提高识别性能和降低计算成本具有重要意义。特征选择主要有两个功能:减少特征数量、降维,使模型泛化能力更强,减少过拟合增
特征选择是模式识别和机器学习领域的重要研究课题之一。为了提高准确率, 人们往往最大限度地提取特征信息。然而, 过大的特征向量维数不仅导致计算成本的增加, 复杂运算也随之带来分类识别率的下降。因此, 通过合适的特征选择算法, 去除无关特征和冗余特征, 获得有助于分类的最优特征子集, 对提高识别性能和降低计算成本具有重要意义。
特征选择主要有两个功能:
- 减少特征数量、降维,使模型泛化能力更强,减少过拟合
- 增强对特征和特征值之间的理解
参考资料:
CSDN《几种常用的特征选择方法》
知乎《机器学习中,有哪些特征选择的工程方法?》

引言
模型输入组合以及适当的训练和测试数据长度是限制非线性模型预测精度的主要因素之一(Choubin and Malekian,2017)。为了确定最佳的输入变量集,用于提取相关性最高变量的常见方法包括:加权法、决策树、变量方差和线性方法【 relief(RL)、random forest(RF)、主成分分析(PCA)、相关分析(COR)等】。
Relief 系列算法是一种特征权重算法,根据各个特征和类别的相关性赋予特征不同的权重,权重小于某个阈值的特征将被移除,算法中特征与类别的相关性是基于特征对近距离样本的区分能力。
PCA(主成分分析,Principal Component Analysis)是一种基于方差的多元统计方法,通过降维技术把多个变量化成少数几个主成分变量,用较少的综合变量去解释原来资料中的大部分信息,即用主成分变量来描述原始变量。
COR方法用于检验数值数据集中输入和输出之间的线性关系。
这里选择几种主流的加以介绍。
1 Relief (RL)
参见《特征选择 Relief 方法》
Relief(RL)是Kira和Rendell(1992)开发的一种分析算法,它采用基于过滤方法的随机搜索方法来选择特征。在降低输入数据维数方面,RL算法遵循四个步骤:(i)给出每个排序相关特征的最终目标,(ii)使用随机选择的样本更新排序,(iii)排序并去除具有最低排序的特征,以及(iv)使用阈值对单个特征进行排序。
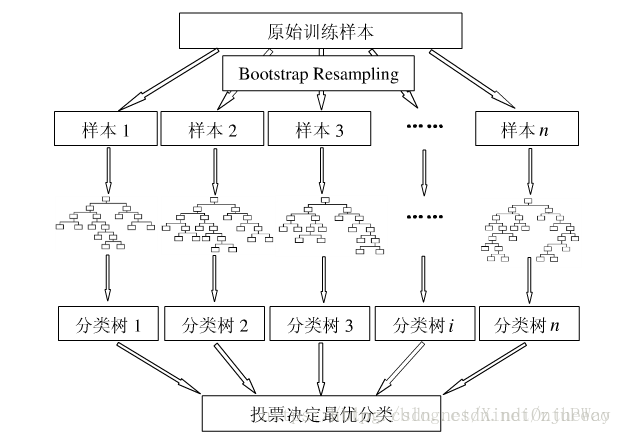
2 random forest(随机森林,RF)
参考资料
利用随机森林选择特征可参看论文Variable selection using Random Forests。用随机森林进行特征重要性评估的思想其实很简单,说白了就是看看每个特征在随机森林中的每颗树上做了多大的贡献,然后取个平均值,最后比一比特征之间的贡献大小。
好了,那么这个贡献是怎么一个说法呢?通常可以用基尼指数(Gini index)或者袋外数据(OOB)错误率作为评价指标来衡量。
2.1 袋外数据错误率
计算某个特征的重要性时,具体步骤如下:
1)对每一颗决策树,选择相应的袋外数据(out of bag,OOB)计算袋外数据误差,记为。
所谓袋外数据是指,每次建立决策树时,通过重复抽样得到一个数据用于训练决策树,这时还有大约1/3的数据没有被利用,没有参与决策树的建立。这部分数据可以用于对决策树的性能进行评估,计算模型的预测错误率,称为袋外数据误差。
这已经经过证明是无偏估计的,所以在随机森林算法中不需要再进行交叉验证或者单独的测试集来获取测试集误差的无偏估计。
2)随机对袋外数据OOB所有样本的特征加入噪声干扰(可以随机改变样本在特征
处的值),再次计算袋外数据误差,记为
。
3)假设森林中有NN棵树,则特征的重要性=
。这个数值之所以能够说明特征的重要性是因为,如果加入随机噪声后,袋外数据准确率大幅度下降(即
上升),说明这个特征对于样本的预测结果有很大影响,进而说明重要程度比较高。
2.2 举个例子
借用利用随机森林对特征重要性进行评估的例子。
以UCI上葡萄酒的例子为例,首先导入数据集。
import pandas as pd
url = 'http://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data'
df = pd.read_csv(url, header = None)
df.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue', 'OD280/OD315 of diluted wines', 'Proline']看下数据的信息
df.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 178 entries, 0 to 177
Data columns (total 14 columns):
Class label 178 non-null int64
Alcohol 178 non-null float64
Malic acid 178 non-null float64
Ash 178 non-null float64
Alcalinity of ash 178 non-null float64
Magnesium 178 non-null int64
Total phenols 178 non-null float64
Flavanoids 178 non-null float64
Nonflavanoid phenols 178 non-null float64
Proanthocyanins 178 non-null float64
Color intensity 178 non-null float64
Hue 178 non-null float64
OD280/OD315 of diluted wines 178 non-null float64
Proline 178 non-null int64
dtypes: float64(11), int64(3)
memory usage: 19.5 KB可见除去class label之外共有13个特征,数据集的大小为178。按照常规做法,将数据集分为训练集和测试集。
from sklearn.cross_validation import train_test_split
from sklearn.ensemble import RandomForestClassifier
x, y = df.iloc[:, 1:].values, df.iloc[:, 0].values
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size = 0.3, random_state = 0)
feat_labels = df.columns[1:]
forest = RandomForestClassifier(n_estimators=10000, random_state=0, n_jobs=-1)
forest.fit(x_train, y_train)
好了,这样一来随机森林就训练好了,其中已经把特征的重要性评估也做好了,我们拿出来看下。
importances = forest.feature_importances_
indices = np.argsort(importances)[::-1]
for f in range(x_train.shape[1]):
print("%2d) %-*s %f" % (f + 1, 30, feat_labels[indices[f]], importances[indices[f]]))输出的结果为:
1) Color intensity 0.182483
2) Proline 0.158610
3) Flavanoids 0.150948
4) OD280/OD315 of diluted wines 0.131987
5) Alcohol 0.106589
6) Hue 0.078243
7) Total phenols 0.060718
8) Alcalinity of ash 0.032033
9) Malic acid 0.025400
10) Proanthocyanins 0.022351
11) Magnesium 0.022078
12) Nonflavanoid phenols 0.014645
13) Ash 0.013916对的就是这么方便。如果要筛选出重要性比较高的变量的话,这么做就可以
threshold = 0.15
x_selected = x_train[:, importances > threshold]
x_selected.shape输出为
(124,3)
3 降维法
当特征选择完成后,可以直接训练模型了,但是可能由于特征矩阵过大,导致计算量大,训练时间长的问题,因此降低特征矩阵维度也是必不可少的。常见的降维方法有主成分分析法(PCA)和线性判别分析(LDA),线性判别分析本身也是一个分类模型。PCA和LDA有很多的相似点,其本质是要将原始的样本映射到维度更低的样本空间中,但是PCA和LDA的映射目标不一样:PCA是为了让映射后的样本具有最大的发散性;而LDA是为了让映射后的样本有最好的分类性能。所以说PCA是一种无监督的降维方法,而LDA是一种有监督的降维方法。
3.1 主成分分析法(PCA)
使用decomposition库的PCA类选择特征的代码如下:
from sklearn.decomposition import PCA
#主成分分析法,返回降维后的数据
#参数n_components为主成分数目
PCA(n_components=2).fit_transform(iris.data)3.2 线性判别分析法(LDA)
使用lda库的LDA类选择特征的代码如下:
from sklearn.lda import LDA
#线性判别分析法,返回降维后的数据
#参数n_components为降维后的维数
LDA(n_components=2).fit_transform(iris.data, iris.target)4 灰色关联度法(Grey relational analysis,GRA )
PDF《2020 Prediction of gas emission based on grey-generalized regression neural network》
百度文库《灰色关联分析方法》
博客《灰色关联分析法步骤》
知网论文《基于LSTM神经网络的柴油机NOx排放预测》
4.1 算法
灰色关联度分析是一种用于多因素分析的统计方法,通过灰色关联度来衡量相似或相异程度。相比其他分析方法,灰色关联度分析在样本多少及样本有无规律的情况下同样适用,因此,将灰色关联度分析用于数据提取以获得模型的输入,减少建模时间,提高计算效率。数据处理的流程如下:
(1)确定灰色关联度分析法的比较矩阵和参考矩阵,即:
通过除以矩阵每列的最大值将量纲归一化为 :
(2)计算灰色关联系数为 :
式中:ρ 为分辨系数,通常为 0.5。
(3)计算灰色关联度,比较矩阵和参考矩阵的灰色关联度为:
根据上式可以得到一系列灰色关 联度,通过灰色关联度的大小次序可以判断出各变量对 输出变量的影响程度。
4.2 应用举例
利用灰色关联分析对6位教师工作状况进行综合分析
1.分析指标包括:专业素质、外语水平、教学工作量、科研成果、论文、著作与出勤.
2.对原始数据经处理后得到以下数值,见下表

3.确定参考数据列:
![]()
4.计算 , 见下表

5.求最值
6.依据式,ρ取0.5计算,得
同理得出其它各值,见下表:

7.分别计算每个人各指标关联系数的均值(关联序):
8.如果不考虑各指标权重(认为各指标同等重要),六个被评价对象由好到劣依次为1号,5号,3号,6号,2号,4号.
即:
5 相关系数法
参考资料:
《Pearson, Spearman, Kendall 三大相关系数简单介绍》
《统计学之三大相关性系数(pearson、spearman、kendall)》
《Python+pandas计算数据相关系数(person、Kendall、spearman)》
使用相关系数法,先要计算各个特征对目标值的相关系数以及相关系数的P值。常见的三个相关性系数(pearson, spearman, kendall)反应的都是两个变量之间变化趋势的方向以及程度,其值范围为-1到+1,0表示两个变量不相关,正值表示正相关,负值表示负相关,值越大表示相关性越强。
5.1 person correlation coefficient(皮尔森相关性系数)(连续变量)
公式如下:
python代码实现:
a) Dataframe.corr(method='pearson') #返回相关关系矩阵
b) from scipy.stats import normaltest, probplot
normaltest(a) #返回统计数和检验P值, 样本要求>20。
probplot(np.array(x,y), dist="norm", plot=pylab) #绘制P图,若在对角线,则相关性强。
5.2 Spearman correlation coefficient(斯皮尔曼相关性系数)(连续变量)
斯皮尔曼相关性系数,通常也叫斯皮尔曼秩相关系数。“秩”,可以理解成就是一种顺序或者排序,那么它就是根据原始数据的排序位置进行求解,这种表征形式就没有了求皮尔森相关性系数时那些限制。下面来看一下它的计算公式:
其中表示数据的数量,
表示两个数据次序的差值:
比如这里有两个变量X、Y,刚开始这两个变量中的值都是随意排放的,位置从1-6。先将它们进行排序:

排序前数字11位置在1,排序后变成了5;490排序前的位置是2,排序后的位置变成了1。以此类推,得到了变量X 和Y排序后的位置。
- X:(5,1,4,2,3,6)
- Y:(6,1,5,2,4,3)
因此第一个位置的di=6-5=1,平方后还是1,以此类推计算所有平方的加和,然后得到斯皮尔曼相关系数:ρs= 1-6*(1+1+1+9)/6*35=0.657。
python代码实现 :
a) Dataframe.corr(method='spearman') #返回相关关系矩阵
b) from scipy.stats import spearmanr
spearmanr(array) #返回 Spearman 系数(系数矩阵)和检验P值, 样本要求>20。
5.3 kendall correlation coefficient(肯德尔相关性系数)(有序分类变量)
肯德尔相关性系数,又称肯德尔秩相关系数,它也是一种秩相关系数,不过它所计算的对象是分类变量。分类变量可以理解成有类别的变量,可以分为
(1)无序的,比如性别(男、女)、血型(A、B、O、AB);
(2)有序的,比如肥胖等级(重度肥胖,中度肥胖、轻度肥胖、不肥胖)。
通常需要求相关性系数的都是有序分类变量。举个例子。比如评委对选手的评分(优、中、差等),我们想看两个(或者多个)评委对几位选手的评价标准是否一致;或者医院的尿糖化验报告,想检验各个医院对尿糖的化验结果是否一致,这时候就可以使用肯德尔相关性系数进行衡量。

python代码实现:
a) Dataframe.corr(method='kendall') #返回相关关系矩阵
b) from scipy.stats import kendalltau
kendalltau(x, y) #返回系数和P值
更多推荐
 已为社区贡献3条内容
已为社区贡献3条内容









所有评论(0)