
简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
R中运行函数enrichPathway时报错如下解决方法,查看help文档,将organism参数值改为mouse

参考https://blog.csdn.net/weixin_44022515/article/details/122022228。#参考https://blog.csdn.net/qq_39241986/article/details/128496017。
画出来的图的纵坐标是按照字母顺序排序的,也可以设置想要的顺序。
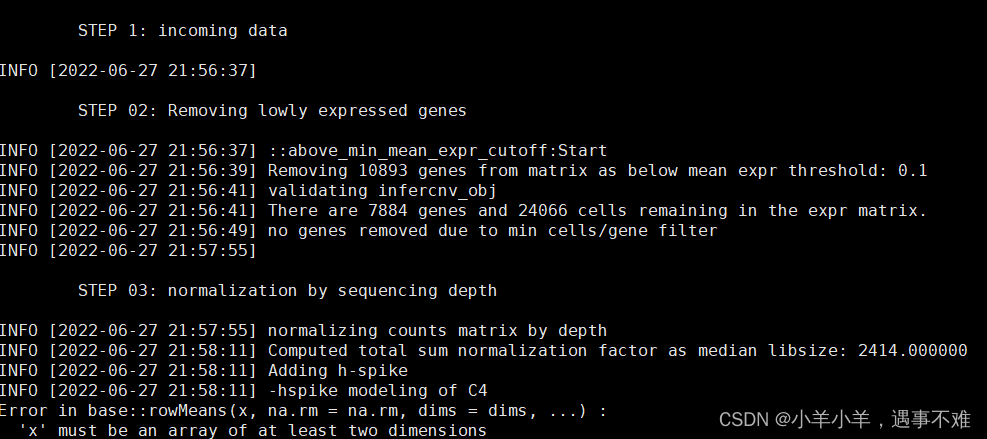
跑infercnv的过程中报错Error in base::rowMeans(x, na.rm = na.rm, dims = dims, ...) :'x' must be an array of at least two dimensionsinfercnv::run函数跑到第三步骤就进行不下去了,网上搜索解决办法,发现github上有人给出建议参考Error in base::rowMean
报错原因github token过期,重新申请一个即可,将新的token保存到R中即可。#再重新运行下载包的代码,下载成功。

在R中做数据筛选时报错Error in `[.data.frame`(all, all$cell_suspension.selected_cell_type == :选择了未定义的列错误原因:需要根据行或者列筛选解决办法:若按行筛选需要用all[#筛选条件,]若按列筛选需要用all[,#筛选条件]注意“,”的位置...

> table(adult1_mut_1$SRR8991002==0)Error in adult1_mut_1$SRR8991002 :$ operator is invalid forR中出现如上报错查看数据类型> class(adult1_mut_1)[1] "matrix" "array"> adult1_mut_1=as.data.frame(adult1_mut_1)










