
简介
该用户还未填写简介
擅长的技术栈
未填写擅长的技术栈
可提供的服务
暂无可提供的服务
Nature methods| 使用scPROTEIN工具让你的单细胞蛋白组学数据分析更上一层楼
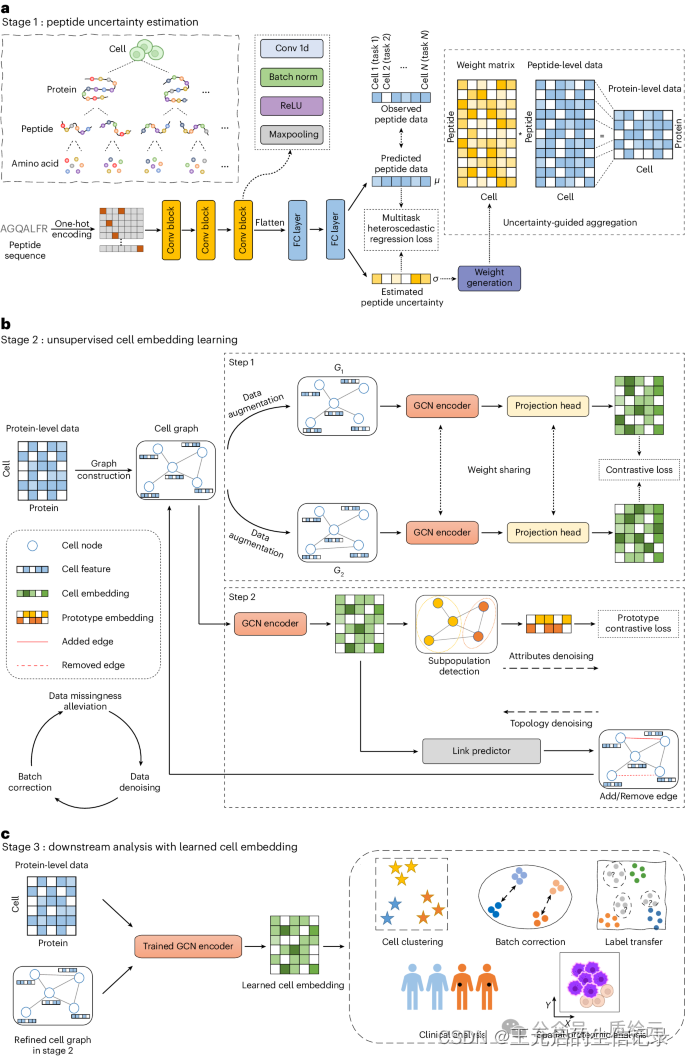
首先,对于提供原始肽信号强度的数据集,我们提出了一个多任务异方差回归模型来估计肽定量的不确定性,并以不确定性引导的方式将肽含量聚合到蛋白质水平(图a)。为了提高构建蛋白质丰度数据的精度,建议采用为肽分配不确定性权重的方法来提高蛋白质丰度数据的准确性。其次,由于样品制备过程中的样品损失,并不是所有的组成肽内容都被输送到质谱仪,这对单细胞蛋白质组学来说是一个至关重要的问题。肽注入质谱仪后,在数据依赖型
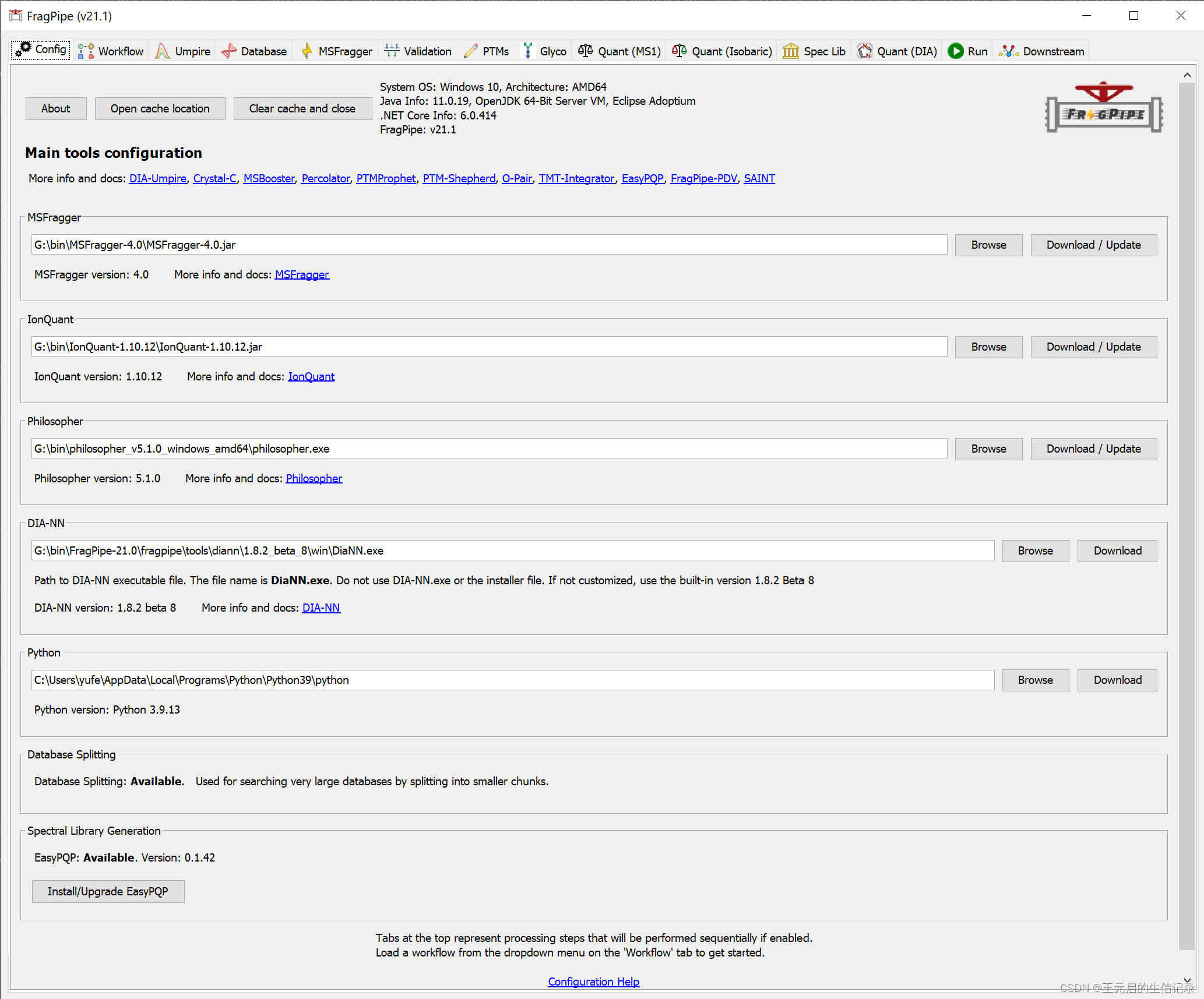
探索深度蛋白质组学的利器:FragPipe
FragPipe是一款强大的Java GUI工具,旨在为基于质谱的蛋白质组学数据分析提供全方位的支持。这款开源项目是Nesvilab实验室的杰作,集成了MSFragger这一超快速的搜索引擎和一系列下游处理工具,如Philosopher,Crystal-C以及PTM-Shepherd,确保了从数据检索到结果解释的全程高效。
MSDIAL|让你轻松学会最简单的代谢/蛋白组学数据处理
今天分享一下代谢组学数据处理常用的一个软件—MSDIAL,该软件可以下载后离线使用,目前是已经更新到MSDIAL5版本,可以针对蛋白组学数据进行处理。可以进行代谢组学、外泌体组学、脂质组学、蛋白组学、糖蛋白组学等分析。新版本对数据依赖性采集和数据非依赖性采集的数据都可进行分析,主要包括GC-MS、LC-MS(DDA)、SWATH-MS、AIF-MS、LC-IM-MS(PASEF)、LC-IM-AI

到底了










