简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
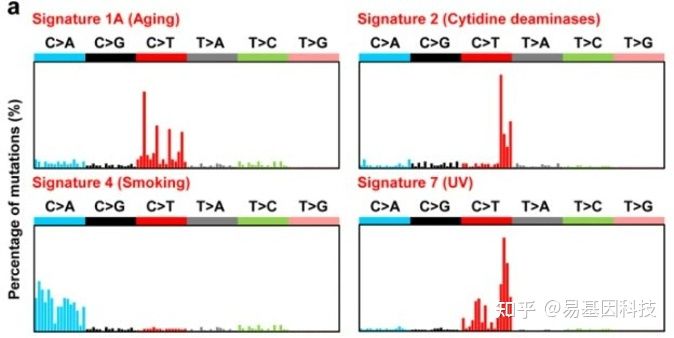
随着宏病毒研究的日益火热,许多小伙伴都在想自己曾经测过的宏基因组数据能否再深挖一波?当然可以!前面几期小编给大家抛砖引玉介绍了组装软件以及病毒数据库的基本知识,这期我们就来聊一聊,如果不依赖于数据库的注释结果,我们可以把病毒序列从宏基因组数据中狙击出来吗?美国南加州大学定量计算生物学中心孙丰珠教授课题组曾在2017年开发并报道了VirFinder[1],该软件可以基于细菌与病毒序列的k-mer差异
摘要目的:本研究是为了评估大肠癌相关的甲基化DNA标志物(MDM)在原发性和转移性大肠癌中的一致性,为利用血浆检测远端复发或转移的大肠癌提供可行性。实验设计:选择了一组已报道的与大肠癌相关的MDM标志物。用甲基化特异性定量PCR检测原发性和转移性大肠癌组织中的MDM。用靶标富集长探针定量放大信号法测定血浆中的MDMs。用随机森林模型对原发性大肠癌患者血浆样本中MDM的进行预测。该模型算法在预先采集
近日,美国波士顿东北大学Miten Jain团队评估了牛津纳米孔测序(Oxford Nanopore Technologies, ONT)技术在检测人类原代组织DNA甲基化中的性能,特别比较了 ONT R9和升级版R10测序芯片在检测人体外细胞系、脑组织和血液样本的5-甲基胞嘧啶(5-methylcytosine, 5mC)数据差异,并与其他测序技术(PacBio长读长测序和Illumina亚硫酸

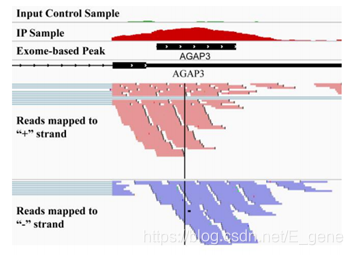
大家好,这里是易基因,此前我们已讲解许多关于RNA甲基化相关实验设计和研究思路套路,今天,我们来点新干货,从生物信息学分析的角度来独家解析MeRIP-Seq数据的分析利器——exome Peak。通过exomePeak包能够进行RNA甲基化位点peak鉴定、peak定量以及差异分析,一起来看看这款经典软件的内部原理吧。关于exomePeakexomePeak是目前主流的MeRIP-seq(m6A-
随着宏病毒研究的日益火热,许多小伙伴都在想自己曾经测过的宏基因组数据能否再深挖一波?当然可以!前面几期小编给大家抛砖引玉介绍了组装软件以及病毒数据库的基本知识,这期我们就来聊一聊,如果不依赖于数据库的注释结果,我们可以把病毒序列从宏基因组数据中狙击出来吗?美国南加州大学定量计算生物学中心孙丰珠教授课题组曾在2017年开发并报道了VirFinder[1],该软件可以基于细菌与病毒序列的k-mer差异
随着宏病毒研究的日益火热,许多小伙伴都在想自己曾经测过的宏基因组数据能否再深挖一波?当然可以!前面几期小编给大家抛砖引玉介绍了组装软件以及病毒数据库的基本知识,这期我们就来聊一聊,如果不依赖于数据库的注释结果,我们可以把病毒序列从宏基因组数据中狙击出来吗?美国南加州大学定量计算生物学中心孙丰珠教授课题组曾在2017年开发并报道了VirFinder[1],该软件可以基于细菌与病毒序列的k-mer差异
上期介绍完《基于深度学习的病毒序列识别》后,不少小伙伴后台私信小编要求讲解一下DeepVirFinder使用指南,这里就给大家安排上。DeepVirFinder[1]使用深度学习方法预测病毒序列。该方法对短病毒序列具有较好的预测精度,可用于从宏基因组数据中预测序病毒列。与基于k-mer的VirFinder方法相比,DeepVirFinder通过使用卷积神经网络(CNN)显著提高了预测精度。CNN可
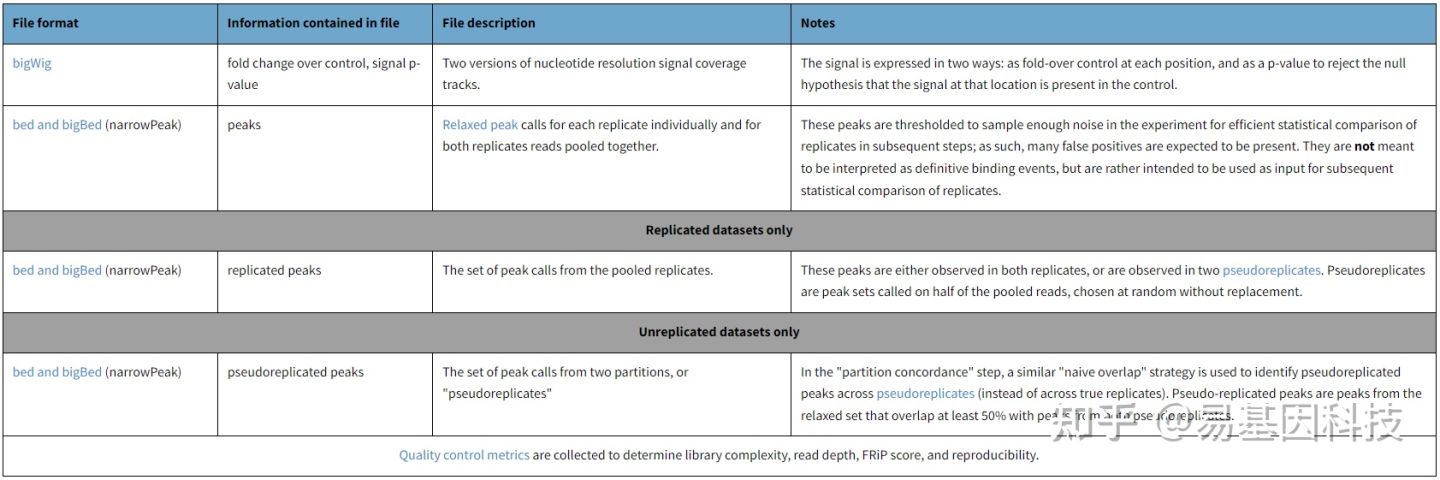
以表观组学研究中的ChIP-seq为例,ENCODEConsortium使用不同的表观基因组分析,并制定对应的分析方案和指南,具有绝对的权威和参考意义。组蛋白ChIP-seq和转录因子ChIP-seq的流程具有相同的比对步骤,但在信号和peakcalling方法以及随后的重复统计处理方面有所不同。组蛋白ChIP-seq和转录因子ChIP-seq的流程具有相同的比对步骤,但在信号和peakcalli
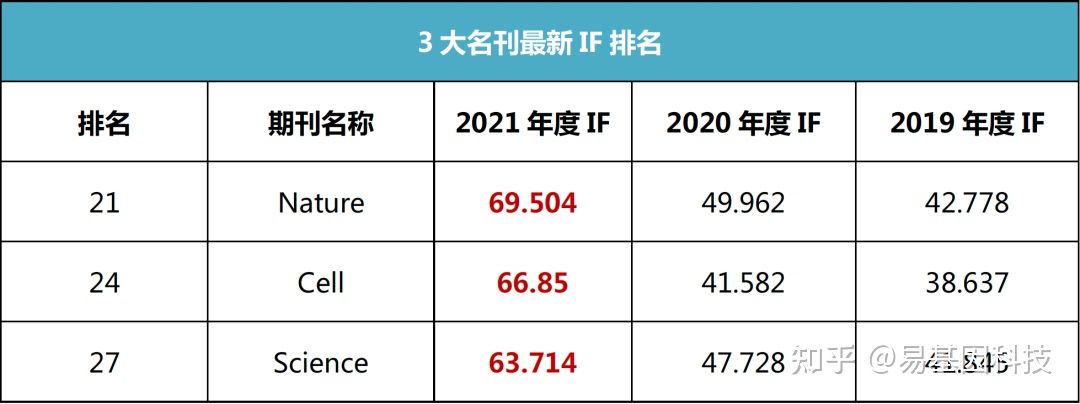
北京时间2022年6月28日,Clarivate Analytics官方发布最新年度(2021年)期刊引用报告(JCR),全球期刊影响因子(Impact factor,缩写IF)出炉。世界公认的三大名刊NCS世界公认的三大名刊主要是指:Nature,Cell,Science。整体来说今年三大刊的表现非常亮眼,影响因子分数均有较大提升,其中cell上升分数最多。其中Nature最新IF 69.504
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。DNA甲基化(DNA Methylation)能够在不改变DNA序列的前提下,把甲基添加到DNA分子上,从而实现改变遗传表现的效果。DNA甲基化后,尽管核苷酸顺序未变,但是可能导致染色体的结构发生改变,有可能通过关闭抑癌基因,从而导致肿瘤发生。近年来的大量研究表明,DNA异常甲基化与肿瘤的发生、发展、细胞癌变有着密切的联系。DNA甲基化