使用 SciKit-learn 和 SciPy 构建/搜索 K-Nearest-Neighbour 的速度
问题:使用 SciKit-learn 和 SciPy 构建/搜索 K-Nearest-Neighbour 的速度
我有一大组二维点,希望能够快速查询二维空间中任何点的 k 最近邻的集合。由于它是低维的,KD-Tree 似乎是一个很好的方法。我的初始数据集只会很少更新,因此查询点的时间对我来说应该比构建时间更重要。但是,每次我运行程序时,我都需要重新加载对象,所以我还需要一个可以快速保存和重新加载的结构。
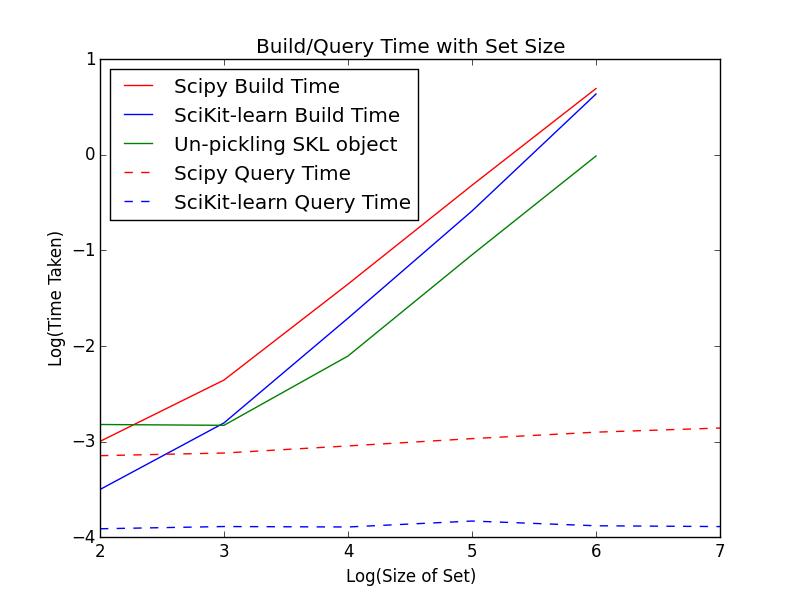
两种现成的选择是 SciPy 和 SciKit-learn 中的 KDTree 结构。下面我介绍了其中两个在大范围列表长度上的构建速度和查询速度。我还对 SciKit-learn 结构进行了腌制,并显示了从腌制中重新加载对象的时间。这些在图表中进行比较,用于生成时序的代码包含在下面。
正如我在图中所示,对于大 N,从 pickle 加载比从头开始构建要快半个数量级,这表明 KDTree 适合我的用例(即频繁重新加载但不经常重新构建)。
比较构建时间的代码:
# Profiling the building time for the two KD-tree structures and re-loading from a pickle
import math, timeit, pickle, sklearn.neighbors
the_lengths = [100, 1000, 10000, 100000, 1000000]
theSciPyBuildTime = []
theSklBuildTime = []
theRebuildTime = []
for length in the_lengths:
dim = 5*int(math.sqrt(length))
nTimes = 50
from random import randint
listOfRandom2DPoints = [ [randint(0,dim),randint(0,dim)] for x in range(length)]
setup = """import scipy.spatial
import sklearn.neighbors
length = """ + str(length) + """
dim = """ + str(dim) + """
from random import randint
listOfRandom2DPoints = [ [randint(0,dim),randint(0,dim)] for x in range(length)]"""
theSciPyBuildTime.append( timeit.timeit('scipy.spatial.KDTree(listOfRandom2DPoints, leafsize=20)', setup=setup, number=nTimes)/nTimes )
theSklBuildTime.append( timeit.timeit('sklearn.neighbors.KDTree(listOfRandom2DPoints, leaf_size=20)', setup=setup, number=nTimes)/nTimes )
theTreeSkl = sklearn.neighbors.KDTree(listOfRandom2DPoints, leaf_size=20, metric='euclidean')
f = open('temp.pkl','w')
temp = pickle.dumps(theTreeSkl)
theRebuildTime.append( timeit.timeit('pickle.loads(temp)', 'from __main__ import pickle,temp', number=nTimes)/nTimes )
比较查询时间的代码:
# Profiling the query time for the two KD-tree structures
import scipy.spatial, sklearn.neighbors
the_lengths = [100, 1000, 10000, 100000, 1000000, 10000000]
theSciPyQueryTime = []
theSklQueryTime = []
for length in the_lengths:
dim = 5*int(math.sqrt(length))
nTimes = 50
listOfRandom2DPoints = [ [randint(0,dim),randint(0,dim)] for x in range(length)]
setup = """from __main__ import sciPiTree,sklTree
from random import randint
length = """ + str(length) + """
randPoint = [randint(0,""" + str(dim) + """),randint(0,""" + str(dim) + """)]"""
sciPiTree = scipy.spatial.KDTree(listOfRandom2DPoints, leafsize=20)
sklTree = sklearn.neighbors.KDTree(listOfRandom2DPoints, leaf_size=20)
theSciPyQueryTime.append( timeit.timeit('sciPiTree.query(randPoint,10)', setup=setup, number=nTimes)/nTimes )
theSklQueryTime.append( timeit.timeit('sklTree.query(randPoint,10)', setup=setup, number=nTimes)/nTimes )
问题:
-
结果:尽管对于非常大的 N,它们越来越接近,但 SciKit-learn 似乎在构建时间和查询时间上都击败了 SciPy。其他人有没有发现这个?
-
数学:有没有更好的结构可用于此?我只在 2D 空间中工作(虽然数据会非常密集,所以蛮力已经出来了),是否有更好的低维 kNN 搜索结构?
-
速度:看起来这两种方法的构建时间在大 N 处越来越近,但我的计算机放弃了我 - 任何人都可以为我验证更大的 N 吗?!谢谢!!重建时间是否也继续大致呈线性增长?
-
实用性:SciPy KDTree 不会腌制。正如这篇文章中所报告的那样,我收到以下错误“PicklingError: Can't pickle : it's not found as scipy.spatial.kdtree.innernode” - 我认为这是因为它是一个嵌套结构。根据这篇文章中报告的答案,嵌套结构可以用莳萝腌制。然而,莳萝给了我同样的错误——为什么会这样?
解答
在我得到答案之前,我想指出,当您有一个使用大量数字的程序时,您应该始终使用numpy 库中的numpy.array来存储此类数据。我不知道你使用的是什么版本的 Python,scikit-learn和SciPy,但我使用的是 Python 3.7.3、scikit-learn 0.21.3 和 SciPy 1.3.0。当我运行您的代码来比较构建时间时,我得到了AttributeError: 'list' object has no attribute 'size'。这个错误是说列表listOfRandom2DPoints没有属性size。问题是sklearn.neighbors.KDTree期望numpy.array具有属性size。类scipy.spatial.KDTree适用于 Python 列表,但正如您在scipy.spatial.KDTree](https://github.com/scipy/scipy/blob/v1.4.1/scipy/spatial/kdtree.py#L241-L942)类的__init__方法的[源代码中看到的那样,第一行是self.data = np.asarray(data),这意味着数据将被转换为numpy.array。
正因为如此,我改变了你的台词:
from random import randint
listOfRandom2DPoints = [ [randint(0,dim),randint(0,dim)] for x in range(length)]
至:
import numpy as np
ListOfRandom2DPoints = np.random.randint(0, dim, size=(length, 2))
(此更改不会影响速度比较,因为更改是在设置代码中进行的。)
现在回答你的问题:
- 就像你说的,scikit-learn 似乎在构建时比 SciPy 更胜一筹。出现这种情况的原因并不是scikit-learn算法更快,而是
sklearn.neighbors.KDTree是在Cython中实现的(链接到源码),而scipy.spatial.KDTree是用纯Python代码编写的(链接到源码))。
(如果你不知道 Cython 是什么,一个过于简单的解释是 Cython 使得用 Python 编写 C 代码成为可能,这样做的主要原因是 C 比 Python 快得多)
SciPy 库在 Cythonscipy.spatial.cKDTree中也有实现(链接到源代码),它的工作方式与scipy.spatial.KDTree相同,如果您比较sklearn.neighbors.KDTree和scipy.spatial.cKDTree的构建时间:
timeit.timeit('scipy.spatial.cKDTree(npListOfRandom2DPoints, leafsizeu003d20)', setupu003dsetup, numberu003dnTimes)
timeit.timeit('sklearn.neighbors.KDTree(npListOfRandom2DPoints, leaf_sizeu003d20)', setupu003dsetup, numberu003dnTimes)
构建时间非常相似,当我运行代码时,scipy.spatial.cKDTree稍微快了一点(大约 20%)。
与查询时间情况非常相似,scipy.spatial.KDTree(纯 Python 实现)比sklearn.neighbors.KDTree(Cython 实现)慢十倍左右,而scipy.spatial.cKDTree(Cython 实现)几乎与sklearn.neighbors.KDTree一样快。我已经测试了最多 N u003d 10000000 的查询时间,并得到了与您相同的结果。无论 N 多少,查询时间都保持不变(这意味着scipy.spatial.KDTree的查询时间对于 N u003d 1000 和 N u003d 1000000 是相同的,对于sklearn.neighbors.KDTree和scipy.spatial.cKDTree的查询时间也是一样的)。这是因为查询(搜索)时间复杂度为 O(logN),即使 N u003d 1000000,logN 也非常小,因此差异太小而无法测量。
2.构建sklearn.neighbors.KDTree算法(__init__类方法)的时间复杂度为O(KNlogN)(关于scikit-learn最近邻算法)所以在你的情况下它会是O(2NlogN),实际上是O(NlogN) .基于sklearn.neighbors.KDTree和scipy.spatial.cKDTree的构建时间非常相似,我假设scipy.spatial.cKDTree的构建算法也具有 O(NlogN) 的时间复杂度。我不是最近邻搜索算法方面的专家,但基于一些在线搜索,我会说对于低维最近邻搜索算法来说,这是尽可能快的。如果你去最近邻搜索维基百科页面你会看到有精确方法和近似方法。k-d 树是精确方法,它是空间划分方法的子类型。在所有空间划分方法中(仅基于 Wikipedia 页面的最近邻搜索的快速精确方法),k-d 树是静态上下文中用于最近邻搜索的低维欧几里得空间的最佳方法(没有很多插入和删除)。此外,如果您查看邻近邻域图中贪婪搜索下的近似方法,您将看到“邻近图方法被认为是近似最近邻搜索的当前最新技术”。当您查看为此方法引用的研究文章时(使用分层可导航小世界图进行高效且稳健的近似最近邻搜索),您会发现此方法的时间复杂度为 O(NlogN)。这意味着对于低维空间,k-d 树(精确方法)与近似方法一样快。目前,我们已经比较了用于最近邻搜索的结构的构建(构建)时间复杂度。所有这些算法的搜索(查询)时间复杂度为 O(logN)。所以我们能得到的最好的结果是构建 O(NlogN) 的复杂度和 O(logN) 的查询复杂度,这就是我们在 k-d 树方法中所拥有的。因此,根据我的研究,我会说 k-d 树是低维最近邻搜索的最佳结构。
(我认为如果有更好(更快)的方法来进行最近邻搜索,那么 scikit-learn 和 SciPy 会实现该方法。同样从理论的角度来看,知道最快的排序算法的时间复杂度为 O(NlogN),拥有时间复杂度小于 O(NlogN) 的最近邻搜索构建算法将是相当令人惊讶的。)
- 就像我说的,您将
sklearn.neighbors.KDTree与 Cython 实现进行比较,将scipy.spatial.KDTree与纯 Python 实现进行比较。理论上sklearn.neighbors.KDTree应该比scipy.spatial.KDTree快,我将它们与 1000000 进行了比较,它们似乎在大 N 处更接近。对于 N u003d 100,scipy.spatial.KDTree比sklearn.neighbors.KDTree慢约 10 倍,对于 N u003d 1000000,scipy.spatial.KDTree慢约两倍如sklearn.neighbors.KDTree。我不确定为什么会发生这种情况,但我怀疑对于大 N,内存成为比操作数量更大的问题。
我检查了重建时间也高达 1000000,它确实线性增加,这是因为函数pickle.loads的持续时间与加载对象的大小成线性比例。
- 对我来说,
sklearn.neighbors.KDTree、scipy.spatial.KDTree和scipy.spatial.cKDTree的酸洗工作,所以我无法重现你的错误。我猜问题是你有旧版本的 SciPy,所以将 SciPy 更新到最新版本应该可以解决这个问题。
(如果您在这个问题上需要更多帮助,您应该在您的问题中添加更多信息。您的 Python 和 SciPy 版本是什么,重现此错误的确切代码以及完整的错误消息?)
更多推荐
 已为社区贡献126445条内容
已为社区贡献126445条内容







所有评论(0)