scipy.cluster.hierarchy 教程 [关闭]
·
问题:scipy.cluster.hierarchy 教程 [关闭]
我试图了解如何操作层次结构集群,但文档太......技术性?......我不明白它是如何工作的。
是否有任何教程可以帮助我开始,逐步解释一些简单的任务?
假设我有以下数据集:
a = np.array([[0, 0 ],
[1, 0 ],
[0, 1 ],
[1, 1 ],
[0.5, 0 ],
[0, 0.5],
[0.5, 0.5],
[2, 2 ],
[2, 3 ],
[3, 2 ],
[3, 3 ]])
我可以轻松地进行层次聚类并绘制树状图:
z = linkage(a)
d = dendrogram(z)
-
现在,如何恢复特定集群?假设在树状图中包含元素
[0,1,2,4,5,6]的那个? -
我怎样才能取回那些元素的值?
解答
层次凝聚聚类(HAC)分为三个步骤:
- 量化数据(
metric参数)
2.集群数据(method参数)
3.选择集群数量
正在做
z = linkage(a)
将完成前两个步骤。由于您没有指定任何参数,因此它使用标准值
1.metric = 'euclidean'
2.method = 'single'
所以z = linkage(a)会给你一个a的单链接层次凝聚聚类。这种聚类是一种解决方案的层次结构。从这个层次结构中,您可以获得有关数据结构的一些信息。你现在可以做的是:
-
检查哪个
metric是合适的,例如。 G。cityblock或chebychev将以不同方式量化您的数据(cityblock、euclidean和chebychev对应于L1、L2和L_inf范数) -
检查
methdos的不同属性/行为(例如single、complete和average) -
检查如何确定簇数,例如。 G。通过阅读关于它的 wiki
-
计算找到的解决方案(聚类)的索引,例如轮廓系数(通过该系数,您可以获得关于点/观测与聚类分配给它的聚类的拟合程度的质量反馈)。不同的索引使用不同的标准来限定聚类。
这是开始的事情
import numpy as np
import scipy.cluster.hierarchy as hac
import matplotlib.pyplot as plt
a = np.array([[0.1, 2.5],
[1.5, .4 ],
[0.3, 1 ],
[1 , .8 ],
[0.5, 0 ],
[0 , 0.5],
[0.5, 0.5],
[2.7, 2 ],
[2.2, 3.1],
[3 , 2 ],
[3.2, 1.3]])
fig, axes23 = plt.subplots(2, 3)
for method, axes in zip(['single', 'complete'], axes23):
z = hac.linkage(a, method=method)
# Plotting
axes[0].plot(range(1, len(z)+1), z[::-1, 2])
knee = np.diff(z[::-1, 2], 2)
axes[0].plot(range(2, len(z)), knee)
num_clust1 = knee.argmax() + 2
knee[knee.argmax()] = 0
num_clust2 = knee.argmax() + 2
axes[0].text(num_clust1, z[::-1, 2][num_clust1-1], 'possible\n<- knee point')
part1 = hac.fcluster(z, num_clust1, 'maxclust')
part2 = hac.fcluster(z, num_clust2, 'maxclust')
clr = ['#2200CC' ,'#D9007E' ,'#FF6600' ,'#FFCC00' ,'#ACE600' ,'#0099CC' ,
'#8900CC' ,'#FF0000' ,'#FF9900' ,'#FFFF00' ,'#00CC01' ,'#0055CC']
for part, ax in zip([part1, part2], axes[1:]):
for cluster in set(part):
ax.scatter(a[part == cluster, 0], a[part == cluster, 1],
color=clr[cluster])
m = '\n(method: {})'.format(method)
plt.setp(axes[0], title='Screeplot{}'.format(m), xlabel='partition',
ylabel='{}\ncluster distance'.format(m))
plt.setp(axes[1], title='{} Clusters'.format(num_clust1))
plt.setp(axes[2], title='{} Clusters'.format(num_clust2))
plt.tight_layout()
plt.show()
给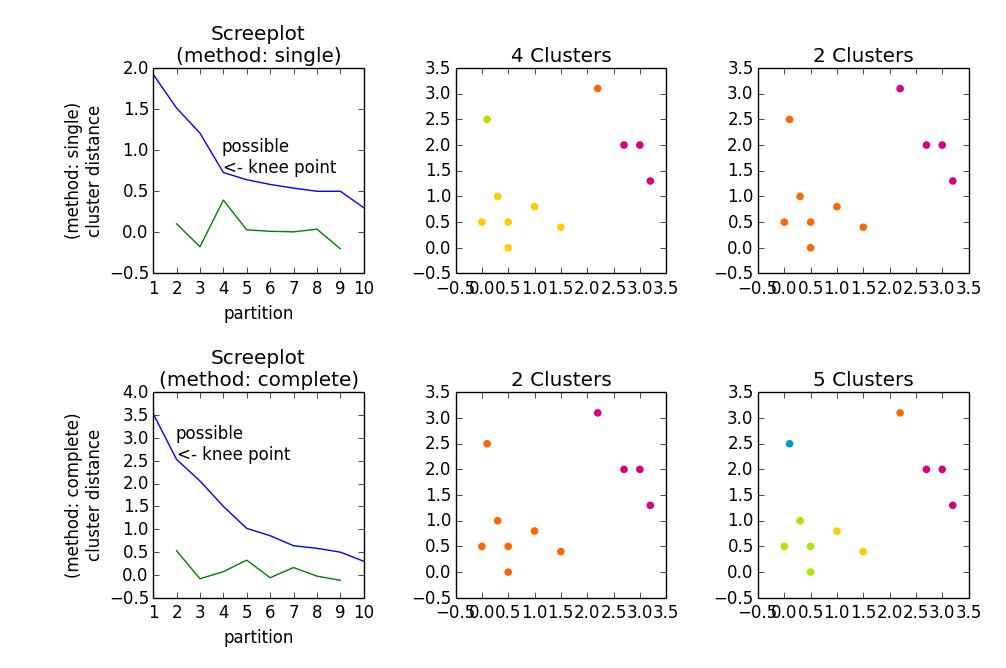
更多推荐
 已为社区贡献126445条内容
已为社区贡献126445条内容







所有评论(0)