更改 seaborn.clustermap 中 ytick 标签的颜色
·
回答问题
是否可以更改 seaborn.clustermap 中 ytick 标签的颜色?
因此,对于seaborn Iris 示例,可以根据物种设置行颜色并绘制集群图:
import seaborn as sns
iris = sns.load_dataset("iris")
species = iris.pop("species")
lut = dict(zip(species.unique(), "rbg"))
row_colors = species.map(lut)
g = sns.clustermap(iris)
并且可以在绘制的行和行标签之间获得 1-1 对应关系:
g.ax_heatmap.yaxis.get_majorticklabels()
无论如何,是否可以使用它根据行_colors 重新着色 ytick 标签?
Answers
我在尝试做同样的事情时发现了这个问题,在进一步查看这个答案@tom 让我走上了自定义刻度标签的轨道后。
您需要通过将刻度标签中的文本映射回颜色字典来为每个单独的刻度指定颜色。
要访问每个刻度,请访问g.ax_heatmap.axes.get_yticklabels(),然后使用get_text()提取刻度的文本。
在 Iris 数据集的特定示例中,刻度标签文本是熊猫系列species的索引,要收集species文本,该文本允许您从lut字典中恢复颜色,您需要将索引转换回int并使用.loc来提取所需的信息。
iris = sns.load_dataset("iris")
species = iris.pop("species")
lut = dict(zip(species.unique(), "rbg"))
row_colors = species.map(lut)
g = sns.clustermap(iris, row_colors=row_colors)
for tick_label in g.ax_heatmap.axes.get_yticklabels():
tick_text = tick_label.get_text()
species_name = species.loc[int(tick_text)]
tick_label.set_color(lut[species_name])
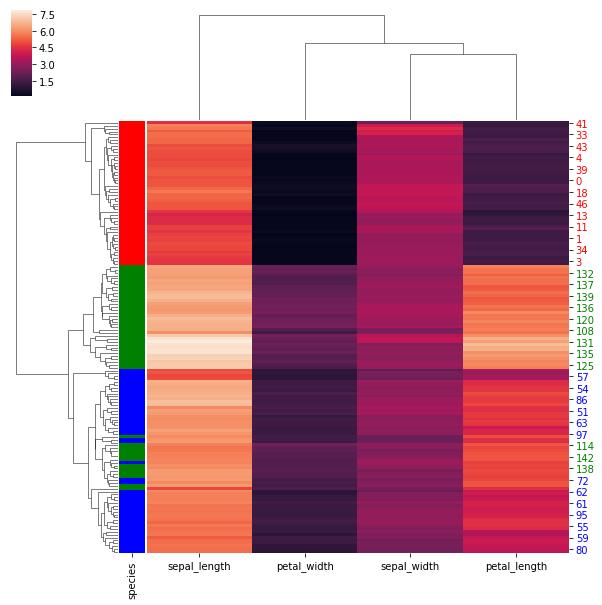
您会注意到树状图的颜色和 y 轴刻度标签(例如 97 和 114)的颜色有些“不匹配”,这是由于图形的大小。使用figsize增加大小可以让您发现隐藏的刻度。 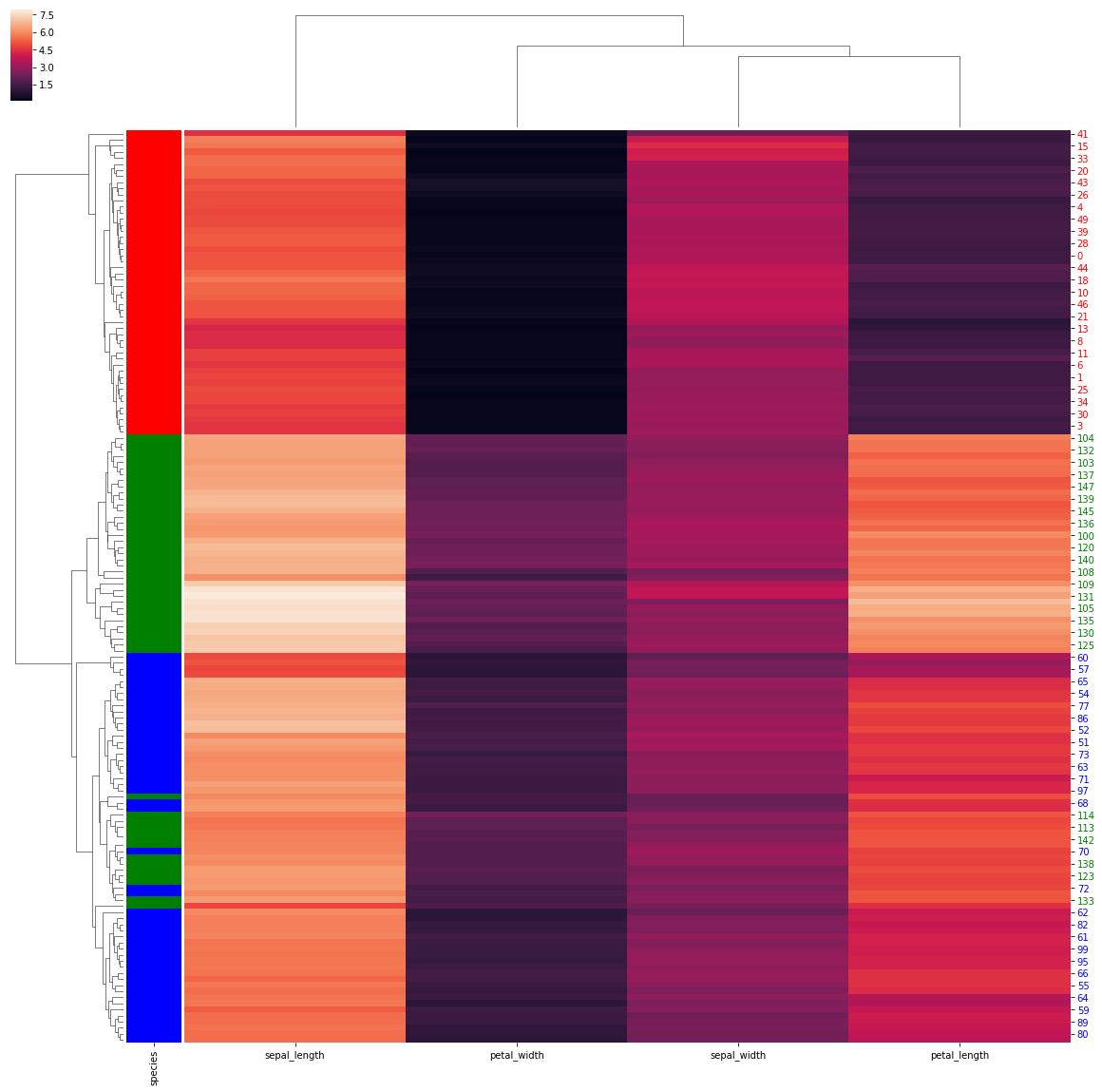
更多推荐
 已为社区贡献126445条内容
已为社区贡献126445条内容







所有评论(0)