将曲线拟合到散点图的边界
回答问题 我正在尝试将曲线拟合到散点图的边界。请参阅此图像以供参考。 我已经使用以下(简化的)代码完成了拟合。它将数据帧切成小的垂直条,然后在那些宽度为width的条中找到最小值,忽略nans。 (函数是单调递减的。) def func(val): """ returns some function of 'val'""" return val * 2 for i in range(0, max_
回答问题
我正在尝试将曲线拟合到散点图的边界。请参阅此图像以供参考。 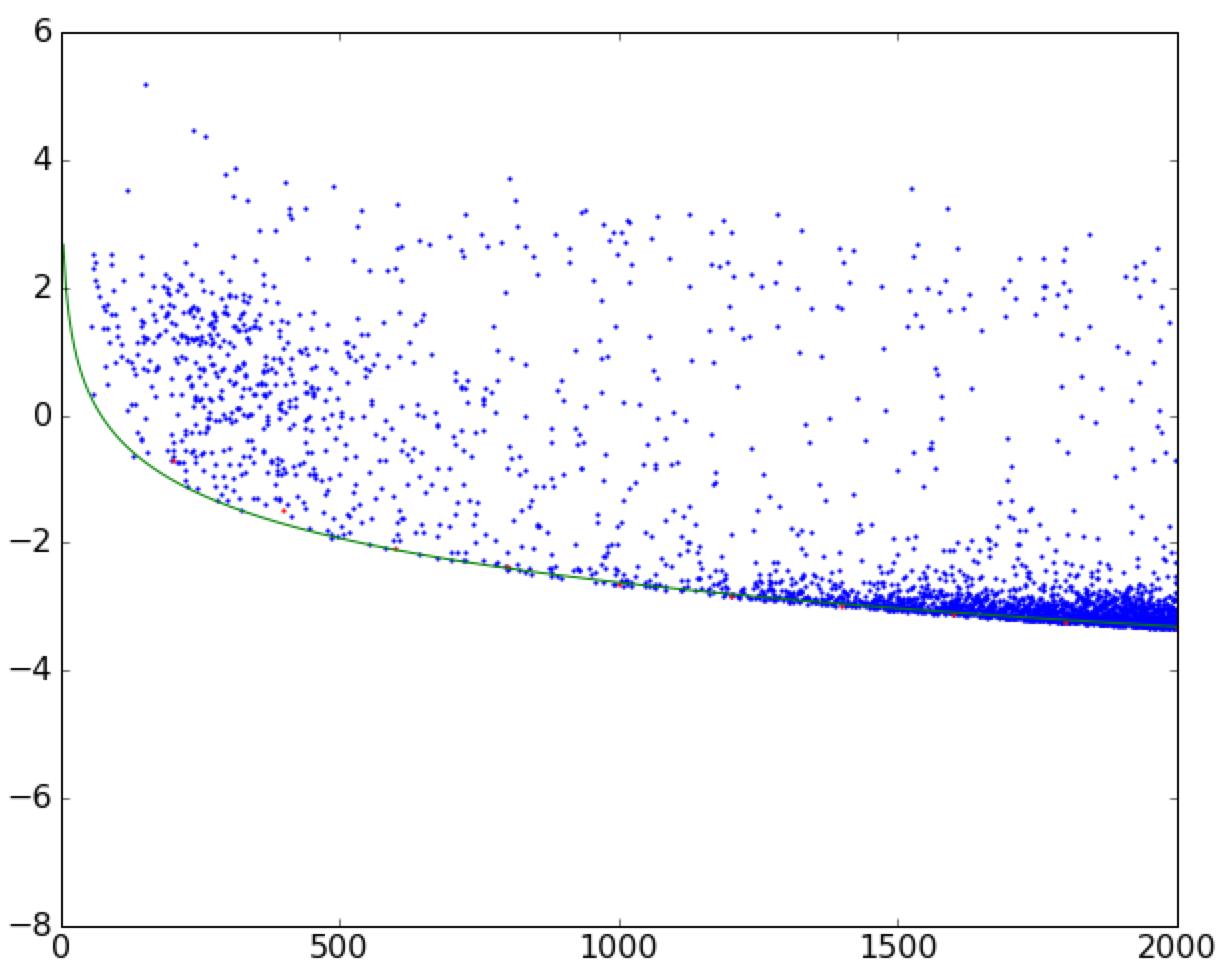
我已经使用以下(简化的)代码完成了拟合。它将数据帧切成小的垂直条,然后在那些宽度为width的条中找到最小值,忽略nans。 (函数是单调递减的。)
def func(val):
""" returns some function of 'val'"""
return val * 2
for i in range(0, max_val, width)):
_df = df[(df.val > i) & (df.val < i + width)] # vertical slice
if np.isnan(np.min(func(_df.val)): # ignore nans
continue
xs.append(i + width)
ys.append(np.min(func(_df.val)))
然后我正在与scipy.optimize.curve_fit配合。我的问题是:有没有更自然或 Pythonic 的方式来做到这一点 - 有没有什么办法可以提高准确性? (例如,通过对点密度较高的散点图区域赋予更高的权重?)
Answers
我发现这个问题真的很有趣,所以我决定试一试。我不知道pythonic或natural,但我想我找到了一种更准确的方法,可以在使用来自_every_点的信息时将边缘拟合到像你这样的数据集。
首先,让我们生成一个看起来像您展示的随机数据。这部分可以很容易地跳过,我只是简单地发布它,以便代码完整且可重现。我使用了两个二元正态分布来模拟这些过密度,并在它们上面撒上一层均匀分布的随机点。然后将它们添加到与您类似的线方程中,并且将线下的所有内容都切断,最终结果如下所示: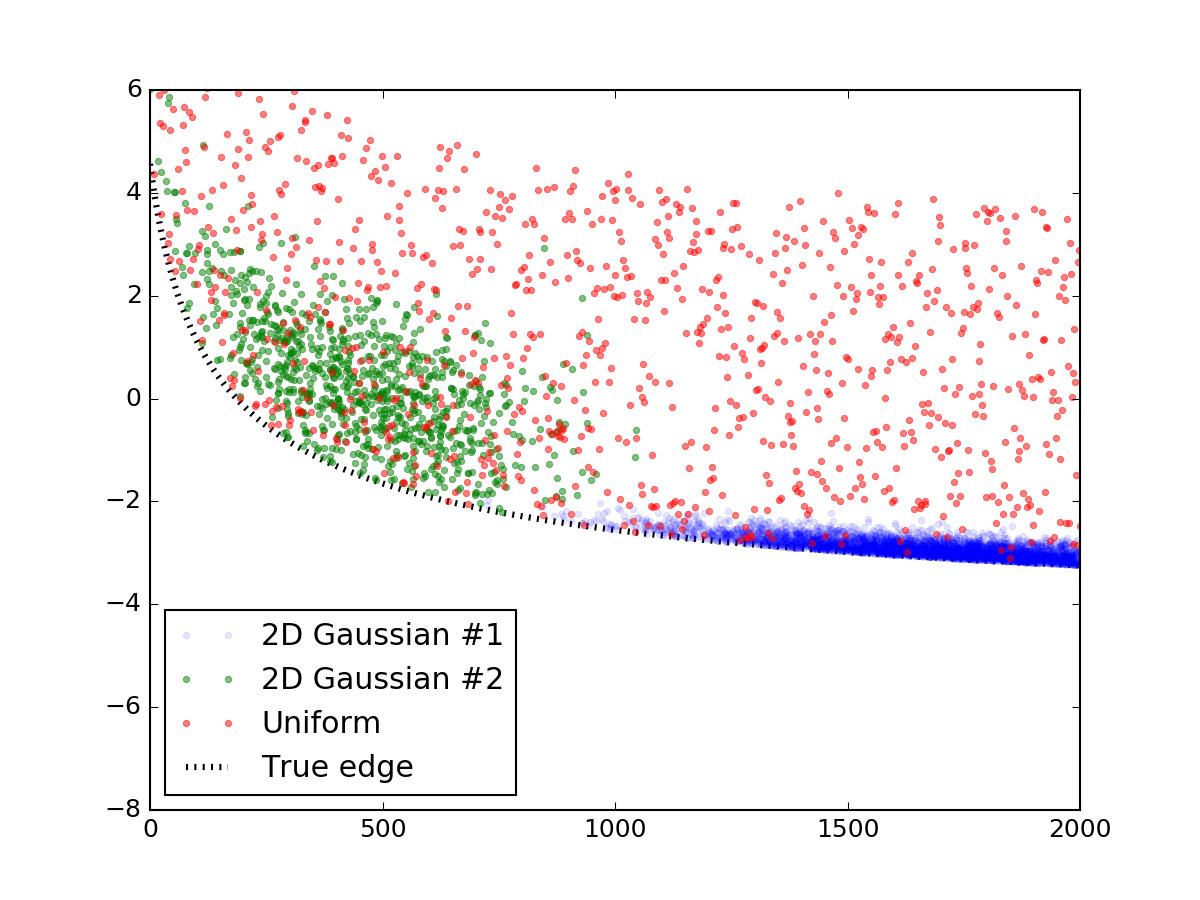
这是制作它的代码片段:
import numpy as np
x_res = 1000
x_data = np.linspace(0, 2000, x_res)
# true parameters and a function that takes them
true_pars = [80, 70, -5]
model = lambda x, a, b, c: (a / np.sqrt(x + b) + c)
y_truth = model(x_data, *true_pars)
mu_prim, mu_sec = [1750, 0], [450, 1.5]
cov_prim = [[300**2, 0 ],
[ 0, 0.2**2]]
# covariance matrix of the second dist is trickier
cov_sec = [[200**2, -1 ],
[ -1, 1.0**2]]
prim = np.random.multivariate_normal(mu_prim, cov_prim, x_res*10).T
sec = np.random.multivariate_normal(mu_sec, cov_sec, x_res*1).T
uni = np.vstack([x_data, np.random.rand(x_res) * 7])
# censoring points that will end up below the curve
prim = prim[np.vstack([[prim[1] > 0], [prim[1] > 0]])].reshape(2, -1)
sec = sec[np.vstack([[sec[1] > 0], [sec[1] > 0]])].reshape(2, -1)
# rescaling to data
for dset in [uni, sec, prim]:
dset[1] += model(dset[0], *true_pars)
# this code block generates the figure above:
import matplotlib.pylab as plt
plt.figure()
plt.plot(prim[0], prim[1], '.', alpha=0.1, label = '2D Gaussian #1')
plt.plot(sec[0], sec[1], '.', alpha=0.5, label = '2D Gaussian #2')
plt.plot(uni[0], uni[1], '.', alpha=0.5, label = 'Uniform')
plt.plot(x_data, y_truth, 'k:', lw = 3, zorder = 1.0, label = 'True edge')
plt.xlim(0, 2000)
plt.ylim(-8, 6)
plt.legend(loc = 'lower left')
plt.show()
# mashing it all together
dset = np.concatenate([prim, sec, uni], axis = 1)
现在我们有了数据和模型,我们可以集思广益如何拟合点分布的边缘。常用的回归方法,如非线性最小二乘scipy.optimize.curve_fit,取数据值y并优化模型的自由参数,使y和model(x)之间的残差最小。非线性最小二乘法是一个迭代过程,它试图在每一步摆动曲线参数以改善每一步的拟合。现在很明显,这是我们_不_想要做的一件事,因为我们希望我们的最小化例程使我们尽可能远离最佳拟合曲线(但不是_太_太远)。
因此,让我们考虑以下功能。它不仅会简单地返回残差,还会在迭代的每一步“翻转”曲线上方的点并将它们也考虑在内。这样,曲线下方的点实际上总是比上方的点多,导致曲线在每次迭代时都向下移动!一旦达到最低点,就找到了函数的最小值,散布的边缘也是如此。当然,这种方法假设您在曲线下方没有异常值 - 但是您的数字似乎并没有受到太大影响。
以下是实现这个想法的函数:
def get_flipped(y_data, y_model):
flipped = y_model - y_data
flipped[flipped > 0] = 0
return flipped
def flipped_resid(pars, x, y):
"""
For every iteration, everything above the currently proposed
curve is going to be mirrored down, so that the next iterations
is going to progressively shift downwards.
"""
y_model = model(x, *pars)
flipped = get_flipped(y, y_model)
resid = np.square(y + flipped - y_model)
#print pars, resid.sum() # uncomment to check the iteration parameters
return np.nan_to_num(resid)
让我们看看如何查找上面的数据:
# plotting the mock data
plt.plot(dset[0], dset[1], '.', alpha=0.2, label = 'Test data')
# mask bad data (we accidentaly generated some NaN values)
gmask = np.isfinite(dset[1])
dset = dset[np.vstack([gmask, gmask])].reshape((2, -1))
from scipy.optimize import leastsq
guesses =[100, 100, 0]
fit_pars, flag = leastsq(func = flipped_resid, x0 = guesses,
args = (dset[0], dset[1]))
# plot the fit:
y_fit = model(x_data, *fit_pars)
y_guess = model(x_data, *guesses)
plt.plot(x_data, y_fit, 'r-', zorder = 0.9, label = 'Edge')
plt.plot(x_data, y_guess, 'g-', zorder = 0.9, label = 'Guess')
plt.legend(loc = 'lower left')
plt.show()
上面最重要的部分是对leastsq函数的调用。确保您小心最初的猜测 - 如果猜测没有落在散点上,则模型可能无法正确收敛。在进行适当的猜测后...
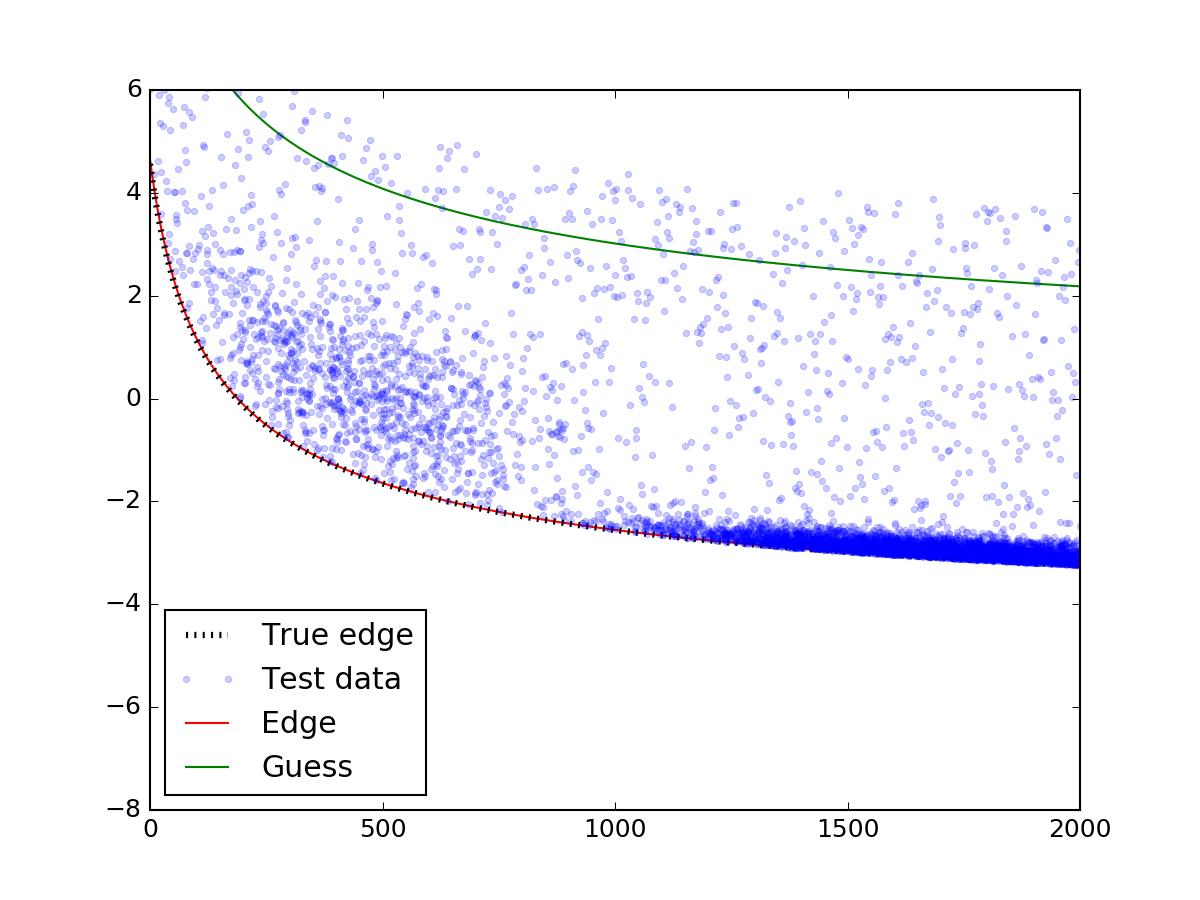
瞧!边缘与真实的完美匹配。
更多推荐
 已为社区贡献126473条内容
已为社区贡献126473条内容








所有评论(0)