如何给sns.clustermap一个预先计算好的距离矩阵?
·
回答问题
通常当我做树状图和热图时,我使用距离矩阵并做一堆SciPy的东西。我想尝试Seaborn但Seaborn想要我的数据以矩形形式(行 u003d 样本,列 u003d 属性,而不是距离矩阵)?
我本质上想使用seaborn作为后端来计算我的树状图并将其附加到我的热图上。这可能吗?如果没有,这是否可以成为未来的功能。
也许我可以调整一些参数,以便它可以采用距离矩阵而不是矩形矩阵?
这是用法:
seaborn.clustermap¶
seaborn.clustermap(data, pivot_kws=None, method='average', metric='euclidean',
z_score=None, standard_scale=None, figsize=None, cbar_kws=None, row_cluster=True,
col_cluster=True, row_linkage=None, col_linkage=None, row_colors=None,
col_colors=None, mask=None, **kwargs)
我的代码如下:
from sklearn.datasets import load_iris
iris = load_iris()
X, y = iris.data, iris.target
DF = pd.DataFrame(X, index = ["iris_%d" % (i) for i in range(X.shape[0])], columns = iris.feature_names)
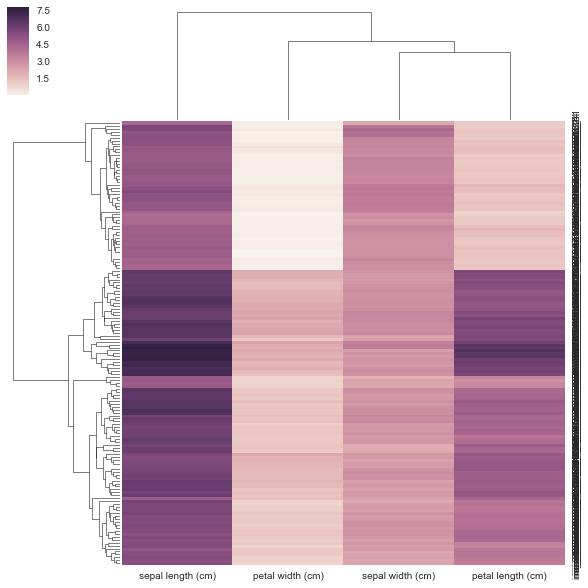
我不认为我的方法在下面是正确的,因为我给它一个预先计算的距离矩阵,而不是它要求的矩形数据矩阵。没有关于如何将相关/距离矩阵与clustermap一起使用的示例,但有https://stanford.edu/~mwaskom/software/seaborn/examples/network_correlations.html但排序不是聚集 w / 普通的sns.heatmap函数。
DF_corr = DF.T.corr()
DF_dism = 1 - DF_corr
sns.clustermap(DF_dism)
Answers
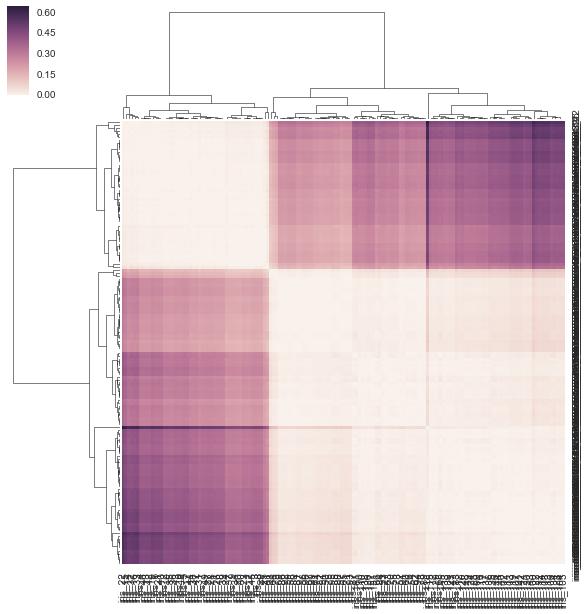
您可以将预先计算的距离矩阵作为链接传递给clustermap():
import pandas as pd, seaborn as sns
import scipy.spatial as sp, scipy.cluster.hierarchy as hc
from sklearn.datasets import load_iris
sns.set(font="monospace")
iris = load_iris()
X, y = iris.data, iris.target
DF = pd.DataFrame(X, index = ["iris_%d" % (i) for i in range(X.shape[0])], columns = iris.feature_names)
DF_corr = DF.T.corr()
DF_dism = 1 - DF_corr # distance matrix
linkage = hc.linkage(sp.distance.squareform(DF_dism), method='average')
sns.clustermap(DF_dism, row_linkage=linkage, col_linkage=linkage)
对于clustermap(distance_matrix)(即,没有传递链接),链接是根据距离矩阵中的行和列的成对距离在内部计算的(完整详细信息请参见下面的注释),而不是直接使用距离矩阵的元素(正确的解决方案) )。结果,输出与问题中的输出有些不同:
注意:如果没有row_linkage传递给clustermap(),则行链接是通过将每一行视为一个“点”(观察)并计算点之间的成对距离在内部确定的。所以行树状图反映了行的相似性。与col_linkage类似,其中每一列都被视为一个点。这个解释应该被添加到文档中。这里修改了文档的第一个示例以明确内部链接计算:
import seaborn as sns; sns.set()
import scipy.spatial as sp, scipy.cluster.hierarchy as hc
flights = sns.load_dataset("flights")
flights = flights.pivot("month", "year", "passengers")
row_linkage, col_linkage = (hc.linkage(sp.distance.pdist(x), method='average')
for x in (flights.values, flights.values.T))
g = sns.clustermap(flights, row_linkage=row_linkage, col_linkage=col_linkage)
# note: this produces the same plot as "sns.clustermap(flights)", where
# clustermap() calculates the row and column linkages internally
更多推荐
 已为社区贡献126445条内容
已为社区贡献126445条内容







所有评论(0)