机器学习降维之PCA(python代码+数据)
加深PCA算法研究
机器学习之PCA
首先分为四个部分来陈述:
- 产生应用背景
- 设计思路
- 案例实践
- 总结
- 附录
一.产生应用背景
主成分分析(Principal Components Analysis),简称PCA,是一种数据降维技术,用于数据预处理。一般我们获取的原始数据维度都很高,比如1000个特征,在这1000个特征中可能包含了很多无用的信息或者噪声,真正有用的特征才100个,那么我们可以运用PCA算法将1000个特征降到100个特征。这样不仅可以去除无用的噪声,还能减少很大的计算量。
在PCA中,数据从原来的坐标系转化到新的坐标系中。当然这里新的坐标系也不是随便设定的,而是应该根据数据本身的特征来设计。通常第一个新坐标轴选择的是原始数据方差最大的方向,第二个坐标轴是与第一个坐标轴正交且具有最大方差的方向。这句话的意思就是,第二个选取的方向应该和第一个方向具有很弱的相关性。
总结
- 降维可以缓解维度灾难问题
- 降维可以在压缩数据的同时让信息损失最小化
- 理解几百个维度的数据结构很困难,两三个维度的数据通过可视化更容易理解
二.设计思路
PCA主成分分析算法,是一种线性降维,将高维坐标系映射到低维坐标系中。
如何选择低维坐标系呢?就是求协方差的特征值和特征向量过协方差矩阵的特征值和特征向量,特征向量代表坐标系,特征值代表映射到新坐标的长度
然后做数据转换。
但是有没有觉得很神奇,为什么求协方差的较大特征向量就是最理想的k维向量?
来个实验项目来看看吧!!!
三.案例实践
知识储备
-
方差:描述波动情况
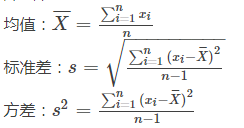
-
协方差:x,y的相关性
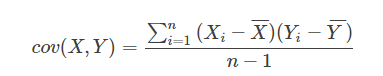
-. 协方差矩阵:注意这是三维的,我们下面工程中用的二维,没有z值
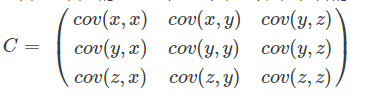
1. 原始数据处理:这里选取二维降一维进行展示,其中x,y是两个维度
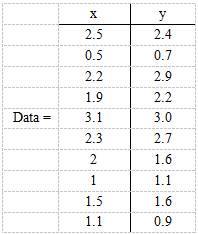
行代表了样例,列代表特征(TF-IDF)分别求x和y的平均值,然后对于所有的样例,都减去对应的均值。这里x的均值是1.81,y的均值是1.91,那么一个样例减去均值后即为(0.69,0.49)
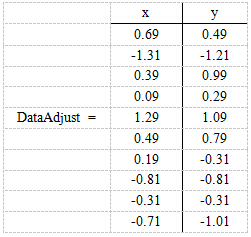
留一个思考1:为什么我们要减去均值,作用是什么?
2. 求协方差矩阵
我们上面协方差矩阵是二维的,所以是二行二列
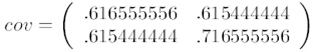
3.求协方差的特征值和特征向量
这边用到线性代数知识,不会的同学可以自行学习
下图第一个是特征值,第二个是特征向量(以下数字各位都是0省略不写了)
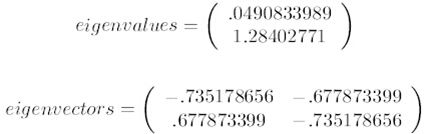
特征值0.0490833989对应特征向量为第一条向量,这里的特征向量都归一化为单位向量。
4.选取最大k个特征值对应的特征向量(其中k是小于维度总数的一个值):
将特征值按照从大到小的顺序排序,选择其中最大的k个,然后将其对应的k个特征向量分别作为列向量组成特征向量矩阵。这里特征值只有两个,我们选择其中最大的那个,这里是1.28402771,对应的特征向量是(-0.677873399, -0.735178656)T。我们是从二维降到一维,所以k取一,并且是最大的。
留两个思考题:
- 思考二: 为什么要选择最大的k?依据是什么?
- 思考三: k在实际运用中选取多少合适?
5.得到新的矩阵
将样本点投影到选取的特征向量上。假设样例数为m,特征数为n,减去均值后的样本矩阵为DataAdjust(mn),
协方差矩阵是nn,
选取的k个特征向量组成的矩阵为EigenVectors(nk)。
那么投影后的数据FinalData为FinalData(101)
= DataAdjust(10*2矩阵) x 特征向量(-0.677873399, -0.735178656)T
得到的结果是下图
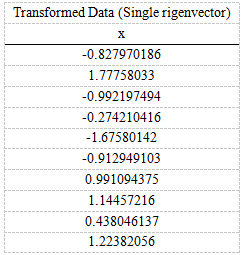
降维完成,同时又达到保真性,我们一会给出python代码进行验证,不使用PCA会怎样
我们回答思考一:为什么数据要减去均值
简单来说把数据中心化,减少过度拟合的可能性。如果有喜欢探索为什么的同学可以参看附录一
我们回答思考二:为什么选取k的特征值越大越好?
最大方差理论: 前辈结论:最好的k维特征是将n维样本点转换为k维后,每一维上的样本方差都很大。
why?
因为投影后的样本点之间方差最大(也可以说是投影的绝对值之和最大)
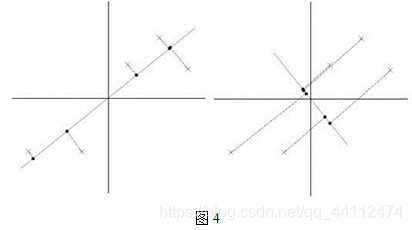
把二维降为一维如果方差不大那么就会在主方向轴投影点会重叠,损失信息很大,并且丢失的向量是很重要的。看下图4,5吧!(图4,5数据过原点已经数据中心化)图四的左半部分是最优的
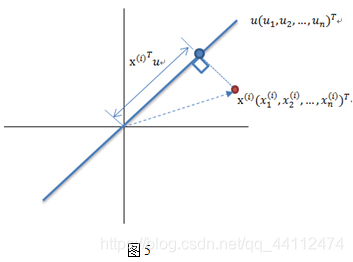
在u>x,u为主向量时。我们可以看到点在u上的投影和u和u的正交向量有关。显然图4左边的投影面比右边的大并且投影点之间的方差大。图五是图四的单个点细节图。
我们总结一下:
特征值在线性代数里面我们求过无数次了,对一个n*n的对称矩阵进行分解,我们可以求出它的特征值和特征向量,就会产生n个n维的正交基,每个正交基会对应一个特征值。然后把矩阵投影到这N个基上,此时特征值的模就表示矩阵在该基的投影长度。
特征值越大,说明矩阵在对应的特征向量上的方差越大,样本点越离散,越容易区分,信息量也就越多。因此,特征值最大的对应的特征向量方向上所包含的信息量就越多,如果某几个特征值很小,那么就说明在该方向的信息量非常少,我们就可以删除小特征值对应方向的数据,只保留大特征值方向对应的数据,这样做以后数据量减小,但有用的信息量都保留下来了。PCA就是这个原理。
我们回答思考三:在n维中选多少个k最合适?
当然,k(特征向量)取得越多越好,k越多熵越大,样本不确定性越大,越接近真实数据。如果k越大就达不到我们说的降维效果了。所以这是个经验之谈
有些研究工作表明,所选的主轴总长度占所有主轴长度之和的大约85% 即可,其实,这只是一个大体的说法,具体选多少个,要看实际情况而定。如下图公式。注:n为样本数,k为我们选的维数
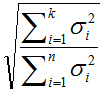
四.总结
步骤回顾
- 去除平均值
- 计算协方差矩阵
- 计算协方差矩阵的特征值和特征向量
- 特征值从大到小排序
- 保留最上面k个特征向量
- 将数据转换到k个向量构件的新空间中
- n维矩阵*k维特征向量=k维矩阵
降维完成!!!
附录一:
为了说明什么是数据的主成分,先从数据降维说起。数据降维是怎么回事儿?假设三维空间中有一系列点,这些点分布在一个过原点的斜面上,如果你用自然坐标系x,y,z这三个轴来表示这组数据的话,需要使用三个维度,而事实上,这些点的分布仅仅是在一个二维的平面上,问题出在哪里?把x,y,z坐标系旋转一下,使数据所在平面与x,y平面重合如果把旋转后的坐标系记为x’,y’,z’,那么这组数据的表示只用x’和y’两个维度表示即可!当然了,如果想恢复原来的表示方式,那就得把这两个坐标之间的变换矩阵存下来。这样就能把数据维度降下来了!看下图进行理解这段话
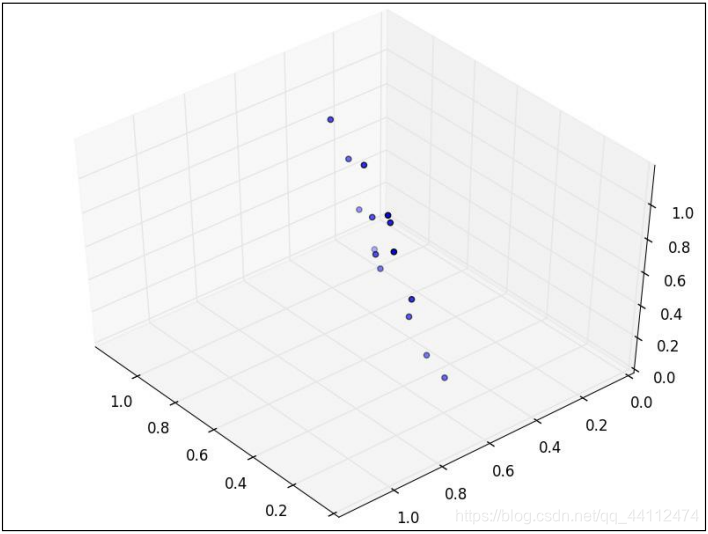
但是,我们要看到这个过程的本质,如果把这些数据按行或者按列排成一个矩阵,那么这个矩阵的秩就是2!这些数据之间是有相关性(有非零解)的,这些数据构成的过原点的向量的最大线性无关组包含2个向量,这就需要平面过原点的!这就是数据中心化的缘故!将坐标原点平移到数据中心,这样原本不相关的数据在这个新坐标系中就有相关性了!增加基向量的正交性。有趣的是,三点一定共面,也就是说三维空间中任意三点中心化后都是线性相关的,一般来讲n维空间中的n个点一定能在一个n-1维子空间中分析!n维欧氏空间中余维度等于一的线性子空间,也就是必须是(n-1)维度。这是平面中的直线、空间中的平面之推广。
附录二:python代码和数据
说明:python代码是为了展示四维数据利用PCA降维和不降维在二维空间的对比
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt #2D绘图库
#计算均值,要求输入数据为numpy的矩阵格式,行表示样本数,列表示特征
def meanX(dataX):
return np.mean(dataX,axis=0)#axis=0表示按照列来求均值,如果输入list,则axis=1
def pca(XMat, k):
average = meanX(XMat)
m, n = np.shape(XMat)
data_adjust = []
avgs = np.tile(average, (m, 1))
data_adjust = XMat - avgs
covX = np.cov(data_adjust.T) #计算协方差矩阵
featValue, featVec= np.linalg.eig(covX) #求解协方差矩阵的特征值和特征向量
index = np.argsort(-featValue) #按照featValue进行从大到小排序
finalData = []
if k > n:
print ("k must lower than feature number")
return
else:
#注意特征向量时列向量,而numpy的二维矩阵(数组)a[m][n]中,a[1]表示第1行值
selectVec = np.matrix(featVec.T[index[:k]]) #所以这里需要进行转置
finalData = data_adjust * selectVec.T
reconData = (finalData * selectVec) + average
return finalData, reconData
#输入文件的每行数据都以\t隔开
def loaddata(datafile):
return np.array(pd.read_csv(datafile,sep=" ",header=-1)).astype(np.float)
def plotBestFit(data1, data2):
dataArr1 = np.array(data1)
dataArr2 = np.array(data2)
m = np.shape(dataArr1)[0]
axis_x1 = []
axis_y1 = []
axis_x2 = []
axis_y2 = []
for i in range(m):
axis_x1.append(dataArr1[i,0])
axis_y1.append(dataArr1[i,1])
axis_x2.append(dataArr2[i,0])
axis_y2.append(dataArr2[i,1])
fig = plt.figure()
ax = fig.add_subplot(111)
ax.scatter(axis_x1, axis_y1, s=50, c='red', marker='s')
ax.scatter(axis_x2, axis_y2, s=50, c='blue')
plt.xlabel('x1'); plt.ylabel('x2');
plt.savefig("outfile.png")
plt.show()
#根据数据集data.txt
def main():
datafile = "data.txt"
XMat = loaddata(datafile)
k = 2
return pca(XMat, k)
if __name__ == "__main__":
finalData, reconMat = main()
plotBestFit(finalData, reconMat)
数据:data.txt文件,注意:使用的是相对路径,把数据放到你的.py代码同一级目录下,并列关系
5.1 3.5 1.4 0.2
4.9 3.0 1.4 0.2
4.7 3.2 1.3 0.2
4.6 3.1 1.5 0.2
5.0 3.6 1.4 0.2
5.4 3.9 1.7 0.4
4.6 3.4 1.4 0.3
5.0 3.4 1.5 0.2
4.4 2.9 1.4 0.2
4.9 3.1 1.5 0.1
5.4 3.7 1.5 0.2
4.8 3.4 1.6 0.2
4.8 3.0 1.4 0.1
4.3 3.0 1.1 0.1
5.8 4.0 1.2 0.2
5.7 4.4 1.5 0.4
5.4 3.9 1.3 0.4
5.1 3.5 1.4 0.3
5.7 3.8 1.7 0.3
5.1 3.8 1.5 0.3
5.4 3.4 1.7 0.2
5.1 3.7 1.5 0.4
4.6 3.6 1.0 0.2
5.1 3.3 1.7 0.5
4.8 3.4 1.9 0.2
5.0 3.0 1.6 0.2
5.0 3.4 1.6 0.4
5.2 3.5 1.5 0.2
5.2 3.4 1.4 0.2
4.7 3.2 1.6 0.2
4.8 3.1 1.6 0.2
结束!!!
参考博客1:https://www.jianshu.com/p/f9c6b36395f6
参考博客代码:https://blog.csdn.net/Dream_angel_Z/article/details/50760130
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)