R语言数据处理学习记录--用自己的数据完成mental test(使用ggcor包)
ggcor的目标是提供一组可以用来快速可视化相关矩阵的函数,大家可以在GitHub上很方便的获取关于这个包的全部信息,它可以实现图形的绘制。这里给出的是一个备用源,国内的gitee上也有很多转的,如果有小伙伴无法访问GitHub可以使用gitee:;GitHub上也有给出,除了这样的安装方式以外还可以本地安装,因为有时GitHub连接不上,本地安装可以避免这个问题。
R语言数据处理学习记录–用自己的数据完成mental test(使用ggcor包)
注:本文仅作为自己的学习记录以备复习查阅
关于科学上网:https://www.zhihu.com/question/498939985
一 ggcor包的简介及其安装
ggcor的目标是提供一组可以用来快速可视化相关矩阵的函数,大家可以在GitHub上很方便的获取关于这个包的全部信息,它可以实现Correlation plot、Mantel test plot、Circular heatmap、General heatmap图形的绘制。
GitHub地址:https://github.com/mj163163/ggcor-1
这里给出的是一个备用源,国内的gitee上也有很多转的,如果有小伙伴无法访问GitHub可以使用gitee:
gitee地址:https://gitee.com/future-doctor/ggcor?_from=gitee_search
;
ggcor包的安装:
# install.packages("devtools")
devtools::install_github("houyunhuang/ggcor")
GitHub上也有给出,除了这样的安装方式以外还可以本地安装,因为有时GitHub连接不上,本地安装可以避免这个问题。
其他安装方法:
1 安装devtools或remotes包(Rstudio中),再使用devtools或者remotes安装GitHub上的包
install.packages("devtools")
install.packages("remotes")
devtools::install_github('houyunhuang/ggcor')
2 本地安装
我们在GitHub或者gitee上将zip包下载下来(xxx-master.zip),使用命令(路径啥的记得自己修改一下):
remotes::install_local("D:/ggcor-master.zip",upgrade = F,dependencies = T)
一般来说,本地安装都能很好的解决。
二 ggcor实现mental test的代码
首先加载一些有用的依赖包:
#加载包
library(vegan)
library(dplyr)
library(ggcor)
library(ggplot2)#这个包是用于图片导出,更清晰
GitHub上给出的数据是读取它本身给的例子:
data("varechem", package = "vegan")
data("varespec", package = "vegan")
这里我们展示不用例子数据的方法(事先准备好自己的数据):
我再解释一些关于这个数据,最好是按照例子中的数据格式整理好然后转成csv文件再导入:
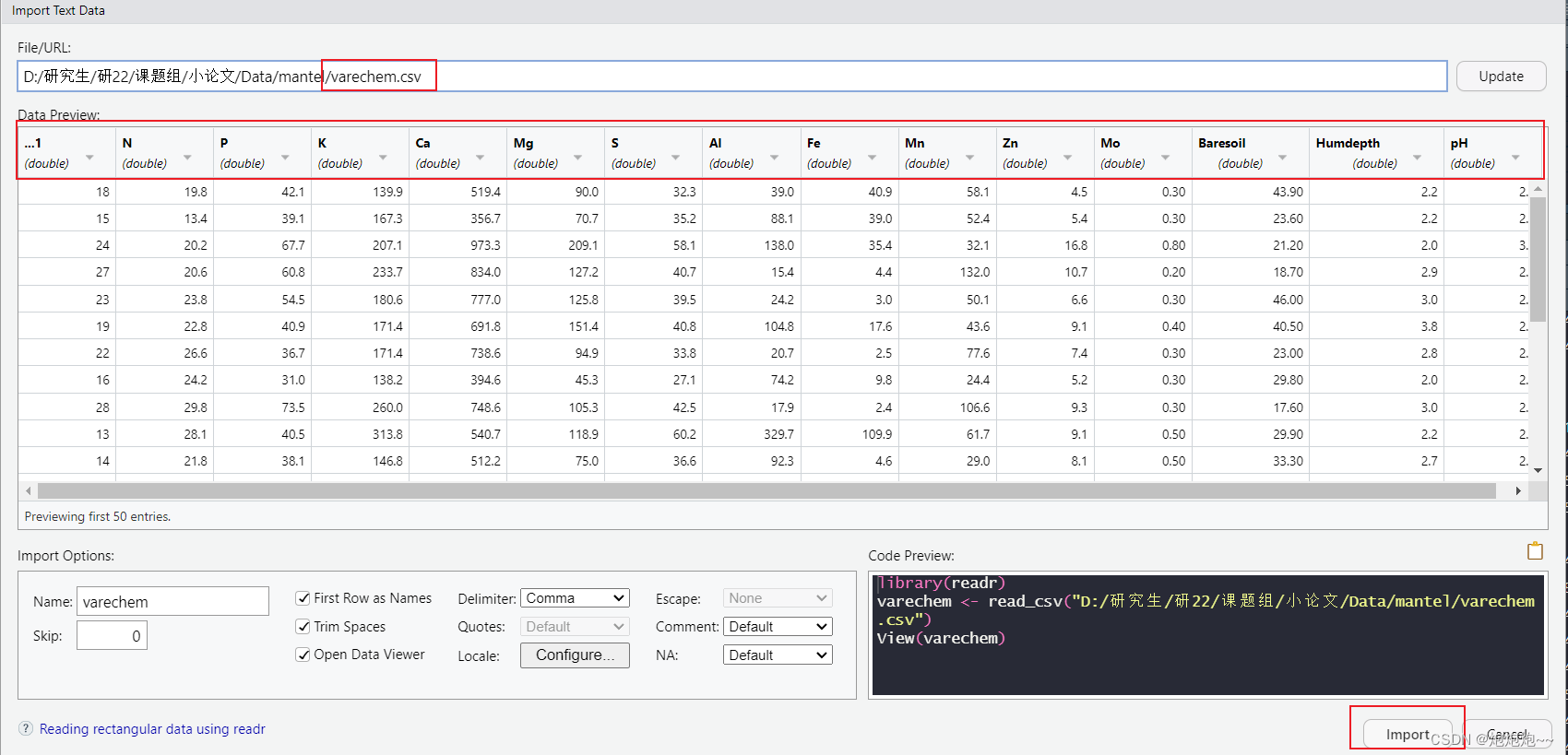
数据导入之后我们进行下一步,计算出mental test 的 R值和P值,这里的varespec和varechem是例子的数据集名,各位在用的时候记得更改为自己的:
mantel <- mantel_test(varespec, varechem, #这里要改成你自己的dataset
spec.select = list(Spec01 = 1:7,
Spec02 = 8:18,
Spec03 = 19:37,
Spec04 = 38:44)) %>% #定义有几种分组
mutate(rd = cut(r, breaks = c(-Inf, 0.2, 0.4, Inf),
labels = c("< 0.2", "0.2 - 0.4", ">= 0.4")),#定义Mantel的R值范围
pd = cut(p.value, breaks = c(-Inf, 0.01, 0.05, Inf),
labels = c("< 0.01", "0.01 - 0.05", ">= 0.05")))#定义Mantel检验的p值范围
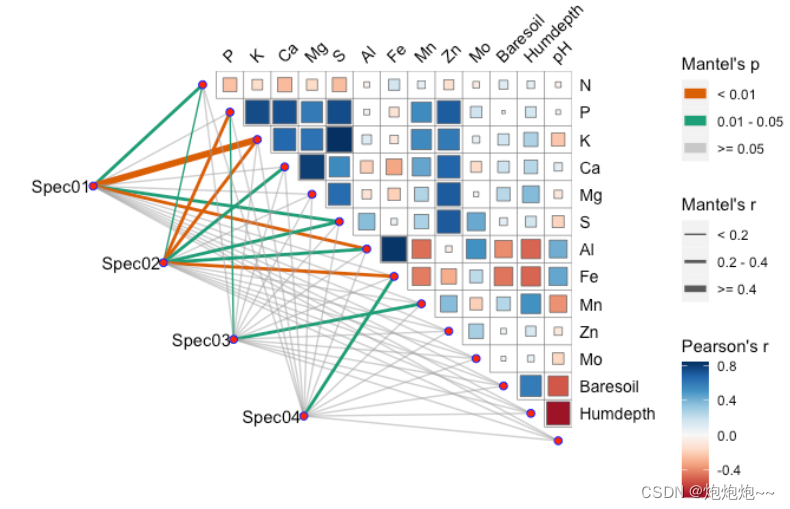
完成之后会在Rstudio的右侧出现运算的结果(这里又是我自己的数据了):

最后一步就是让计算结果可视化起来,这里放作者给的代码,其中颜色线条粗细和一些字都是可以更改和调整的(数据集名称记得调换):
quickcor(varechem, type = "upper") +
geom_square() +
anno_link(aes(colour = pd, size = rd), data = mantel) +
scale_size_manual(values = c(0.5, 1, 2)) +
scale_colour_manual(values = c("#D95F02", "#1B9E77", "#A2A2A288")) +
guides(size = guide_legend(title = "Mantel's r",
override.aes = list(colour = "grey35"),
order = 2),
colour = guide_legend(title = "Mantel's p",
override.aes = list(size = 3),
order = 1),
fill = guide_colorbar(title = "Pearson's r", order = 3))

由于我们要将生成的图片导出更清晰的格式,将可视化结果赋值到photo(当然这里可以取任何名字)
photot <- quickcor(varechem, type = "upper") +
geom_square() +
anno_link(aes(colour = pd, size = rd), data = mantel) +
scale_size_manual(values = c(0.5, 1, 2)) +
scale_colour_manual(values = c("#D95F02", "#1B9E77", "#A2A2A288")) +
guides(size = guide_legend(title = "Mantel's r",
override.aes = list(colour = "grey35"),
order = 2),
colour = guide_legend(title = "Mantel's p",
override.aes = list(size = 3),
order = 1),
fill = guide_colorbar(title = "Pearson's r", order = 3))
使用ggplot2导出生成的图片:
ggsave("D:/mantel.tiff",photo,width=8,height=6)
整个过程其实都非常简单,代码作者也给的很清晰,大家在使用时注意修改,多试一试,后续再发一个关于linkET包的使用教程,有任何问题和困难欢迎私信交流。
以上!!!!!
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容










所有评论(0)