GEO芯片数据下载和探针ID转换(保姆级教程)
R语言安装、GEO芯片数据下载、探针ID转换
GEO芯片数据下载和探针ID转换(保姆级教程)
本文章主要参考了:
①R 语言的安装(详细教程)
②GEO芯片数据下载和探针ID转换
一、问题描述
探针ID转换
我们需要的基因表达量信息在NCBI的GEO数据库中对应的编号为GSE95394,搜索后结果如下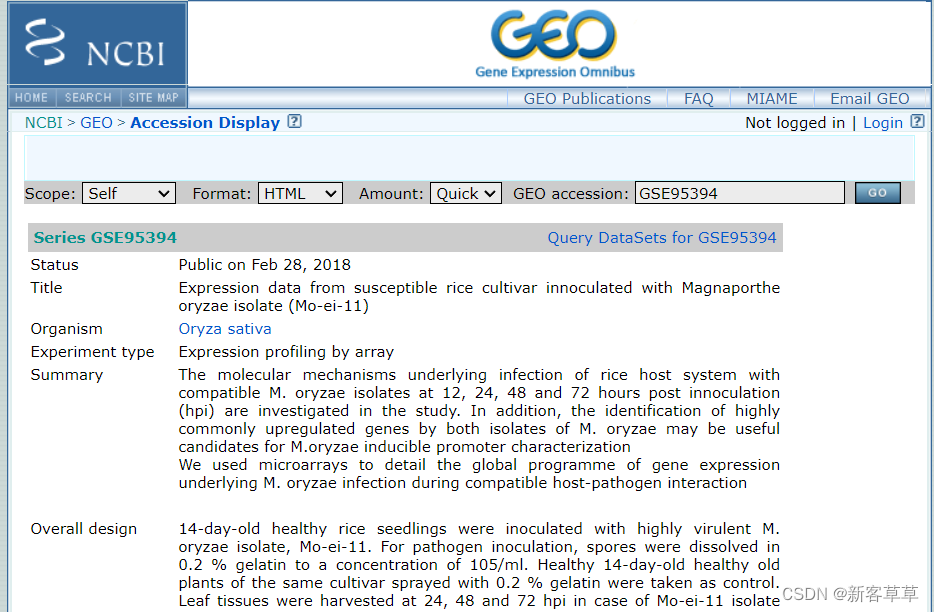
来到页面底部,这里的series Matrix File(s)是已经处理好所有样本对应各基因的表达量数据
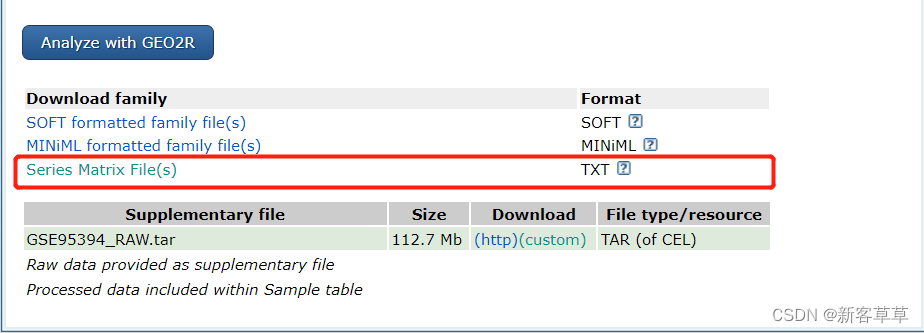
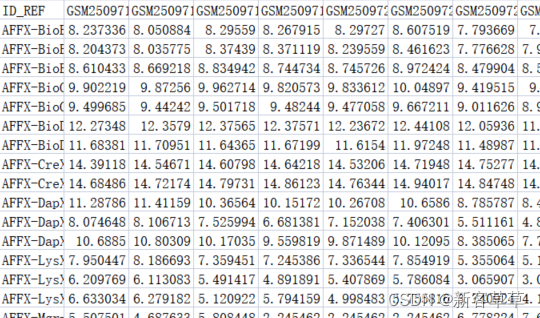
下载并打开后如下图,发现左边并不是基因的名字,而是探针ID号,我们需要将探针ID转换为基因名才能进一步处理
数据是否预处理过
此外还需要注意的是,选择其中某个样本点击进去
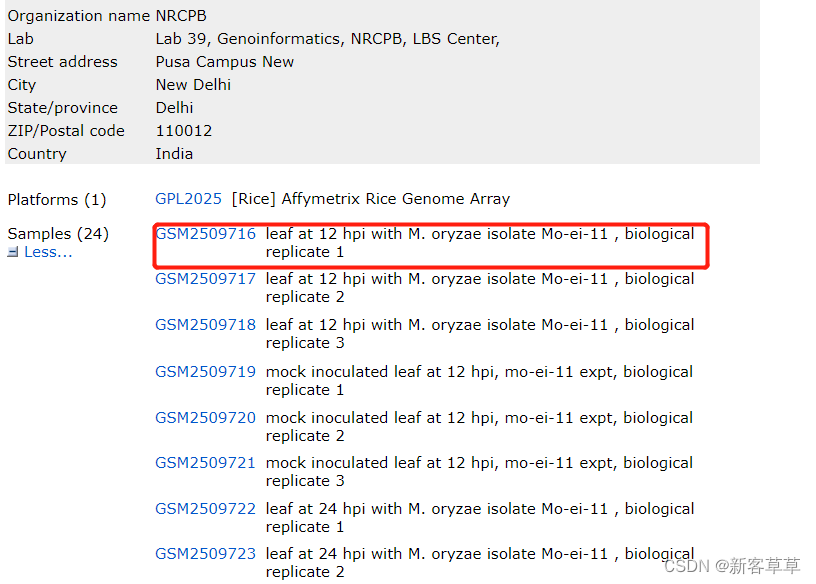
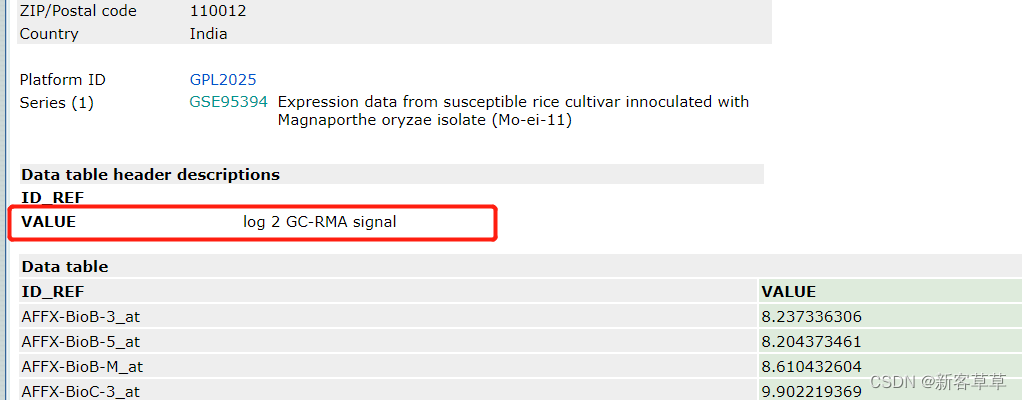
结果如下,这表明你下载的表达量是经过了log2(N+1)转化后的,如果没做特殊说明的话,这里的N一般表示基因数量,即counts,那么你下载到的表达量就是log2(counts+1),这是需要我们注意的
二、Rstudio的安装(建议阅读,避免后续转换时出错)
安装包的下载
注意:由于我们需要使用WGCNA等包,所以对R以及RStudio的版本是有一定要求的(要求>4.1.3)
这里我已经将合适的版本上传至百度网盘,注意:这三个安装文件都需要下载,缺一不可
链接:https://pan.baidu.com/s/1pbgbCVQf69sEk7_tK8SGSw
提取码:4aeh
安装步骤
①可以在D盘中新建一个R文件夹,将这三个文件都放在R文件夹里面,然后安装路径也都在这个R文件夹里面。(为什么安装包要和最终的安装路径放一块?因为不这样的话,RStudio运行时可能检测不到R-4.2.2安装文件夹里面etc文件夹中的Rprofile.site,这个文件的作用我稍后会解释)
②按照R-4.2.2-win、rtools、RStudio顺序依次安装(顺序不能乱),安装时都选择默认选项即可。
③因为有些R中的包依赖于java所以还需要进行java的环境配置:java 环境配置(详细教程)
④运行RStudio:在安装的RStudio>bin文件夹里面,有个rstudio.exe,双击打开即可
⑤镜像源的设置:
使用记事本打开这个文件
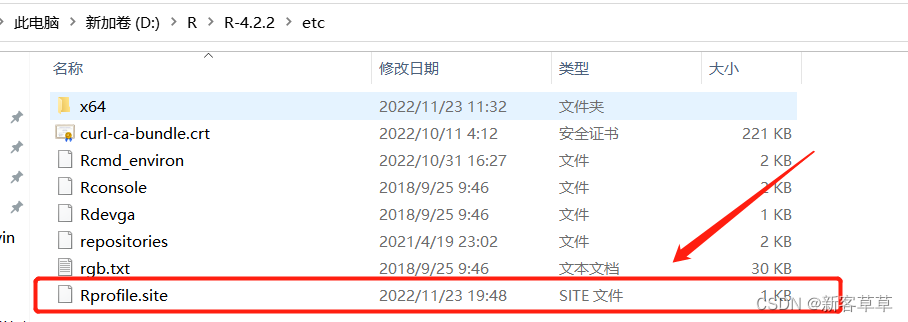
将下面这段代码复制并粘贴到最后
## 设置镜像
local({r <- getOption("repos")
r["CRAN"] <- "https://mirrors.tuna.tsinghua.edu.cn/CRAN/"
options(repos=r)}
)
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
## 设置下载方式
options("download.file.method"="libcurl")
options("url.method"="libcurl")
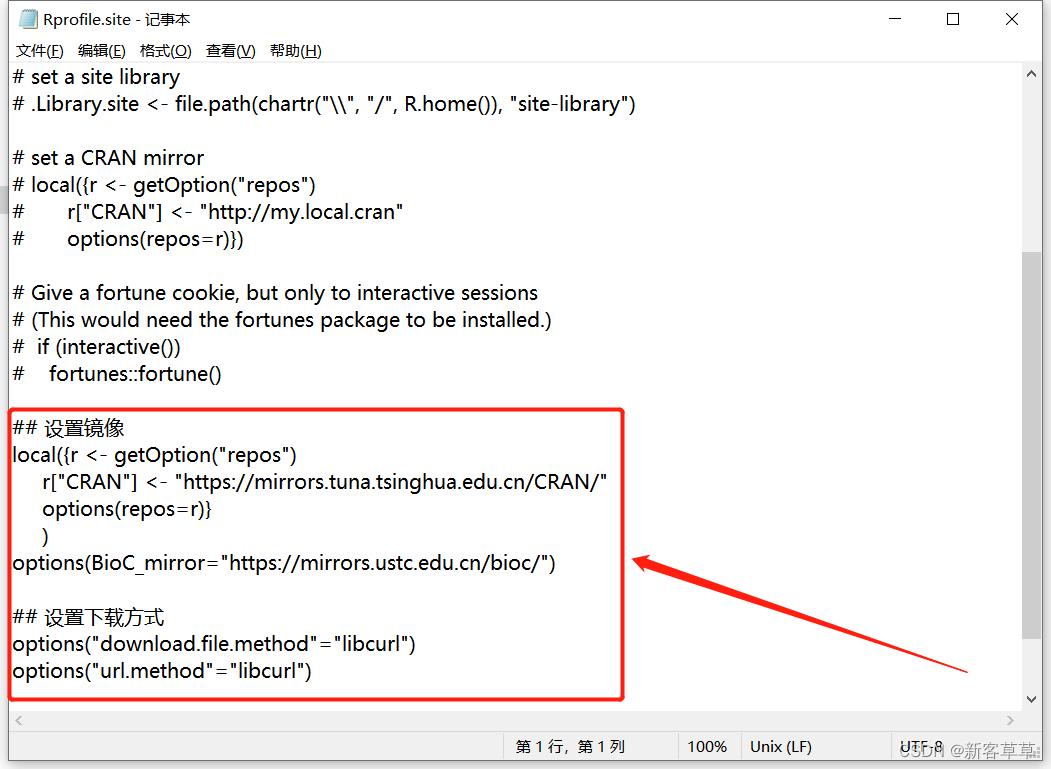
这是在RStudio中输入一下命令:
getOption("repos")
getOption("BioC_mirror")
返回以下结果则表示设置成功
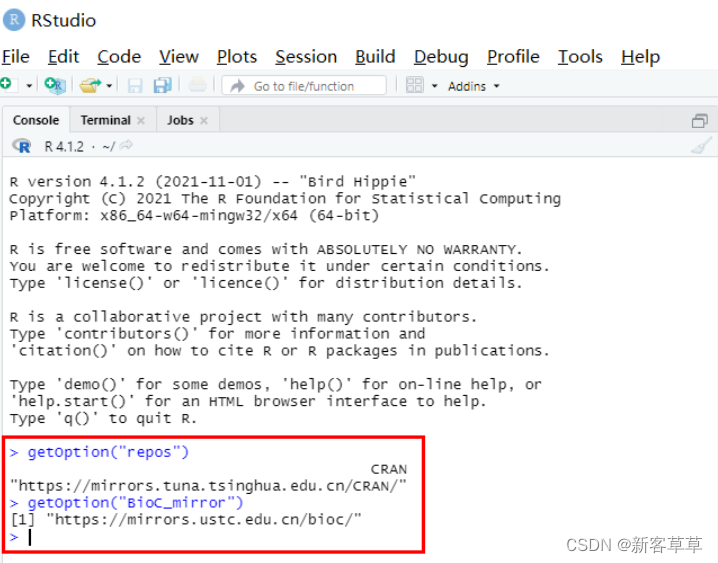
如果不设置好镜像源,可能后续再安装R包的时候,会出现找不到的情况,所以这是非常有必要的!
三、(正文)芯片数据下载和ID转换
注意:如果你是严格按照上面的步骤安装RStudio,那么这一步将不会出错,否则会遇到各种问题难以继续,如果已经错误地安装RStudio等软件最后发现很多代码无法实现,那么需要把已经安装的R、RStudio、rtools全部删除干净,然后再按照上面的步骤安装(来自作者踩坑无数的嘱咐)
相关设置和包的加载
进行相关设置
setwd("D:\\R\\data") # 将工作目录设置为D:\R\data,这里根据实际情况设置
options(stringAsFactors = F) # # 避免引入factor
Sys.setenv("VROOM_CONNECTION_SIZE"=131072*600) # 设置内存,避免不够用
有关R包的加载
library(GEOquery)
library(limma)
library(affy)
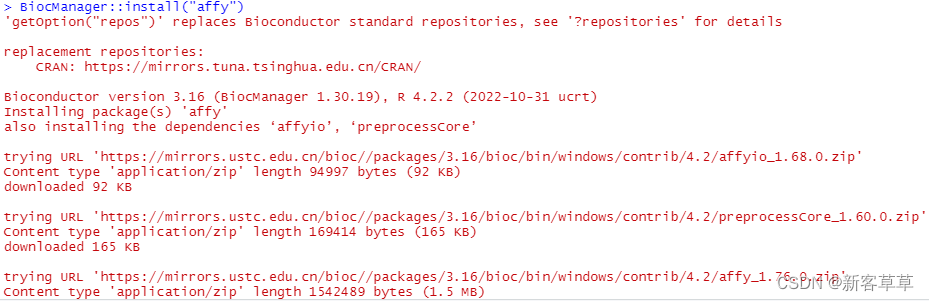
如果某个包你没有安装,那么可能会出现如下提示

安装下面方式安装即可(注意:使用install.packages(“affy”)有时会安装失败,所以还是建议使用下面的方式)
BiocManager::install("affy")
数据下载
这里下载本文章开头需要的数据:GSE95394,读者可以修改为自己需要下载的数据编号
gset <- getGEO('GSE95394', destdir=".",
+ AnnotGPL = T, ## 注释文件
+ getGPL = T) ## 平台文件
稍后文件会被下载到当前工作目录中,然后输入
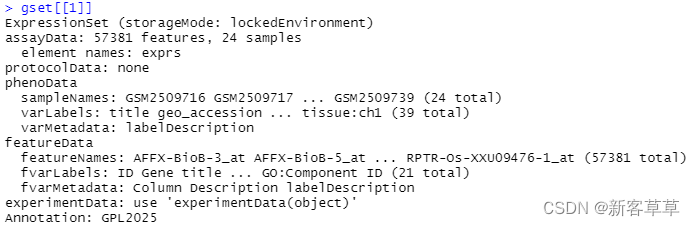
gset[[1]]
会显示该文件的相关信息(基因数量、平台、所用芯片等)
再输入以下命令
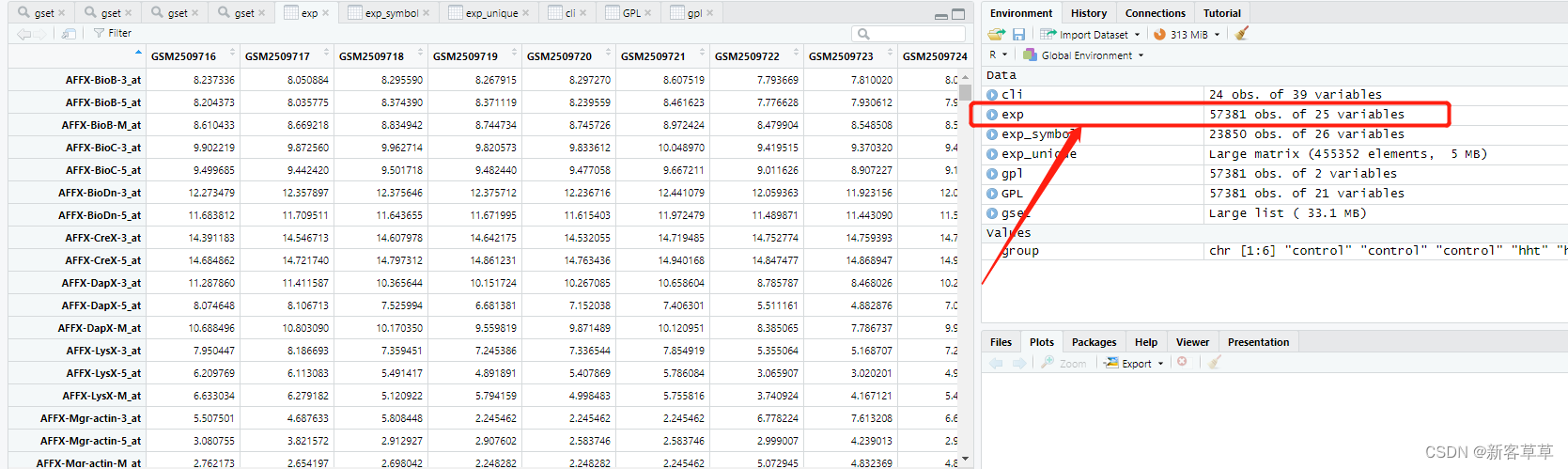
exp<-exprs(gset[[1]])
exp便代表了该表达矩阵,选中查看如图,这也是我们下载的数据直接打开的内容
cli<-pData(gse[[1]]) ## 获取临床信息
group<-c(rep("control",3),rep("hht",3)) ## 查看分组信息
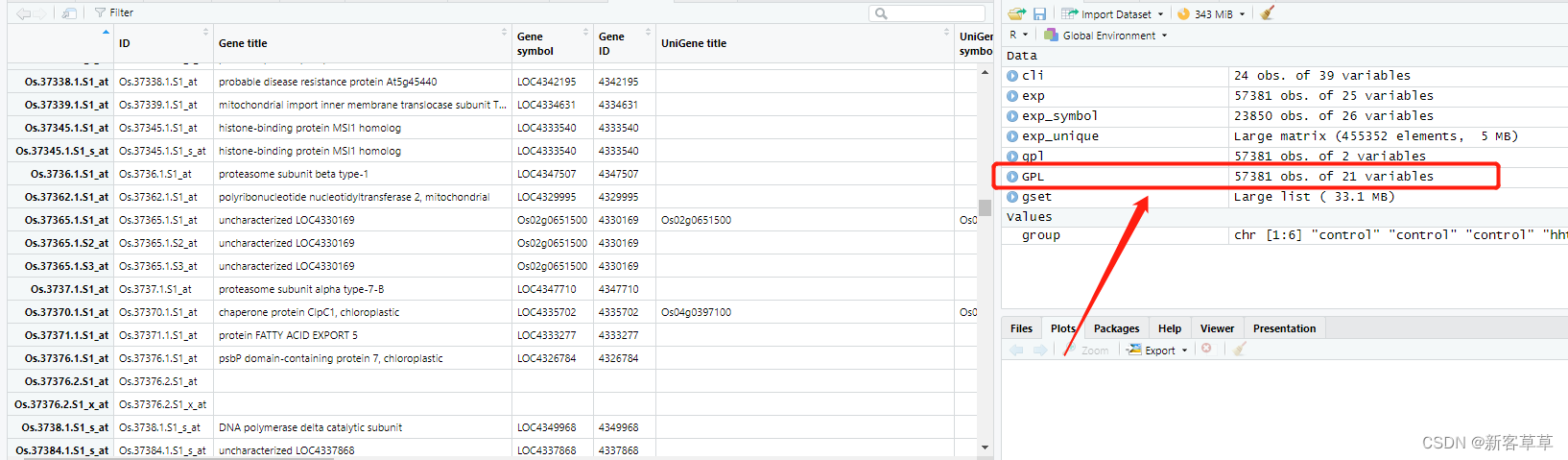
GPL<-fData(gset[[1]] ## 获取平台信息
查看GPL如下图,可以看到在该平台中芯片ID对应的各种数据库的ID号
注意可以左右拉动,这里我需要的是第一列(ID)和第三列(Gene symbol),那么可以将它提取出来,如果你需要别的列数据,那么修改下面的参数即可
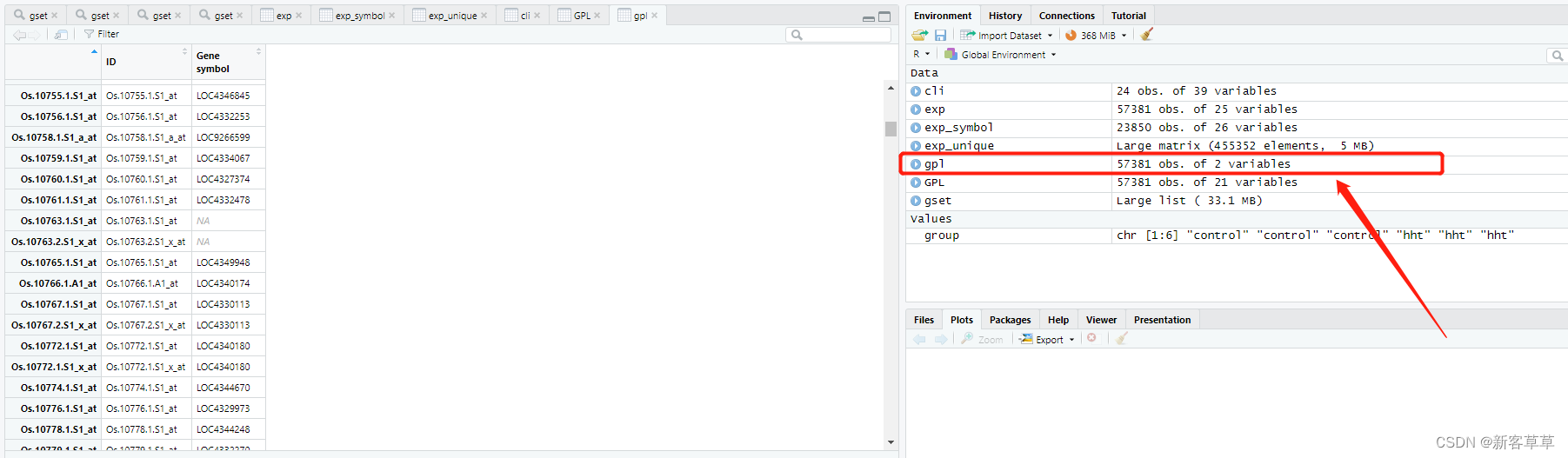
gpl<-GPL[,c(1,3)]
打开后如下图,我们会发现并不是所有芯片ID都有Gene symbol与其对应的,没有对应的Gene symbol我们直接忽略即可
此外,我们还会发现有的芯片ID会有两个Gene symbol与之对应并用“///”分隔开,如下图所示,那么我们取第一个即可
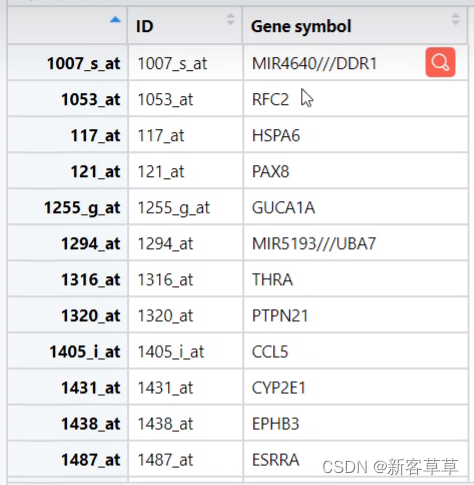
执行下面代码后,便通过只取第一个Gene symbol来达到去重效果,这时再打开gpl发现已经没有对应多个Gene symbol的情况了
gpl$`Gene symbol`<-data.frame(sapply(gpl$`Gene symbol`,function(x)unlist(strsplit(x,"///"))[1]),stringsAsFactors=F)[,1]
接下来对表达矩阵exp进行探针ID转化,首先将其转换为数据框的形式,否则一会会报错
exp<-as.data.frame(exp)
此时虽然我们能看到exp的探针ID号(最左侧),但这个ID号并不是真正在列中,所以我们还需要将探针ID放到数据框里面,执行下面命令
exp$ID<-rownames(exp) # 增加新的一列(最后一列),存放基因ID信息
接下来通过将gpl合并到exp中,从而在exp中将芯片ID号转化为Gene symbol
exp_symbol<-merge(exp,gpl,by="ID")
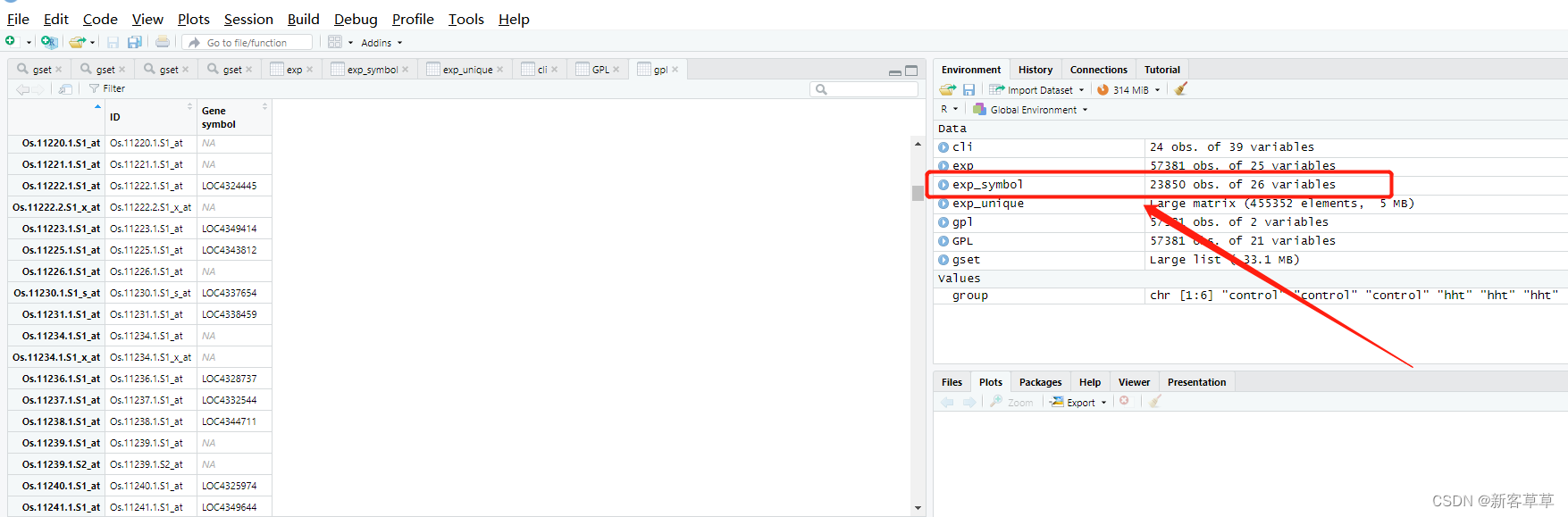
不过我们发现Gene symbol这里仍然有一些未能匹配上的(N/A),这时需要去除这些匹配不上的
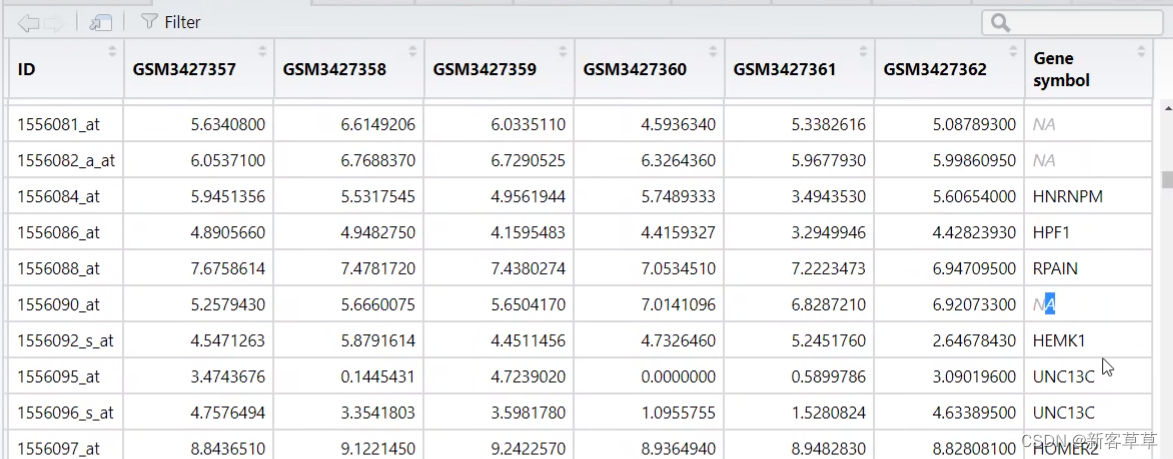
输入以下命令即可删除这些探针ID匹配不上的
exp_symbol<-na.omit(exp_symbol)
此时我们发现还有一些Gene symbol是重复的,需要我们去重
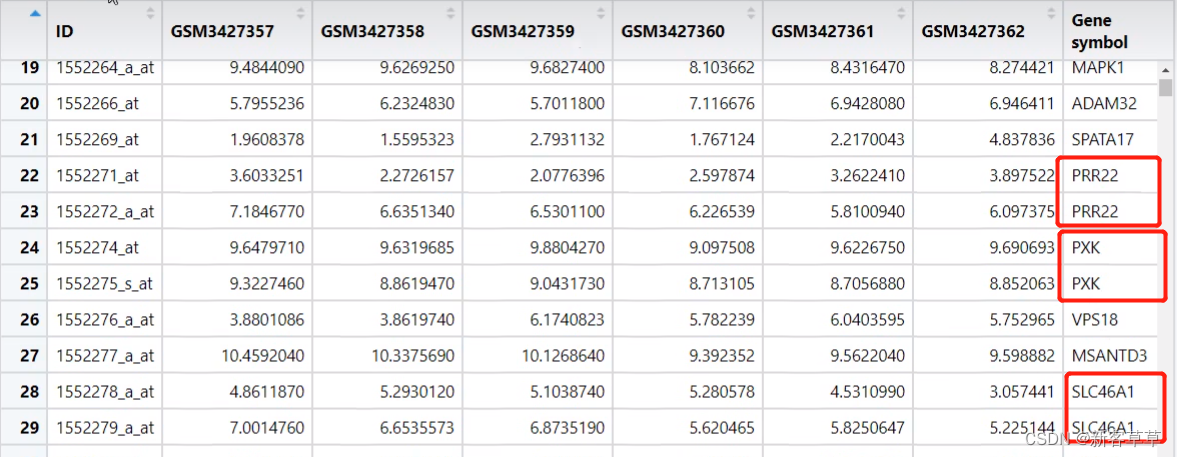
首先查看有多少重复的Gene symbol
table(duplicated(exp_symbol$`Gene symbol`))
结果如下图,说明有4877个重复的

输入以下命令进行去重操作同时将矩阵转换成标准的表达矩阵形式
exp_unique<-avereps(exp_symbol[,-c(1,ncol(exp_symbol))],ID=exp_symbol$`Gene symbol`)
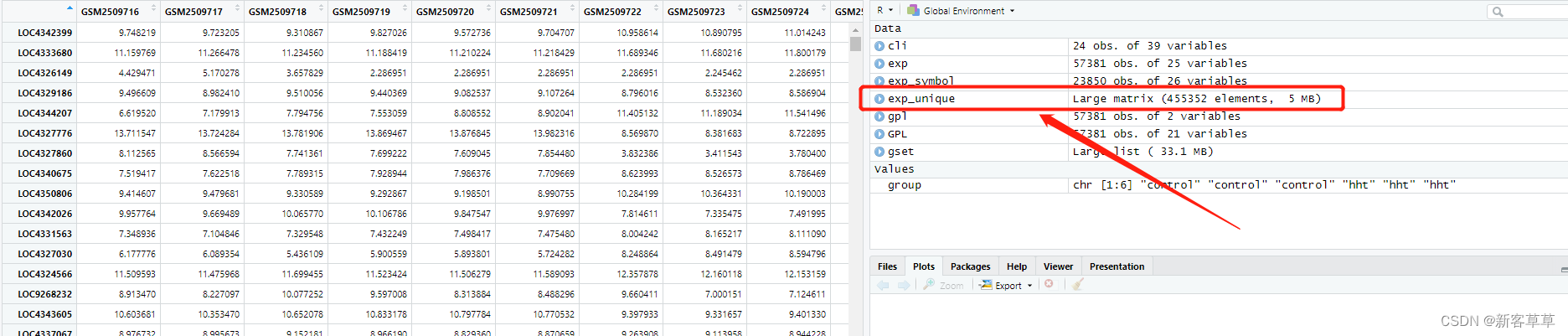
这个exp_unique就是我们需要的以Gene symbol为行标的表达矩阵,最后将它保存为csv格式
write.csv(exp_unique,"D:/desktop/aaa.csv")
大功告成~
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容










所有评论(0)