如何在 DAMODEL 云平台上高效训练 UNet 模型
UNet 是一种在医学图像分割领域非常成功的深度学习模型,因其独特的编码器-解码器结构和跳跃连接,使得它在小数据集上能够很好地捕获细节和边缘信息。尤其是在如眼底血管分割等任务中,UNet 展现了卓越的表现。本文将结合 DAMODEL 平台,深入介绍如何高效地使用 UNet 模型进行医学图像分割任务的训练与测试,并分享如何通过云计算资源加速深度学习模型的开发和优化。UNet 作为一种强大的图像分割模
文章目录
📑前言
UNet 是一种在医学图像分割领域非常成功的深度学习模型,因其独特的编码器-解码器结构和跳跃连接,使得它在小数据集上能够很好地捕获细节和边缘信息。尤其是在如眼底血管分割等任务中,UNet 展现了卓越的表现。本文将结合 DAMODEL 平台,深入介绍如何高效地使用 UNet 模型进行医学图像分割任务的训练与测试,并分享如何通过云计算资源加速深度学习模型的开发和优化。
一、UNet 的结构与工作原理
UNet 最早由 Olaf Ronneberger 等人在 2015 年提出,用于生物医学图像的语义分割任务。该网络采用了典型的对称结构,由编码器和解码器两个部分组成。编码器部分通过连续的卷积层和池化层,逐渐减少特征图的空间分辨率,提取高层次的特征。解码器部分则通过反卷积和跳跃连接,将编码器提取的特征逐步还原到与原图像相同的分辨率,从而生成精准的分割结果。
UNet 的关键在于跳跃连接(skip connections),它将编码器中不同阶段的特征直接传递给解码器的对应阶段。这种设计保证了在分割细节丰富的区域(例如边缘)时,网络不会丢失过多的空间信息。
二、数据准备
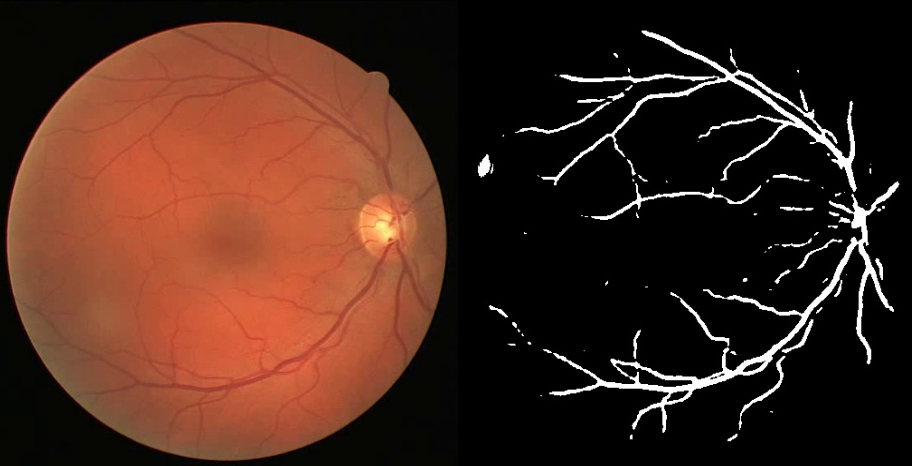
在 UNet 模型的应用中,数据准备是一个非常重要的环节。以眼底血管分割为例,输入模型的数据通常包括原始的眼底图像以及对应的标注掩码(即每个像素点是血管或背景的二值图像)。在使用 DAMODEL 平台时,用户可以通过以下步骤准备和上传数据:
2.1 数据预处理
首先,需要对图像进行标准化和尺寸调整,确保所有输入的图像大小一致。常用的预处理操作包括图像的灰度化、归一化以及随机裁剪、旋转等数据增强操作。
2.2 上传数据
在 DAMODEL 平台上,用户可以方便地将本地数据集上传至云实例。通过 DAMODEL 提供的文件管理工具,用户只需选择本地文件并点击上传,数据就会被安全地存储在云端,供训练过程调用。
2.3 数据格式转换
对于医学图像数据,通常会使用特定的格式(如 DICOM 或 TIFF)。用户需要确保数据格式符合 UNet 模型输入的要求,如果需要,可以通过 Python 进行格式转换。下述代码展示了如何将 DICOM 图像转换为 PNG 格式:
import pydicom
from PIL import Image
import numpy as np
def dicom_to_png(dicom_path, output_path):
dicom = pydicom.read_file(dicom_path)
img = dicom.pixel_array
img = (img - np.min(img)) / (np.max(img) - np.min(img)) * 255
img = Image.fromarray(img.astype('uint8'))
img.save(output_path)
三、在 DAMODEL 云平台上进行训练
DAMODEL 平台提供了强大的云计算资源,尤其是支持 GPU 的实例,极大地加速了深度学习模型的训练过程。以下是如何在 DAMODEL 上进行 UNet 训练的关键步骤:
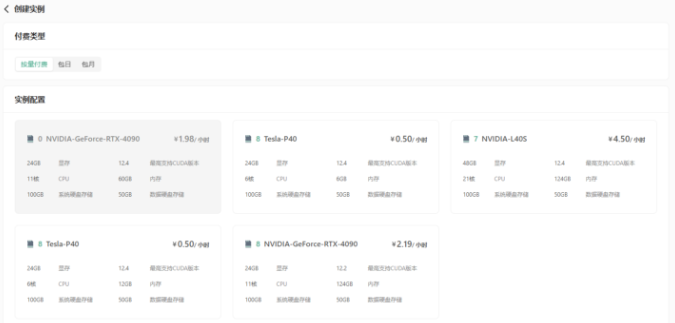
3.1 创建云实例
在 DAMODEL 平台上,用户可以轻松创建带有 GPU 支持的云实例。GPU 可以显著提升大规模数据集的训练速度,尤其是对于 UNet 这样复杂的深度学习模型。用户只需选择所需的计算资源配置,并启动实例。

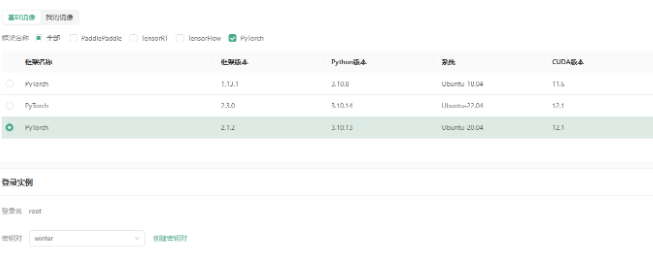
3.2 配置训练环境
一旦实例启动,接下来便是配置训练环境。DAMODEL 提供了预装了常用深度学习框架(如 PyTorch 和 TensorFlow)的环境,用户可以直接使用这些环境运行代码。如果需要特定的库,也可以通过 pip 或 conda 安装。
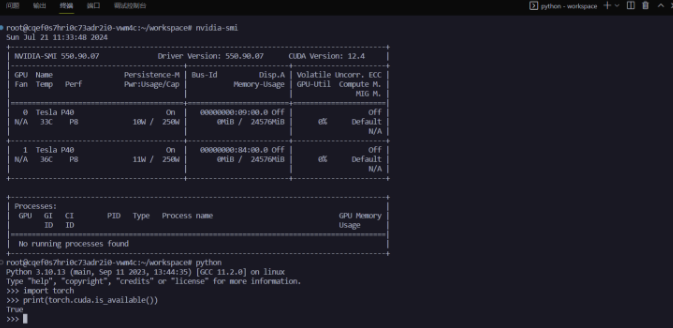
3.3 模型训练
在准备好数据和环境之后,便可以开始训练模型。
使用 PyTorch 实现 UNet 的训练流程如下:
import torch
import torch.nn as nn
import torch.optim as optim
from unet_model import UNet # UNet 网络结构定义在 unet_model.py 中
# 定义超参数
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model = UNet(n_channels=1, n_classes=2).to(device)
optimizer = optim.Adam(model.parameters(), lr=0.001)
criterion = nn.CrossEntropyLoss()
# 训练循环
for epoch in range(epochs):
model.train()
running_loss = 0.0
for inputs, labels in train_loader:
inputs, labels = inputs.to(device), labels.to(device)
# 前向传播
outputs = model(inputs)
loss = criterion(outputs, labels)
# 反向传播与优化
optimizer.zero_grad()
loss.backward()
optimizer.step()
running_loss += loss.item()
print(f'Epoch [{epoch+1}/{epochs}], Loss: {running_loss/len(train_loader)}')
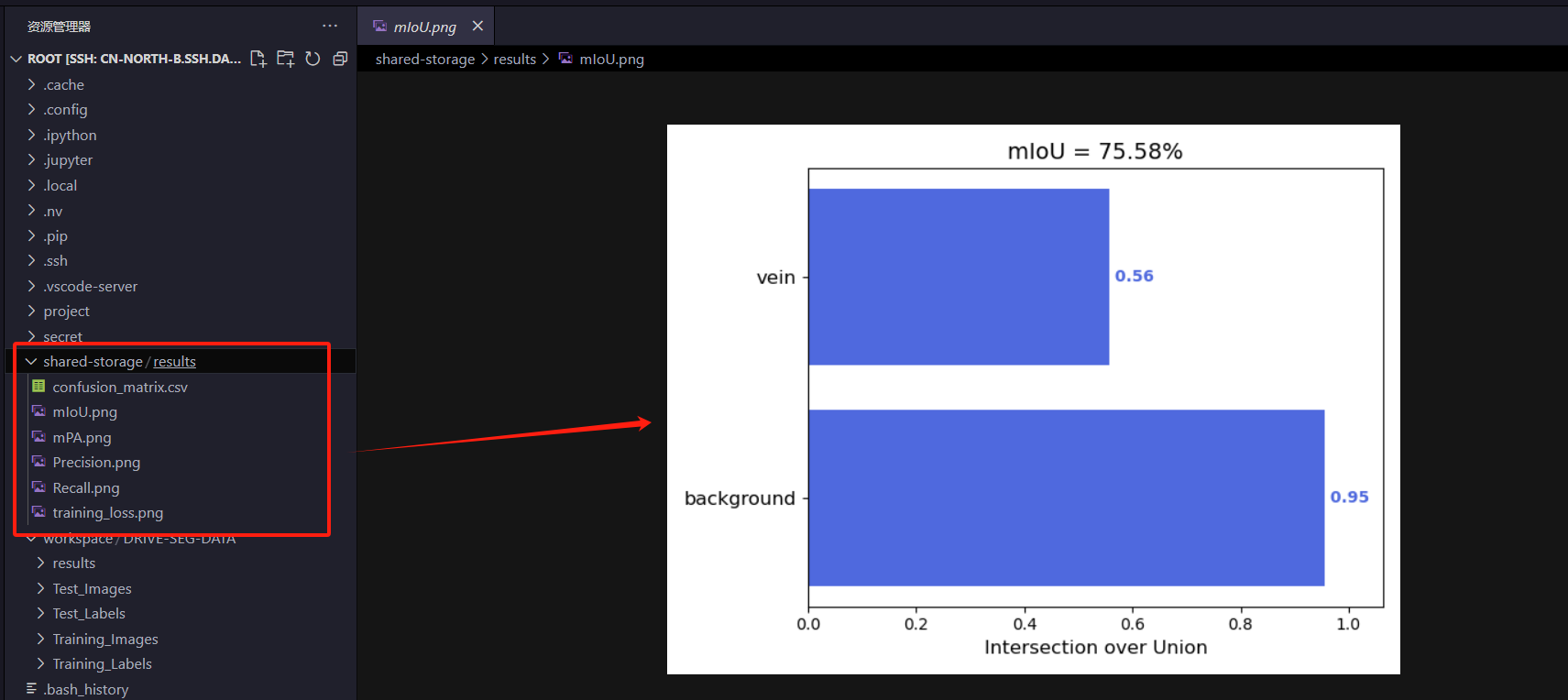
四、模型测试与验证
在训练完成后,需要对模型进行验证,以评估其在未见数据上的表现。常见的评估指标包括精度(Accuracy)、交并比(IoU, Intersection over Union)以及 Dice 系数。使用 DAMODEL 云平台,用户可以快速运行模型的验证,并生成分割结果图像。以下是测试模型的基本流程:
model.eval()
with torch.no_grad():
for inputs, labels in test_loader:
inputs = inputs.to(device)
outputs = model(inputs)
# 计算评估指标,并可视化分割结果
用户可以将测试结果下载到本地,或者直接在 DAMODEL 平台上进行可视化分析。
五、总结
UNet 作为一种强大的图像分割模型,在医学领域尤其受到关注。通过 DAMODEL 平台,用户可以充分利用云计算资源,快速进行模型的训练与测试。该平台不仅提供了便捷的实例管理,还支持大规模数据集的处理和深度学习模型的加速,适合各类医学影像分析任务。
通过本文的介绍,相信读者已经对如何在 DAMODEL 上使用 UNet 进行图像分割有了清晰的了解,并可以在实际项目中应用这一流程,提高工作效率。
更多推荐
 已为社区贡献18条内容
已为社区贡献18条内容









所有评论(0)