如何设计qPCR引物序列
qPCR引物序列设计
感谢李三多师弟的倾囊相授!
- 文献中寻找序列
- PrimerBank 中寻找(https://pga.mgh.harvard.edu/primerbank/)
本次着重第二种方法
打开PubMed的Gene界面(https://www.ncbi.nlm.nih.gov/),输入需要查询的基因名称(如METTL3),确认好种属(如小鼠),查询Gene ID



复制Gene ID,打开 PrimerBank,依次选好检索条件,点击Submit查询(可能等待时间稍长)。(https://pga.mgh.harvard.edu/primerbank/)

结果如下:

查看的细节如下:

*
比如挑中这一对,到BLAST里验证:

找出序列后,在Pubmed(https://www.ncbi.nlm.nih.gov/)里的BLAST进行模拟。

页面往下滑,选择Primer-BLAST。


由于在新窗口打开结果,我们可以尝试下一对引物,如复制粘贴Primer pair 2的正反引物进行比较。
操作同上。

出现这个页面的话点Check查看结果

共验证了四对引物,来看看结果比较:




通过序列和Length定位到这个序列:

挑选引物的细节*:
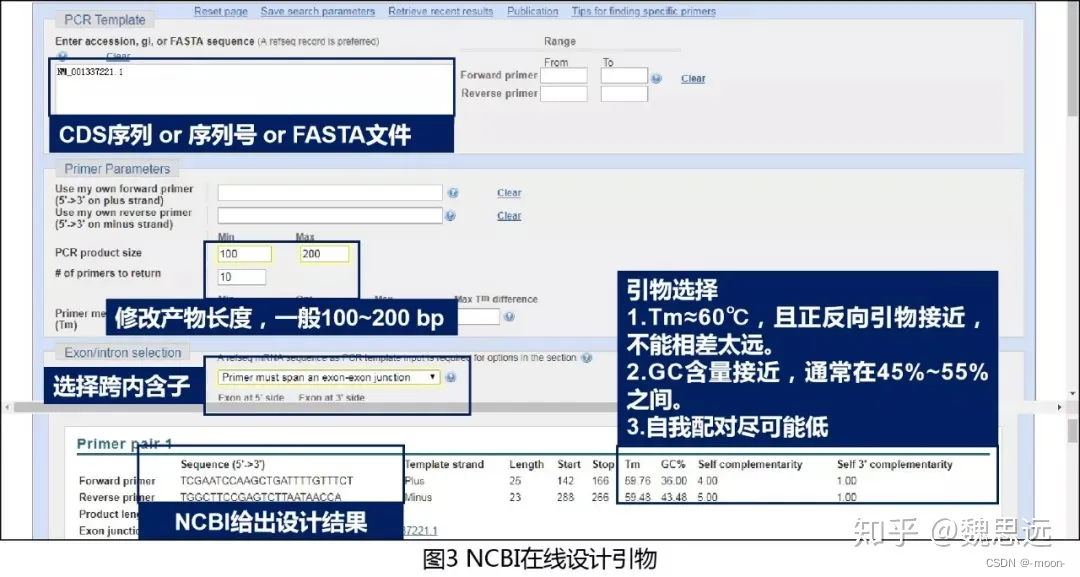
①Forward和Reverseprimers 退火温度(Tm) 55-75℃,60℃为宜,且相差不超过4℃。更严格的标准有:Tm相差最好不超过1
②引物的GC含量一般为40-60%,以45-55%为宜,过高或过低都不利于引发反应。上下游引物的 GC含量不能相差太大
③给出的数对引物中,Amplicon Size一般小于200,大于200初筛即可淘汰。PCR扩增产物长度: 引物的产物大小不要太大,一般在80-250bp之间都可;80-150bp最为合适(可以延长至300 bp)
④尽可能多尝试,找出最合适的
⑤Self complementarity0-3为理想状态;self 3’ complementarity 严格<1/4碱基数。总的来说,越低越好。对应的数值越小,非特异性扩增和引物二聚体就越少。Max Self Complementarity 默认值是8.00,Max 3’ Self Complementarity 默认值是3.00
⑥引物长度一般在15-30碱基之间。引物长度18-27bp,不大于38bp
⑦3’端碱基一般不用A,最好用T,3’端避开密码子第三位,3’端不应该有超过3个连续的C或G
⑧transcript variant是转录本的意思,具体含义可以自行百度。大多数基因都有好几个转录本,转录本越多说明这个引物越好。
⑨跨内含子的勾选,参考如下图:若出不来结果,尝试取消此筛选
⑩若别无他选,有其他干扰的物质的产物量大于1000,PCR扩增不出来此干扰产物,可考虑
参考来源:
https://www.zhihu.com/question/457819740
https://zhuanlan.zhihu.com/p/76211729
https://blog.sciencenet.cn/home.php?mod=space&uid=1509670&do=blog&quickforward=1&id=1094003
https://zhuanlan.zhihu.com/p/103537195
https://www.biomart.cn/experiment/793/2714179.htm
https://www.biomart.cn/experiment/430/457/741/43975.htm
https://www.jianshu.com/p/5fba2bc168d4
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容










所有评论(0)