Quantile g-computation的介绍及R实现
介绍qgcomp是一个实现g-computation的软件包,用于分析暴露混合物的影响。分位数g-computation产生了所有暴露同时增加一个分位数的效果的估计。因此,它估计的 "混合物效应 "对研究暴露混合物(如空气污染、饮食和水污染)很有用。 使用为因果效应估计而开发的术语,分位数g计算估计了边际结构模型的参数,该模型描述了在对所有暴露进行联合干预的情况下预期潜在结果的变化,可能以混杂因素
目录
介绍
qgcomp是一个实现g-computation的软件包,用于分析暴露混合物的影响。分位数g-computation产生了所有暴露同时增加一个分位数的效果的估计。因此,它估计的 "混合物效应 "对研究暴露混合物(如空气污染、饮食和水污染)很有用。 使用为因果效应估计而开发的术语,分位数g计算估计了边际结构模型的参数,该模型描述了在对所有暴露进行联合干预的情况下预期潜在结果的变化,可能以混杂因素为条件。在可交换性、因果一致性、阳性、无干扰和正确的模型规范的假设下,这个模型产生了干预对整个混合物的因果效应。虽然这些假设不一定完全满足,但它们为如何解释qgcomp拟合的结果提供了一个有用的路线图,以及在确保准确的模型规格和选择足以控制共同污染物混杂的暴露方面应该花费的努力。
模型
假设我们有一个结果Y,一些暴露X,可能还有一些其他协变量(例如潜在的混杂因素),用ZZ表示。
quantile g-computation的基本模型是一个联合边际结构模型,由以下公式给出
E(YXq|Z,ψ,η)=g(ψ0+ψ1Sq+ηZ)
其中g(⋅)是广义线性模型中的链接函数(例如,在Logistic模型中Y=1的概率的反logit函数),ψ0是模型截距,η是一组协变量的模型系数,Sq是一个代表暴露的联合值的 "指数"。Quantile g-computation(默认情况下)将所有暴露X转化为Xq,它是 "分数",具有离散值0,1,2等,代表暴露的分类 "bin"。默认情况下,有四个bin,每个暴露的四分位切点均匀分布,所以Xq=0意味着X低于该暴露的观察到的25百分位。指数Sq代表所有暴露被设置为相同的值(同样,在默认情况下,离散值0,1,2,3)。因此,参数ψ1量化了结果的预期变化,鉴于所有暴露同时增加一个四分位数,可能会对Z进行调整。
这种特殊的模型形式有一些细微的差别,在qgcomp软件包中可以找到,下面将进行探讨。在quantile g-computation中存在一种特殊情况,可以实现快速拟合:线性/相加暴露效应。在这里,我们模拟了 "预量化 "数据,其中暴露X1,X2,X3只能以相等的比例取值0,1,2,3。结果的基础模型是由线性回归给出的。
E(Y|X,β)=β0+β1X1+β2X2+β3X3
β0=0,β1=0.25,β2=-0.1,β3=0.05,X1与X2强正相关(ρ=0.95),与X3负相关(ρ=-0.3)。在这个简单的设定中,参数ψ1将等于β系数之和(0.2)。在这里,我们看到qgcomp估计的值非常接近0.2(随着我们增加样本量,估计值将会越来越接近0.2)。
library("qgcomp")
set.seed(543210)
qdat = simdata_quantized(n=5000, outcomtype="continuous", cor=c(.95, -0.3), b0=0, coef=c(0.25, -0.1, 0.05), q=4)
head(qdat)
## x1 x2 x3 y
## 1 2 2 3 0.5539253
## 2 2 3 1 -0.1005701
## 3 2 2 3 0.3782267
## 4 1 2 2 -0.4557419
## 5 0 0 1 0.6124436
## 6 1 1 2 0.7569574
cor(qdat[,c("x1", "x2", "x3")])
## x1 x2 x3
## x1 1.00000 0.95552 -0.30368
## x2 0.95552 1.00000 -0.29840
## x3 -0.30368 -0.29840 1.00000
qgcomp(y~x1+x2+x3, expnms=c("x1", "x2", "x3"), data=qdat)
## Scaled effect size (positive direction, sum of positive coefficients = 0.3)
## x1 x3
## 0.775 0.225
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.0966)
## x2
## 1
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.0016583 0.0359609 -0.07214 0.068824 -0.0461 0.9632
## psi1 0.2035810 0.0219677 0.16053 0.246637 9.2673 <2e-16qgcomp包的使用
在这里,我们使用来自qgcomp包的metal数据集的一个运行实例来演示该包的一些特征和方法。
也就是说,下面的例子展示了该包在以下方面的使用。1. 在连续(正常)结果的量化暴露的线性模型下,快速估计暴露效应 2. 估算二元结果的混合效应的条件和边际几率/风险比率 3. 在估计混合物效应时调整非暴露的协变量 4. 通过包括乘积项,允许单个暴露和整个混合物的非线性和非同质效应 5. 使用qgcomp拟合时间-事件模型来估计暴露混合物的条件和边际危险比。
对于估计暴露混合效应的类似方法,可以在gQWS软件包的帮助文件中看到说明性的例子,它实现了加权量化和(WQS)的回归,以及在 https://jenfb.github.io/bkmr/overview.html链接中描述的贝叶斯核机回归。
来自软件包qgcomp的metal数据集,包括一组模拟井水暴露和两个健康结局(一个是连续的,一个是二进制/时间-事件)。暴露被转换为平均值=0.0,标准偏差=1.0。这些数据被用来展示qgcomp软件包的使用和功能。
library("qgcomp")
library("knitr")
library("ggplot2")
data("metals", package="qgcomp")
head(metals)
## arsenic barium cadmium calcium chloride chromium
## 1 0.09100165 0.08166362 15.0738845 -0.7746662 -0.15408335 -0.05589104
## 2 0.17018302 -0.03598828 -0.7126486 -0.6857886 -0.19605499 -0.03268488
## 3 0.13336869 0.09934014 0.6441992 -0.1525231 -0.17511844 -0.01161098
## 4 -0.52570747 -0.76616263 -0.8610256 1.4472733 0.02552401 -0.05173287
## 5 0.43420529 0.40629920 0.0570890 0.4103682 -0.24187403 -0.08931824
## 6 0.71832662 0.19559582 -0.6823437 -0.8931696 -0.03919936 -0.07389407
## copper iron lead magnesium manganese mercury
## 1 1.99438050 19.1153352 21.072630908 -0.5109546 2.07630966 -1.20826726
## 2 -0.02490169 -0.2039425 -0.010378362 -0.1030542 -0.36095395 -0.68729723
## 3 0.25700811 -0.1964581 -0.063375935 0.9166969 -0.31075240 0.44852503
## 4 0.75477075 -0.2317787 -0.002847991 2.5482987 -0.23350205 0.20428158
## 5 -0.09919923 -0.1698619 -0.035276281 -0.5109546 0.08825996 1.19283834
## 6 -0.05622285 -0.2129300 -0.118460981 -1.0059145 -0.30219838 0.02875033
## nitrate nitrite ph selenium silver sodium
## 1 1.3649492 -1.0500539 -0.7125482 0.23467592 -0.8648653 -0.41840695
## 2 -0.1478382 0.4645119 0.9443009 0.65827253 -0.8019173 -0.09112969
## 3 -0.3001660 -1.4969868 0.4924330 0.07205576 -0.3600140 -0.11828963
## 4 0.3431814 -0.6992263 -0.4113029 0.23810705 1.3595205 -0.11828963
## 5 0.0431269 -0.5041390 0.3418103 -0.02359910 -1.6078044 -0.40075299
## 6 -0.3986575 0.1166249 1.2455462 -0.61186017 1.3769466 1.83722597
## sulfate total_alkalinity total_hardness zinc mage35 y
## 1 -0.1757544 -1.31353389 -0.85822417 1.0186058 1 -0.6007989
## 2 -0.1161359 -0.12699789 -0.67749970 -0.1509129 0 -0.2022296
## 3 -0.1616806 0.42671890 0.07928399 -0.1542524 0 -1.2164116
## 4 0.8272415 0.99173604 1.99948142 0.1843372 0 0.1826311
## 5 -0.1726845 -0.04789549 0.30518957 -0.1529379 0 1.1760472
## 6 -0.1385631 1.98616621 -1.07283447 -0.1290391 0 -0.4100912
## disease_time disease_state
## 1 6.168764e-07 1
## 2 4.000000e+00 0
## 3 4.000000e+00 0
## 4 4.000000e+00 0
## 5 1.813458e+00 1
## 6 2.373849e+00 1
例1,线性模型
# we save the names of the mixture variables in the variable "Xnm"
Xnm <- c(
'arsenic','barium','cadmium','calcium','chromium','copper',
'iron','lead','magnesium','manganese','mercury','selenium','silver',
'sodium','zinc'
)
covars = c('nitrate','nitrite','sulfate','ph', 'total_alkalinity','total_hardness')
# Example 1: linear model
# Run the model and save the results "qc.fit"
system.time(qc.fit <- qgcomp.noboot(y~.,dat=metals[,c(Xnm, 'y')], family=gaussian()))
## user system elapsed
## 0.020 0.001 0.027
# user system elapsed
# 0.011 0.002 0.018
# contrasting other methods with computational speed
# WQS regression
#system.time(wqs.fit <- gwqs(y~NULL,mix_name=Xnm, data=metals[,c(Xnm, 'y')], family="gaussian"))
# user system elapsed
# 35.775 0.124 36.114
# Bayesian kernel machine regression (note that the number of iterations here would
# need to be >5,000, at minimum, so this underestimates the run time by a factor
# of 50+
#system.time(bkmr.fit <- kmbayes(y=metals$y, Z=metals[,Xnm], family="gaussian", iter=100))
# user system elapsed
# 81.644 4.194 86.520
#first note that qgcomp is very fast
# View results: scaled coefficients/weights and statistical inference about
# mixture effect
qc.fit
## Scaled effect size (positive direction, sum of positive coefficients = 0.39)
## calcium iron barium silver arsenic mercury sodium chromium
## 0.72216 0.06187 0.05947 0.03508 0.03447 0.02451 0.02162 0.02057
## cadmium zinc
## 0.01328 0.00696
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.124)
## magnesium copper lead manganese selenium
## 0.475999 0.385299 0.074019 0.063828 0.000857
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value
## (Intercept) -0.356670 0.107878 -0.56811 -0.14523 1e-03
## psi1 0.266394 0.071025 0.12719 0.40560 2e-04
与其他估计 "混合效应"(一次性改变所有暴露的影响)的方法相比,Quantile g-computation的一个优势是它的计算效率非常高。与WQS(gWQS包)和贝叶斯核机器回归(bkmr包)等方法相比,Quantile g-computation可以提供快很多个数量级的结果。例如,上面的例子中,Quantile g-computation比WQS回归快3000倍,我们估计比贝叶斯核机器回归的速度要快几十万倍。
量子g计算在估计过程中产生了固定的权重,与WQS回归类似。然而,请注意,来自qgcomp.noboot的权重可以是负的或正的。当所有的效应都是线性的且方向相同时("方向同质性"),量化g-computation相当于大样本中的加权量化和回归。
来自量化g计算的总体混合效应(psi1)被解释为对每个暴露增加一个量化的结果的影响,可能以协变量为前提。鉴于总体暴露效应,权重被认为是固定的,因此没有置信区间或P值。
例2:条件OR,logistic模型中的边际OR
这个例子介绍了在qgcomp中通过qgcomp.noboot函数使用二元结果的情况,该函数产生的是条件性OR,或qgcomp.boot产生的是边缘性OR或风险/流行率。当模型中包含有非暴露协变量(如混杂因素)时,这些参数不会相互相等,因为OR是不可折叠的(两者仍然有效)。边际参数将产生人群平均暴露效应的估计值,由于比条件性的几率有更好的可解释性,所以通常更有意义。此外,在风险比可以有效估计的情况下,一般不估计OR,所以qgcomp.boot将默认估计二元数据的风险比(使用qgcomp.boot时设置rr=FALSE以允许估计OR)。
qc.fit2 <- qgcomp.noboot(disease_state~., expnms=Xnm,
data = metals[,c(Xnm, 'disease_state')], family=binomial(),
q=4)
qcboot.fit2 <- qgcomp.boot(disease_state~., expnms=Xnm,
data = metals[,c(Xnm, 'disease_state')], family=binomial(),
q=4, B=10,# B should be 200-500+ in practice
seed=125, rr=FALSE)
qcboot.fit2b <- qgcomp.boot(disease_state~., expnms=Xnm,
data = metals[,c(Xnm, 'disease_state')], family=binomial(),
q=4, B=10,# B should be 200-500+ in practice
seed=125, rr=TRUE)
# Compare a qgcomp.noboot fit:
qc.fit2
## Scaled effect size (positive direction, sum of positive coefficients = 0.392)
## barium zinc chromium magnesium silver sodium
## 0.3520 0.2002 0.1603 0.1292 0.0937 0.0645
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.696)
## selenium copper arsenic calcium manganese cadmium mercury lead
## 0.2969 0.1627 0.1272 0.1233 0.1033 0.0643 0.0485 0.0430
## iron
## 0.0309
##
## Mixture log(OR) (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## (Intercept) 0.26362 0.51615 -0.74802 1.27526 0.5107 0.6095
## psi1 -0.30416 0.34018 -0.97090 0.36258 -0.8941 0.3713
# and a qgcomp.boot fit:
qcboot.fit2
## Mixture log(OR) (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## (Intercept) 0.26362 0.38110 -0.48332 1.01056 0.6917 0.4891
## psi1 -0.30416 0.27478 -0.84272 0.23439 -1.1070 0.2683
# and a qgcomp.boot fit, where the risk/prevalence ratio is estimated,
# rather than the odds ratio:
qcboot.fit2b
## Mixture log(RR) (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## (Intercept) -0.56237 0.15716 -0.87040 -0.25434 -3.5783 0.0003458
## psi1 -0.16373 0.13611 -0.43049 0.10304 -1.2029 0.2290108
例3:调整协变量,绘制估计值
在下面的代码中,我们用qgcomp(family = "gaussian")运行一个产妇年龄调整的线性模型。此外,我们绘制了权重以及混合斜率,产生了整体模型的置信度,代表了在95%的试验中,每个联合暴露值都有望包含真正的回归线的界限(所谓的回归线的95%'pointwise'的界限)。点式比较界线,用图上的误差条表示,表示参照特定的四分位数(可由用户指定,如下图),对每个四分位数的预期结果差异进行比较。(这些界限类似于bkmr包在绘制所有暴露的总体效应时创建的界限),点式界限可以通过pointwisebound.boot函数获得。为了避免混淆 "顺时针回归 "和 "顺时针比较 "的界限,顺时针回归的界限在图中被称为 "模型置信区",因为它们在应用于线性模型拟合时产生的估计界限与R中的预测函数的类型相同。
请注意,底层的回归模型是在量化的 "分数 "上,其取值为整数的0,1,...,q。为了绘图的目的(当绘制来自qgcomp.boot的回归线结果时),分位数得分被转化为分位数(范围=[0-1])。这并不是一个完美的对应,因为Quantile g-computation模型将分位数得分视为一个连续变量,但分位数类别包括一个分位数得分范围。为了可视化,我们把图的两端固定在第一个和最后一个量化分界点的中点上,所以如果'q'发生变化,图的范围也会略有变化。
qc.fit3 <- qgcomp.noboot(y ~ mage35 + arsenic + barium + cadmium + calcium + chloride +
chromium + copper + iron + lead + magnesium + manganese +
mercury + selenium + silver + sodium + zinc,
expnms=Xnm,
metals, family=gaussian(), q=4)
qc.fit3
## Scaled effect size (positive direction, sum of positive coefficients = 0.381)
## calcium barium iron silver arsenic mercury chromium zinc
## 0.74466 0.06636 0.04839 0.03765 0.02823 0.02705 0.02344 0.01103
## sodium cadmium
## 0.00775 0.00543
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.124)
## magnesium copper lead manganese selenium
## 0.49578 0.35446 0.08511 0.06094 0.00372
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value
## (Intercept) -0.348084 0.108037 -0.55983 -0.13634 0.0014
## psi1 0.256969 0.071459 0.11691 0.39703 0.0004
plot(qc.fit3)
qcboot.fit3 <- qgcomp.boot(y ~ mage35 + arsenic + barium + cadmium + calcium + chloride +
chromium + copper + iron + lead + magnesium + manganese +
mercury + selenium + silver + sodium + zinc,
expnms=Xnm,
metals, family=gaussian(), q=4, B=10,# B should be 200-500+ in practice
seed=125)
qcboot.fit3
## Mixture slope parameters (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI t value
## (Intercept) -0.342787 0.114983 -0.56815 -0.11742 3e-03
## psi1 0.256969 0.075029 0.10991 0.40402 7e-04
p = plot(qcboot.fit3)
plot(qcboot.fit3, pointwiseref = 3)
pointwisebound.boot(qcboot.fit3, pointwiseref=3)
## quantile quantile.midpoint y.expected mean.diff se.diff ul.pw
## 0 0 0.125 -0.34278746 -0.5139387 0.15005846 -0.04867828
## 1 1 0.375 -0.08581809 -0.2569694 0.07502923 0.06123650
## 2 2 0.625 0.17115127 0.0000000 0.00000000 0.17115127
## 3 3 0.875 0.42812064 0.2569694 0.07502923 0.57517523
## ll.pw
## 0 -0.6368966
## 1 -0.2328727
## 2 0.1711513
## 3 0.2810660
qgcomp:::modelbound.boot(qcboot.fit3)
## quantile quantile.midpoint y.expected se.pw ll.pw ul.pw
## 0 0 0.125 -0.34278746 0.11498301 -0.56815001 -0.117424905
## 1 1 0.375 -0.08581809 0.04251388 -0.16914376 -0.002492425
## 2 2 0.625 0.17115127 0.04065143 0.09147593 0.250826606
## 3 3 0.875 0.42812064 0.11294432 0.20675384 0.649487428
## ll.simul ul.simul
## 0 -0.4622572 -0.161947578
## 1 -0.1191843 -0.005233921
## 2 0.1386622 0.223888629
## 3 0.3081934 0.566961520



从第一个图中,我们看到来自qgcomp.noboot函数的权重,其中包括正负效应方向。当权重都在null的单边时,这些图很容易解释,因为权重对应的是每个暴露的总体效应的比例。WQS在模型中使用了一个约束条件,强迫所有的权重都在同一方向--不幸的是,这种约束条件导致了有偏见的效应估计。qgcomp软件包采取了不同的方法,允许 "权重 "可能朝任何一个方向发展,表明有些暴露可能是有益的,有些是有害的,或者由于使用小规模或中等规模的样本,可能存在抽样变化(或者,更常见的是系统偏差,如未测量的混合)。qgcomp中的 "权重 "对应于所有暴露在同一方向上的效应的比例,但在其他情况下,它们对应于特定方向上的效应的比例,与整体 "混合 "效应相比,它可能很小(或很大)。注意:图中的左边和右边不应相互比较,因为条形图的长度只对应于相对于同一方向的其他效应的大小。条形图的深浅与总体效应大小相对应--在这种情况下,图形右边(正)的条形图更深,因为总体 "混合 "效应是正的。因此,阴影允许人们在左右两边进行非正式的比较:一个大的、深色阴影的条形图表示比一个大的、浅色阴影的条形图有更大的独立效应。
使用qgcomp.boot还可以让我们评估总暴露效应的线性(第二幅图)。类似的输出可用于WQS(gWQS包),尽管当暴露效应为非线性时,WQS的结果通常不太容易解释(见下文如何用qgcomp.boot做这个)。
qcboot.fit3对象的图(使用g-computation与bootstrap方差)给出了暴露的联合干预水平的预测。它还显示了一个平滑的(图形)拟合。一般来说,我们不能把数据叠加在这个图上,因为回归线对应的是一次可能的许多暴露的变化。因此,通过拟合允许非线性效应的模型来探索非线性是很有用的。
例4:非线性(和非同质性)
让我们用qgcomp(和qgcomp.boot)的另一个特点来结束:处理非线性。这里有一个例子,我们使用R语言的一个特点来拟合有交互项的模型。我们使用y~. + .^2作为模型公式,它拟合了一个模型,允许模型中的每个预测因子都有二次项。
类似的方法可以用来包括暴露之间的交互项,以及暴露和协变量之间的交互项。
qcboot.fit4 <- qgcomp(y~. + .^2,
expnms=Xnm,
metals[,c(Xnm, 'y')], family=gaussian(), q=4, B=10, seed=125)
plot(qcboot.fit4)
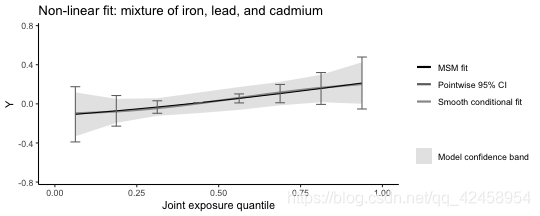
请注意,允许所有暴露的非线性效应会在整体暴露效应中引起明显的非线性趋势。平滑化的回归线仍然在边际线性模型的置信区间内(默认情况下,联合暴露的总体效应被假定为线性,尽管这一假定可以通过qgcomp.boot中的 "程度 "参数放宽,如下所示。
qcboot.fit5 <- qgcomp(y~. + .^2,
expnms=Xnm,
metals[,c(Xnm, 'y')], family=gaussian(), q=4, degree=2,
B=10, rr=FALSE, seed=125)
qgcomp::pointwisebound.boot(qcboot.fit5)
## quantile quantile.midpoint y.expected mean.diff se.diff ul.pw
## 0 0 0.125 -0.89239044 0.0000000 0.0000000 -0.8923904
## 1 1 0.375 -0.18559680 0.7067936 0.6560598 1.1002568
## 2 2 0.625 0.12180659 1.0141970 0.6480848 1.3920295
## 3 3 0.875 0.02981974 0.9222102 0.2803460 0.5792877
## ll.pw
## 0 -0.8923904
## 1 -1.4714504
## 2 -1.1484163
## 3 -0.5196482
qgcomp:::modelbound.boot(qcboot.fit5)
## quantile quantile.midpoint y.expected se.pw ll.pw ul.pw
## 0 0 0.125 -0.89239044 0.73835876 -2.3395470 0.554766138
## 1 1 0.375 -0.18559680 0.09639511 -0.3745278 0.003334151
## 2 2 0.625 0.12180659 0.12502104 -0.1232301 0.366843327
## 3 3 0.875 0.02981974 0.82096521 -1.5792425 1.638881990
## ll.simul ul.simul
## 0 -2.08917795 0.23867330
## 1 -0.32419926 -0.02762852
## 2 -0.02981037 0.39857469
## 3 -1.10497070 1.47916561
plot(qcboot.fit5)

理想情况下,平滑拟合看起来与模型预测的回归线非常相似。
例5:比较模型拟合和进一步探索非线性的问题
在有多种暴露的情况下探索非线性拟合是具有挑战性的。如上所述,探索非线性的一种方法是包括所有的双向交互项(包括二次项,或 "自我交互")。有时这种方法并不可取,因为模型中的项的数量会变得非常大,或者因为需要某种模型选择程序,这有可能引起过度拟合(有偏见的估计和标准误差太小)。如果没有一套先验的非线性项,我们发现最好是采取一种不依赖统计学意义的默认方法(例如,采取所有的二阶项),或者干脆坦白地说,寻找非线性模型是探索性的,不应该依赖稳健推断。核心机器回归等方法可能是很好的替代品,或者是探索非线性的补充方法。
注意:qgcomp必须根据所选择的量化指标,用具有少量可能值的暴露量来拟合回归模型。根据包的默认值,这是q=4,但只用四个点很难完全考察非线性拟合,所以我们建议探索更大的q值,这将改变效果估计(即模型系数意味着暴露的变化较小,所以结果的预期变化也会减少)。
在这里,我们研究了默认的和探索性的混合物的一对一策略,可以在qgcomp中使用较小的暴露子集(铁、铅、镉)来实现,我们通过相关矩阵来选择。暴露之间的高相关性可能来自于一个共同的来源,所以小的混合物子集可能有助于研究与对共同环境来源或一系列行为的干预有关的假设。请注意,尽管只有3个我们感兴趣的暴露被认为是感兴趣的混合物,但我们仍然可以对测量的暴露进行调整。请注意,我们需要一个新的R包来帮助探索非线性:splines。请注意,必须使用qgcomp.boot才能产生下面的图形,因为qgcomp.noboot不计算必要的数量。
使用样条曲线探索曝光的相关子集的非线性的图形方法。
library(splines)
# find all correlations > 0.6 (this is an arbitrary choice)
cormat = cor(metals[,Xnm])
idx = which(cormat>0.6 & cormat <1.0, arr.ind = TRUE)
newXnm = unique(rownames(idx)) # iron, lead, and cadmium
qc.fit6lin <- qgcomp.boot(y ~ iron + lead + cadmium +
mage35 + arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc,
expnms=newXnm,
metals, family=gaussian(), q=8, B=10)
qc.fit6nonlin <- qgcomp.boot(y ~ bs(iron) + bs(cadmium) + bs(lead) +
mage35 + arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc,
expnms=newXnm,
metals, family=gaussian(), q=8, B=10, degree=2)
qc.fit6nonhom <- qgcomp.boot(y ~ bs(iron)*bs(lead) + bs(iron)*bs(cadmium) + bs(lead)*bs(cadmium) +
mage35 + arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc,
expnms=newXnm,
metals, family=gaussian(), q=8, B=10, degree=3)
# it helps to place the plots on a common y-axis, which is easy due
# to dependence of the qgcomp plotting functions on ggplot
pl.fit6lin <- plot(qc.fit6lin, suppressprint = TRUE, pointwiseref = 4)
pl.fit6nonlin <- plot(qc.fit6nonlin, suppressprint = TRUE, pointwiseref = 4)
pl.fit6nonhom <- plot(qc.fit6nonhom, suppressprint = TRUE, pointwiseref = 4)
pl.fit6lin + coord_cartesian(ylim=c(-0.75, .75)) +
ggtitle("Linear fit: mixture of iron, lead, and cadmium")
pl.fit6nonlin + coord_cartesian(ylim=c(-0.75, .75)) +
ggtitle("Non-linear fit: mixture of iron, lead, and cadmium")
pl.fit6nonhom + coord_cartesian(ylim=c(-0.75, .75)) +
ggtitle("Non-linear, non-homogeneous fit: mixture of iron, lead, and cadmium")



关于图形化方法的注意事项
基础条件模型拟合可以变得非常灵活,其图形表示(通过平滑条件拟合)看起来也非常灵活。简单地将整体(MSM)拟合与这条线相匹配并不是识别拟合模型的可行策略,因为这将忽略潜在的过度拟合。因此,在比较 "平滑条件拟合 "和 "MSM拟合 "时,判断拟合的准确性应谨慎。在这里,甚至连线性趋势的统计证据都很少,这使得平滑条件拟合看起来是超拟合的。可以关闭平滑条件拟合,如下图所示。
qc.overfit <- qgcomp.boot(y ~ bs(iron) + bs(cadmium) + bs(lead) +
mage35 + bs(arsenic) + bs(magnesium) + bs(manganese) + bs(mercury) +
bs(selenium) + bs(silver) + bs(sodium) + bs(zinc),
expnms=Xnm,
metals, family=gaussian(), q=8, B=10, degree=1)
qc.overfit
## Mixture slope parameters (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI t value
## (Intercept) -0.064420 0.123768 -0.307001 0.178162 0.6030
## psi1 0.029869 0.031972 -0.032795 0.092534 0.3507
plot(qc.overfit, pointwiseref = 5)
plot(qc.overfit, flexfit = FALSE, pointwiseref = 5)


例6:允许非线性的其他杂项方式
请注意,这些是作为如何包括非线性的例子,而不是作为适当模型选择的示范。事实上,由于参数较多,qc.fit7b在小到中等规模的样本中通常是个坏主意。
# using indicator terms for each quantile
qc.fit7a <- qgcomp.boot(y ~ factor(iron) + lead + cadmium +
mage35 + arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc,
expnms=newXnm,
metals, family=gaussian(), q=8, B=20, deg=2)
# underlying fit
summary(qc.fit7a$fit)$coefficients
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.052981109 0.08062430 -0.6571357 5.114428e-01
## factor(iron)1 0.046571725 0.09466914 0.4919420 6.230096e-01
## factor(iron)2 -0.056984300 0.09581659 -0.5947227 5.523395e-01
## factor(iron)3 0.143813131 0.09558055 1.5046276 1.331489e-01
## factor(iron)4 0.053319057 0.09642069 0.5529835 5.805601e-01
## factor(iron)5 0.303967859 0.09743261 3.1197753 1.930856e-03
## factor(iron)6 0.246259568 0.09734385 2.5297907 1.176673e-02
## factor(iron)7 0.447045591 0.09786201 4.5681217 6.425565e-06
## lead -0.009210341 0.01046668 -0.8799679 3.793648e-01
## cadmium -0.010503041 0.01086440 -0.9667387 3.342143e-01
## mage35 0.081114695 0.07274583 1.1150426 2.654507e-01
## arsenic 0.021755516 0.02605850 0.8348720 4.042502e-01
## magnesium -0.010758356 0.02469893 -0.4355798 6.633587e-01
## manganese 0.004418266 0.02551449 0.1731670 8.626011e-01
## mercury 0.003913896 0.02448078 0.1598763 8.730531e-01
## selenium -0.058085344 0.05714805 -1.0164012 3.100059e-01
## silver 0.020971562 0.02407397 0.8711302 3.841658e-01
## sodium -0.062086322 0.02404447 -2.5821454 1.014626e-02
## zinc 0.017078438 0.02392381 0.7138679 4.756935e-01
plot(qc.fit7a)
# interactions between indicator terms
qc.fit7b <- qgcomp.boot(y ~ factor(iron)*factor(lead) + cadmium +
mage35 + arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc,
expnms=newXnm,
metals, family=gaussian(), q=8, B=10, deg=3)
# underlying fit
#summary(qc.fit7b$fit)$coefficients
plot(qc.fit7b)
# breaks at specific quantiles (these breaks act on the quantized basis)
qc.fit7c <- qgcomp.boot(y ~ I(iron>4)*I(lead>4) + cadmium +
mage35 + arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc,
expnms=newXnm,
metals, family=gaussian(), q=8, B=10, deg=2)
# underlying fit
summary(qc.fit7c$fit)$coefficients
## Estimate Std. Error t value
## (Intercept) -5.910113e-02 0.05182385 -1.140423351
## I(iron > 4)TRUE 3.649940e-01 0.06448858 5.659824144
## I(lead > 4)TRUE -9.004067e-05 0.06181587 -0.001456595
## cadmium -6.874749e-03 0.01078339 -0.637531252
## mage35 7.613672e-02 0.07255110 1.049422029
## arsenic 2.042370e-02 0.02578001 0.792230124
## magnesium -3.279980e-03 0.02427513 -0.135116878
## manganese 1.055979e-02 0.02477453 0.426235507
## mercury 9.396898e-03 0.02435057 0.385900466
## selenium -4.337729e-02 0.05670006 -0.765030761
## silver 1.807248e-02 0.02391112 0.755819125
## sodium -5.537968e-02 0.02403808 -2.303831424
## zinc 2.349906e-02 0.02385762 0.984970996
## I(iron > 4)TRUE:I(lead > 4)TRUE -1.828835e-01 0.10277790 -1.779405131
## Pr(>|t|)
## (Intercept) 2.547332e-01
## I(iron > 4)TRUE 2.743032e-08
## I(lead > 4)TRUE 9.988385e-01
## cadmium 5.241120e-01
## mage35 2.945626e-01
## arsenic 4.286554e-01
## magnesium 8.925815e-01
## manganese 6.701456e-01
## mercury 6.997578e-01
## selenium 4.446652e-01
## silver 4.501639e-01
## sodium 2.169944e-02
## zinc 3.251821e-01
## I(iron > 4)TRUE:I(lead > 4)TRUE 7.586670e-02
plot(qc.fit7c)



请注意探索非线性的一个限制:虽然我们可以使用灵活的函数,如对单个暴露的splines,但整体拟合通过程度参数限制在多项式函数上(这里二次多项式很适合非线性模型,三次多项式很适合非线性/非同质模型--尽管这只是一个非正式的论证,没有说明宽的置信区间)。我们在此注意到,只用了10次自举迭代来计算置信区间(以提高该例子的计算速度),这太低了。
统计学方法使用样条探索暴露的相关子集的非线性问题
图形方法不能清楚地说明哪种模型可能是首选,但我们可以用AIC、AICC或BIC(都是各种形式的信息准则,权衡模型拟合与过度参数化)来比较模型的拟合。这两个标准都表明线性模型的拟合效果最好(AIC和BIC最低),这表明相对于线性拟合,在图形方法中观察到的明显的非线性拟合并不能提高对健康结果的预测,因为包括更多的参数会导致方差的增加。
AIC(qc.fit6lin$fit)
## [1] 676.0431
AIC(qc.fit6nonlin$fit)
## [1] 682.7442
AIC(qc.fit6nonhom$fit)
## [1] 705.6187
BIC(qc.fit6lin$fit)
## [1] 733.6346
BIC(qc.fit6nonlin$fit)
## [1] 765.0178
BIC(qc.fit6nonhom$fit)
## [1] 898.9617
例7:时间-事件分析和并行处理
qgcomp软件包利用Cox比例危害模型作为时间-事件分析的基础模型。qgcomp.noboot拟合参数的解释是一次性增加所有暴露的条件性(关于混杂因素)危险比。qc.survfit1对象展示了用qgcompcox.noboot进行的时间-事件分析。默认绘图与qgcompcox.noboot相似,因为它产生了权重和总体混合效应。
边际危害比率(以及一般的自举量化g计算)使用了一种略微不同的效果估计方法,这使得它比其他qcomp函数在计算上要求更高。为了估计边际危害比,先拟合基础模型,然后在基础模型下用基线危害估计器(Efron's)模拟新的结果--这种模拟需要一个大的样本(由MCsize控制)以保证准确性。这种方法类似于其他生存分析的g计算方法,但这种方法使用精确的生存时间,而不是大多数g计算分析中常见的离散生存时间。绘制qgcompcox.boot对象可以得到一组生存曲线(如qc.survfit2),它包括估计的生存曲线(假设在模型中的协变量的条件下,随机进行删减和晚期进入),这些生存曲线描述了不同水平的联合暴露的条件生存函数(即删减竞争风险)(包括总体平均值--它可能与观察到的生存曲线略有不同,但应该或多或少地一致)。
qgcomp中的所有自举函数都允许通过parallel=TRUE参数进行服饰化(在qc.survfit3中的非线性拟合中展示)。这里只使用了5次bootstrap迭代,这对于推理来说是远远不够的,而且由于设置并行进程时的一些开销,实际上并行处理的速度会更慢。
虽然qgcompcox.boot拟合了一个平滑的危险比函数,但是可以得到特定量值与参考量值的危险比,正如qc.survfit4所展示的那样。与qgcomp.boot图一样,条件模型拟合和MSM拟合重叠在一起,作为判断MSM与条件拟合程度的一种方式(以及是否应通过程度参数从整体拟合中添加或删除非线性项--我们在此注意到,我们不知道量化这些线条之间差异的统计测试,因此这取决于用户的判断,这些图作为视觉效果提供,以帮助进行探索性数据分析)。
# non-bootstrapped version estimates a marginal structural model for the
# confounder-conditional effect
survival::coxph(survival::Surv(disease_time, disease_state) ~ iron + lead + cadmium +
arsenic + magnesium + manganese + mercury +
selenium + silver + sodium + zinc +
mage35,
data=metals)
## Call:
## survival::coxph(formula = survival::Surv(disease_time, disease_state) ~
## iron + lead + cadmium + arsenic + magnesium + manganese +
## mercury + selenium + silver + sodium + zinc + mage35,
## data = metals)
##
## coef exp(coef) se(coef) z p
## iron -0.056447 0.945117 0.156178 -0.361 0.7178
## lead 0.440735 1.553849 0.203264 2.168 0.0301
## cadmium 0.023325 1.023599 0.105502 0.221 0.8250
## arsenic -0.003812 0.996195 0.088897 -0.043 0.9658
## magnesium 0.099399 1.104507 0.064730 1.536 0.1246
## manganese -0.014065 0.986033 0.064197 -0.219 0.8266
## mercury -0.060830 0.940983 0.072918 -0.834 0.4042
## selenium -0.231626 0.793243 0.173655 -1.334 0.1823
## silver 0.043169 1.044114 0.070291 0.614 0.5391
## sodium 0.057928 1.059638 0.063883 0.907 0.3645
## zinc 0.057169 1.058835 0.047875 1.194 0.2324
## mage35 -0.458696 0.632107 0.238370 -1.924 0.0543
##
## Likelihood ratio test=23.52 on 12 df, p=0.02364
## n= 452, number of events= 205
qc.survfit1 <- qgcomp.cox.noboot(survival::Surv(disease_time, disease_state) ~ .,expnms=Xnm,
data=metals[,c(Xnm, 'disease_time', 'disease_state')], q=4)
qc.survfit1
## Scaled effect size (positive direction, sum of positive coefficients = 0.32)
## barium zinc magnesium chromium silver sodium iron
## 0.3432 0.1946 0.1917 0.1119 0.0924 0.0511 0.0151
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.554)
## selenium copper calcium arsenic manganese cadmium lead mercury
## 0.2705 0.1826 0.1666 0.1085 0.0974 0.0794 0.0483 0.0466
##
## Mixture log(hazard ratio) (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## psi1 -0.23356 0.24535 -0.71444 0.24732 -0.9519 0.3411
plot(qc.survfit1)
# bootstrapped version estimates a marginal structural model for the population average effect
#library(survival)
qc.survfit2 <- qgcomp.cox.boot(Surv(disease_time, disease_state) ~ .,expnms=Xnm,
data=metals[,c(Xnm, 'disease_time', 'disease_state')], q=4,
B=5, MCsize=1000, parallel=TRUE)
qc.survfit2
## Mixture log(hazard ratio) (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## psi1 -0.23360 0.46321 -1.1415 0.67427 -0.5043 0.614
p2 = plot(qc.survfit2, suppressprint = TRUE)
p2 + labs(title="Linear log(hazard ratio), overall and exposure specific")
qc.survfit3 <- qgcomp.cox.boot(Surv(disease_time, disease_state) ~ . + .^2,expnms=Xnm,
data=metals[,c(Xnm, 'disease_time', 'disease_state')], q=4,
B=5, MCsize=1000, parallel=TRUE)
qc.survfit3
## Mixture log(hazard ratio) (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## psi1 -0.21122 0.35664 -0.91023 0.48779 -0.5922 0.5537
p3 = plot(qc.survfit3, suppressprint = TRUE)
p3 + labs(title="Non-linear log(hazard ratio) overall, linear exposure specific ln-HR")
qc.survfit4 <- qgcomp.cox.boot(Surv(disease_time, disease_state) ~ . + .^2,expnms=Xnm,
data=metals[,c(Xnm, 'disease_time', 'disease_state')], q=4,
B=5, MCsize=1000, parallel=TRUE, degree=2)
## Warning in fitter(X, Y, istrat, offset, init, control, weights = weights, :
## Loglik converged before variable 1,2 ; coefficient may be infinite.
qc.survfit4
## Mixture log(hazard ratio) (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI Z value Pr(>|z|)
## psi1 -2.86220 13.71090 -29.7351 24.0107 -0.2088 0.8346
## psi2 0.87417 4.64894 -8.2376 9.9859 0.1880 0.8508
# examining the overall hazard ratio as a function of overall exposure
hrs_q = exp(matrix(c(0,0,1,1,2,4,3,9), ncol=2, byrow=TRUE)%*%qc.survfit4$msmfit$coefficients)
colnames(hrs_q) = "Hazard ratio"
print("Hazard ratios by quartiles (min-25%,25-50%, 50-75%, 75%-max)")
## [1] "Hazard ratios by quartiles (min-25%,25-50%, 50-75%, 75%-max)"
hrs_q
## Hazard ratio
## [1,] 1.0000000
## [2,] 0.1369648
## [3,] 0.1077738
## [4,] 0.4872055
p4 = plot(qc.survfit4, suppressprint = TRUE)
p4 + labs(title="Non-linear log(hazard ratio), overall and exposure specific")




例8:聚类
个人或群体层面的聚类意味着存在个人或群体层面的特征,导致观察结果之间的协方差(例如,结果的个体内方差可能比个体间方差低得多)。对于线性模型,误差项被假定为观察值之间是独立的,而聚类则打破了这一假定。放宽这一假设的方法包括经验方差估计和适合聚类的稳健方差估计(例如通过R的sandwich包)。qgcomp.boot可以用来对暴露的独立效应以及暴露的整体效应进行聚类一致的标准误差估计。这是用qgcomp.boot的id参数完成的(该参数只能处理单一变量,因此对嵌套聚类可能并不有效)。
下面是一个简单的例子,有一个模拟的暴露。首先,暴露数据被 "预量化"(这样就可以用其他方法验证标准误差是否合适--这只是为了证明聚类的效果,而不是为了展示一个建议的做法)。接下来在qgcomp中使用单组分混合物对数据进行分析--同样,这也是为了验证。qgcomp.noboot的结果得到的混合物效应naive的标准误差为0.0310。
set.seed(2123)
N = 250
t = 4
dat <- data.frame(row.names = 1:(N*t))
dat <- within(dat, {
id = do.call("c", lapply(1:N, function(x) rep(x, t)))
u = do.call("c", lapply(1:N, function(x) rep(runif(1), t)))
x1 = rnorm(N, u)
y = rnorm(N) + u + x1
})
# pre-quantize
expnms = c("x1")
datl = quantize(dat, expnms = expnms)
qgcomp.noboot(y~ x1, data=datl$dat, family=gaussian(), q = NULL)
## Including all model terms as exposures of interest
## Scaled effect size (positive direction, sum of positive coefficients = 0.955)
## x1
## 1
##
## Scaled effect size (negative direction, sum of negative coefficients = 0)
## None
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.463243 0.057934 -0.57679 -0.34969 -7.996 3.553e-15
## psi1 0.955015 0.031020 0.89422 1.01581 30.787 < 2.2e-16
# neither of these ways yields appropriate clustering
#qgcomp.noboot(y~ x1, data=datl$dat, id="id", family=gaussian(), q = NULL)
#qgcomp.boot(y~ x1, data=datl$dat, family=gaussian(), q = NULL, MCsize=1000)
而qgcomp.boot结果(MCsize=5000,B=500)产生的修正标准误差为0.0398,比naive的估计值更接近sandwich估计值0.0409(为了显示,还包括了第二次qgcomp.boot拟合,其自举迭代次数更少,MCsize更小,但为了减少运行时间,更准确的结果被注释出来)。在这里,未修正的拟合的标准误差太低,但这可能并不总是这样的。
# clustering by specifying id parameter on
qgcomp.boot(y~ x1, data=datl$dat, id="id", family=gaussian(), q = NULL, MCsize=1000, B = 5)
## Including all model terms as exposures of interest
##
## Note: using all possible values of exposure as the
## intervention values
## Mixture slope parameters (bootstrap CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.463243 0.084446 -0.62875 -0.29773 -5.4856 5.223e-08
## psi1 0.955015 0.055037 0.84714 1.06289 17.3521 < 2.2e-16
#qgcomp.boot(y~ x1, data=datl$dat, id="id", family=gaussian(), q = NULL, MCsize=1000, B = 500)
# Mixture slope parameters (bootstrap CI):
#
# Estimate Std. Error Lower CI Upper CI t value
# (Intercept) -0.4632 0.0730 -0.606 -0.32 3.3e-10
# psi1 0.9550 0.0398 0.877 1.03 0
# This can be verified using the `sandwich` package
#fitglm = glm(y~x1, data=datl$dat)
#sw.cov = sandwich::vcovCL(fitglm, cluster=~id, type = "HC0")[2,2]
#sqrt(sw.cov)
# [1] 0.0409
例9:部分效果
回到我们原来的例子(并对协变量进行调整),注意qgcomp.*.noboot对象的默认输出包括 "正/负系数之和"。这些可以被解释为 "部分混合效应 "或具有特定方向系数的暴露的效应。这可以通过qgcomp的 "权重 "图来显示,所有对某一特定部分效应有贡献的暴露都在图的同一侧。不幸的是,这并不能产生真正的部分效应的有效推断,因为它是一个以拟合结果为条件的参数,因此并不代表适合于假设检验和置信区间的先验假设的类型。另一种思考方式是,它是一个数据适应性参数,因此会受到过度拟合问题的影响,这些问题与从阶梯式变量选择程序中进行推断的问题相似。
(qc.fit.adj <- qgcomp.noboot(y~.,dat=metals[,c(Xnm, covars, 'y')], expnms=Xnm, family=gaussian()))
## Scaled effect size (positive direction, sum of positive coefficients = 0.441)
## calcium barium iron arsenic silver chromium cadmium zinc
## 0.76111 0.06133 0.05854 0.02979 0.02433 0.02084 0.01395 0.01195
## mercury sodium
## 0.01130 0.00688
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.104)
## copper magnesium lead manganese selenium
## 0.44654 0.42124 0.09012 0.03577 0.00631
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.460408 0.134007 -0.72306 -0.19776 -3.4357 0.0006477
## psi1 0.337539 0.089358 0.16240 0.51268 3.7774 0.0001805
plot(qc.fit.adj)

幸运的是,有一种方法可以估计 "部分效应"。一种方法是样本分割,即首先将数据随机分割成一个 "训练 "数据集和一个 "验证 "数据集。对一个系数是否为正的评估发生在 "训练 "数据集中,然后在验证数据中对正/负的部分效应进行估计。基本的模拟可以表明,这种程序可以产生有效的(通常称为 "诚实的")假设检验和部分效应的置信区间,只要在组合数据集中没有单独的数据探索来选择模型。在qgcomp包中,将数据集划分为 "训练 "和 "验证 "集是由用户完成的,这就避免了在包含聚类(我们应该根据聚类而不是观测值进行划分)或每个个体的多个观测值(一个个体的所有观测值应该被划分在一起)的数据集上简单地进行划分可能出现的问题。下面是一个在每个个体包含一个观察值的数据集上进行简单分割的例子,这个数据集没有聚类。样本分割的缺点是精度的损失,因为最终的 "验证 "数据集只包括原始样本量的一小部分。因此,对部分效应的估计最适合于大样本量。我们还注意到,这些局部效应只有在所有效应都是线性和加性的情况下才能得到很好的定义,因为一个变量对局部效应的贡献是正还是负,取决于该变量的值,所以对 "局部效应 "的有效估计只限于使用qgcomp.\*.noboot对象进行推断的情况。
# 40/60% training/validation split
set.seed(1231124)
trainidx <- sample(1:nrow(metals), round(nrow(metals)*0.4))
valididx <- setdiff(1:nrow(metals),trainidx)
traindata <- metals[trainidx,]
validdata <- metals[valididx,]
dim(traindata) # 181 observations = 40% of total
## [1] 181 26
dim(validdata) # 271 observations = 60% of total
## [1] 271 26
然后,qgcomp软件包促进了对这些分区数据的分析,允许对部分效应进行有效的估计和假设检验。qgcomp.partials函数被用来估计部分效应。请注意,具有 "负效应大小 "的变量与代表我们对这些数据进行第一轮分析的qc.fit对象中的总体分析略有不同。这是可以预期的,也是这种方法的一个特点:数据的不同随机子集将产生不同的估计效应。如果真正的效应是空的,那么估计的效应将围绕空值从正到负变化,而样本分割是区分反映整个数据集潜在模式的估计值和仅仅由于小样本和中等样本固有的自然抽样变化的估计值的重要方法。
splitres <- qgcomp.partials(fun="qgcomp.noboot", f=y~., q=4,
traindata=traindata[,c(Xnm, covars, "y")],validdata=validdata[,c(Xnm, covars, "y")], expnms=Xnm)
splitres
##
## Variables with positive effect sizes in training data: calcium, mercury, iron, barium, cadmium, silver, chromium, manganese, zinc
## Variables with negative effect sizes in training data: copper, sodium, arsenic, selenium, magnesium, lead
## Partial effect sizes estimated in validation data
## Positive direction Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.416523 0.148975 -0.70851 -0.12454 -2.7959 0.005560
## psi1 0.314599 0.098888 0.12078 0.50842 3.1814 0.001643
##
## Negative direction Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) 0.072880 0.111731 -0.14611 0.29187 0.6523 0.5148
## psi1 -0.022265 0.073428 -0.16618 0.12165 -0.3032 0.7620
plot(splitres$pos.fit)

与我们的总体结果一致,金属对模拟结果y的总体影响是由主要由钙驱动的金属的正效应驱动的。psi=0.31的 "部分正效应与原始拟合中给出的[部分正效应(0.44)]相比有所减弱,但与原始拟合中的整体效应(psi1=0.34)相似,这反映出原始部分效应估计会因过度拟合而出现更多的极端值,但整体拟合不会对过度拟合那么敏感,在暴露效应都在同一方向的情况下有效地估计部分效应。我们注意到,铬、镉、汞、银和锰的效应方向是负的,尽管这些都是基于训练数据中的正相关而选择的。这表明这些变量的效应接近于空值,其方向取决于使用哪一个数据子集。这一特征允许对假设进行有效的测试--在没有暴露有效应的全局性空值中,变量的特征是在训练和验证数据集之间随机切换效应方向,这将产生接近空值的部分效应估计值,且P值很大。
需要注意的是:当存在多个具有小的正负效应的暴露时,在具有中等或小的样本量的研究中,部分效应可能会偏向于空值。这是因为在训练集中,一些具有小效应的暴露很可能在其效应方向方面被错误地分类。这意味着,小的负效应可以与大的正效应相加,产生较小的正效应,反之亦然。在某些情况下,积极和消极的部分效应可能是同一方向的。如果个别效果主要在一个方向上,但有些效果较小,可能有错误的分类方向,就会出现这种情况。部分正效应和部分负效应的总和仍将接近总体效应(其中的差异将是由于抽样误差造成的),但可能会有偏向部分效应的空值。
关于部分效应的解释(及其价值)的更大问题留给了分析者。对于具有良好特征的暴露的大型数据集,有可信的暴露子集与结果呈正/负线性相关,划分为负/正部分效应的变量可能有一些实质性意义。在更现实的环境中,即暴露混合物的类型,分区将导致不完全有意义的组。"部分效应 "产生的效果是,在训练数据中由正系数定义的子集中增加所有暴露,同时保持所有其他暴露和混杂因素不变。在与现实世界的相关模式相对应的情况下(例如,正的部分效应中的所有暴露都有一个来源),那么这可以大致解释为对这些暴露的来源进行干预的一种行动。然而,在大多数情况下,解释不会如此清晰,不应期待它映射到潜在的现实世界的干预措施。我们注意到,这不是量化g计算方法的功能,而只是处理暴露混合数据的一般混乱的一部分。
在将效应估计值映射到潜在的真实世界行动方面,一个更合理的方法是根据先前的主题知识选择暴露的子集,正如我们在上面的例子5中所展示的那样。这不需要分割样本,但它的代价是必须对分析中的暴露和结果有更多了解。例如,我们的模拟结果y可能代表了一些结果,我们期望在所谓的 "基本 "金属,或那些对于人类正常功能来说是必要的(少量)的金属中增加,但在 "潜在毒性"(非基本)金属,或那些没有已知的生物功能,更有可能造成伤害,而不是改善生理过程,导致y的改善(更大)值,它可能减少。
下面是必需金属元素的结果。
nonessentialXnm <- c(
'arsenic','barium','cadmium','chromium','lead','mercury','silver'
)
essentialXnm <- c(
'sodium','magnesium','calcium','manganese','iron','copper','zinc','selenium'
)
covars = c('nitrate','nitrite','sulfate','ph', 'total_alkalinity','total_hardness')
(qc.fit.essential <- qgcomp.noboot(y~.,dat=metals[,c(Xnm, covars, 'y')], expnms=essentialXnm, family=gaussian()))
## Scaled effect size (positive direction, sum of positive coefficients = 0.357)
## calcium iron zinc selenium
## 0.908914 0.077312 0.013128 0.000646
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.0998)
## copper magnesium sodium manganese
## 0.4872 0.4304 0.0486 0.0338
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.340130 0.111562 -0.55879 -0.12147 -3.0488 0.002435
## psi1 0.257136 0.074431 0.11125 0.40302 3.4547 0.000604
以下是非必需金属元素的结果:
(qc.fit.nonessential <- qgcomp.noboot(y~.,dat=metals[,c(Xnm, covars, 'y')], expnms=nonessentialXnm, family=gaussian()))
## Scaled effect size (positive direction, sum of positive coefficients = 0.0751)
## barium arsenic silver mercury cadmium
## 0.2827 0.2494 0.2420 0.1763 0.0496
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.0129)
## lead chromium
## 0.888 0.112
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.055549 0.081509 -0.215304 0.10420 -0.6815 0.4959
## psi1 0.062267 0.052508 -0.040648 0.16518 1.1858 0.2363
从这些结果中可以看出,必需金属和矿物质与结果表现出总体上的正联合关系(控制非必需品),而非必需金属和矿物质的部分效应接近于空。这与上面展示的部分效应的数据适应性选择的解释很接近,但从我们可能的干预方式(如增加必需金属和矿物质含量较高的食物的消费)来看,更容易解释。
缺失数据、LOD和多重填补
在使用quantile g-computation进行数据分析时,可以用与标准回归分析相同的方式处理缺失数据。一个常见的方法是完全案例分析。虽然R中的回归函数会在变量取值为NA(在R中表示缺失性)时自动进行完整案例分析,但在使用quantile g-computation时,鼓励人们明确地创建完整案例数据集并使用该完整案例数据集。使用预装的R软件包,这可以通过complete.cases函数来完成。
提出这一建议的原因是,虽然回归分析将在完整的病例上进行(即模型中所有变量的观测值都没有缺失),但每个暴露的定量计算是在逐个暴露的基础上进行的,这可能导致在明确使用仅限于完整病例的数据集与在分析过程中依靠自动删除有NA值的观测值之间出现数字差异。
下面是一个人为的例子,显示了这些差异。运行三个分析:一个是完整数据,一个是完整病例数据(完整病例分析#1),还有一个是砷被随机设置为NA的数据(完整病例分析#2)。
两个完整病例分析之间存在数字差异,这可以追溯到两种方法中 "量化 "暴露(除砷外)的差异,这可以通过qgcompfit对象中的qx数据框找到。
Xnm <- c(
'arsenic','barium','cadmium','calcium','chromium','copper',
'iron','lead','magnesium','manganese','mercury','selenium','silver',
'sodium','zinc'
)
covars = c('nitrate','nitrite','sulfate','ph', 'total_alkalinity','total_hardness')
asmiss = metals
set.seed(1232)
asmiss$arsenic = ifelse(runif(nrow(metals))>0.7, NA, asmiss$arsenic)
cc = asmiss[complete.cases(asmiss[,c(Xnm, covars, "y")]),] # complete.cases gives a logical index to subset rows
dim(metals) # [1] 452 26
## [1] 452 26
dim(cc) # [1] 320 26
## [1] 320 26
这里我们有完整数据的结果(用于比较)。
qc.base <- qgcomp.noboot(y~.,expnms=Xnm, dat=metals[,c(Xnm, covars, 'y')], family=gaussian())
cat("Full data\n")
## Full data
qc.base
## Scaled effect size (positive direction, sum of positive coefficients = 0.441)
## calcium barium iron arsenic silver chromium cadmium zinc
## 0.76111 0.06133 0.05854 0.02979 0.02433 0.02084 0.01395 0.01195
## mercury sodium
## 0.01130 0.00688
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.104)
## copper magnesium lead manganese selenium
## 0.44654 0.42124 0.09012 0.03577 0.00631
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.460408 0.134007 -0.72306 -0.19776 -3.4357 0.0006477
## psi1 0.337539 0.089358 0.16240 0.51268 3.7774 0.0001805
这里我们有一个完整的案例分析结果,在这个分析中,我们将一些暴露设定为缺失,并明确排除了将从分析中剔除的数据。
qc.cc <- qgcomp.noboot(y~.,expnms=Xnm, dat=cc[,c(Xnm, covars, 'y')], family=gaussian())
cat("Complete case analyses\n")
## Complete case analyses
cat(" #1 explicitly remove observations with missing values\n")
## #1 explicitly remove observations with missing values
qc.cc
## Scaled effect size (positive direction, sum of positive coefficients = 0.48)
## calcium iron zinc chromium silver lead barium arsenic
## 0.78603 0.07255 0.04853 0.03315 0.02555 0.01405 0.01285 0.00728
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.117)
## copper magnesium cadmium mercury manganese sodium selenium
## 0.52200 0.23808 0.09082 0.06570 0.04105 0.03921 0.00315
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.509402 0.145471 -0.79452 -0.22428 -3.5017 0.0005315
## psi1 0.362582 0.096696 0.17306 0.55210 3.7497 0.0002119
最后,我们有一个完整的案例分析结果,在这个案例中,我们把一些暴露值设置为缺失,但我们依靠R的自动放弃缺失值的观察。
qc.cc2 <- qgcomp.noboot(y~.,expnms=Xnm, dat=asmiss[,c(Xnm, covars, 'y')], family=gaussian())
cat(" #1 rely on R handling of NA values\n")
## #1 rely on R handling of NA values
qc.cc2
## Scaled effect size (positive direction, sum of positive coefficients = 0.493)
## calcium iron zinc chromium barium silver lead selenium
## 0.76495 0.07191 0.04852 0.03489 0.02151 0.02119 0.01912 0.01444
## arsenic
## 0.00348
##
## Scaled effect size (negative direction, sum of negative coefficients = -0.12)
## copper magnesium sodium manganese cadmium mercury
## 0.5338 0.2139 0.0965 0.0853 0.0501 0.0204
##
## Mixture slope parameters (Delta method CI):
##
## Estimate Std. Error Lower CI Upper CI t value Pr(>|t|)
## (Intercept) -0.51673 0.15001 -0.81074 -0.22273 -3.4447 0.0006520
## psi1 0.37249 0.10014 0.17621 0.56876 3.7196 0.0002376
现在我们可以看到上述方法之间存在差异的原因:当依靠R允许分析数据中暴露的缺失值而放弃缺失值时,暴露的定量将在每个暴露的所有有效值上进行。在完整的病例数据中,将只在有完整观测值的人中计算量值。后者通常是首选,因为每个暴露的量值将在相同的个人样本中计算。
# calculation of arsenic quantiles is identical
all.equal(qc.cc$qx$arsenic_q, qc.cc2$qx$arsenic_q[complete.cases(qc.cc2$qx$arsenic_q)])
## [1] TRUE
# all are equal
all.equal(qc.cc$qx$cadmium_q, qc.cc2$qx$cadmium_q[complete.cases(qc.cc2$qx$arsenic_q)])
## [1] "Mean relative difference: 0.3823529"
# not equal
LOD
混合物中常见的缺失数据形式是由于低于检测极限而缺失的暴露值。处理这类缺失数据的常见方法是估算,要么通过填入小的数字值来代替缺失值,要么通过更正式的参数模型进行多重估算。值得注意的是,在量化g计算中,如果低于检测极限的数值比例低于1/q(量化数),所有适当的缺失数据方法都会得到相同的答案,因为这些数值都是已知的最低类别。因此,如果有3次曝光,每次都有10%的值低于检测限,我们可以估算出低于这些限值的小值(常用的方法是检测限除以2的平方根),然后对估算出的数据进行量子g计算。这种分析利用了这样一个事实,即即使检测极限以下的数值不能确定地知道,在量化g计算中使用的类别分数也是确定的。在有超过1/q%的测量值低于检测限的情况下,软件包qgcomp带有一个方便的函数mice.impute.leftcenslognorm,可用于从左删减的对数正态回归模型中归纳检测限以下的数值。
多重填补
多重归因法使用多个数据集,对各数据集的每个缺失值进行不同的归因。对这些数据集中的每一个进行单独的分析,并使用标准规则对结果进行合并。函数mice.impute.leftcenslognorm可以与mice包对接,以便通过量化g计算对基于多重归因的分析进行有效编程。在没有明确安装mice包的情况下,这里不能包括例子,但是可以在mice.impute.leftcenslognorm的帮助文件中看到一个例子。
FAQ
为什么没有从boot方程中得到权重
用户经常使用qgcomp.*.boot函数,因为他们想对混杂因素进行边际化处理,或拟合非线性的联合暴露函数。在这两种情况下,总体暴露反应将不再对应于模型系数的简单加权平均,所以qgcomp.*.boot函数都不会计算权重。在大多数情况下,权重会根据你所处的联合暴露水平而变化,所以计算权重不是一个简单的命题(如果你使用默认的q=4,你可能不希望报告4套权重)。如果你拟合了一个其他的线性模型,你可以从qgcomp.*.noboot中得到权重,这将非常接近于你通过qgcomp.*.boot函数从线性模型拟合得到的权重,但是要明确,这些权重来自于一个不同的模型,而不是关于联合暴露效应的推断。
我是否需要对暴露的非线性和非加性进行建模?
也许吧。qgcomp的推断对象是对应于联合暴露反应的ψ参数集。事实证明,对于相关暴露,非线性可以伪装成非加性(Belzak and Bauer [2019] Addictive Behaviors)。如果我们推断的是独立效应,那么这种区别将是至关重要的,但如果我们只希望准确预测联合暴露水平的结果,那么可能会发现,如果你通过非加性或特定研究中单个暴露的非线性来模拟联合反应函数的非线性,这并不重要。在qgcomp中拟合的模型仍然有一个关键的假设,即你能够通过参数模型对联合暴露反应进行建模,所以在试图从非可加性(如暴露的二次项)中剔除非线性(如暴露之间的乘积项)时,不应忘记这个假设。需要注意的是参数化建模的重要部分是,我们必须明确告诉模型是非线性的,没有适应非线性的设置会自动发生。探索非线性并不是一个微不足道的努力。
必须使用分位数吗
你可以通过设置q=NULL来关闭 "quantization",或者通过 "break "参数提供你自己的分类切点。如果采用这两个选项中的任何一个,都要由用户来解释结果。
References
Keil, A.P., Buckley, J.P., O’Brien, K.M., Ferguson, K.K., Zhao, S. and White, A.J., 2020. A quantile-based g-computation approach to addressing the effects of exposure mixtures. Environmental health perspectives, 128(4), p.047004.
更多推荐
 已为社区贡献15条内容
已为社区贡献15条内容








所有评论(0)