MRI脑影像分析——多种工具实现Nifti(*.nii)文件读取、处理与写入——把小舞写进脑海里、6mm半高全宽高斯核平滑脑影像、NIFTI文件合并、算fMRI平均图像
Nifti文件格式,是目前各大神经影像分析工具普遍兼容的体素水平的数据格式,也是在进行神经影像研究中最常见的一种数据格式。Matlab从2017b后就引入了专门的Nifti文件的解析函数,SPM12,dpabi等工具包也提供了Nifti文件的解析接口,NIfTI_20140122是专门的Nifti文件的解析处理工具包。python上面的nibabel可以解析Nifti等脑影像文件,nilearn更

| 图源
Nifti(Neuroimaging Informatics Technology Initiative,神经影像信息学技术倡议)文件格式,是目前各大神经影像分析工具普遍兼容的体素水平的数据格式,也是在进行神经影像研究中最常见的一种数据格式。简单点看它,它就是一个三维数组(sMRI)或者四维数组(fMRI、dMRI),再套上一个头部数据。数组里包含的就是图像体素值数据本身,头部数据里包含空间和体素信息,具体的文件结构不必理会,只要能用工具解析它即可。Matlab从2017b后就引入了专门的Nifti文件的解析函数1,SPM12(Statistical Parametric Mapping,统计参数映射),dpabi等工具包也提供了Nifti文件的解析接口,NIfTI_20140122是专门的Nifti文件的解析处理工具包,现在好像没有更新了,不过也比较好用。python上面的nibabel可以解析Nifti等脑影像文件,nilearn更是一个集解析,处理,与分析于一体的工具。以下就针对这些工具,简要探讨他们对Nifti文件的解析,以及简单处理。
copyright©意疏:https://blog.csdn.net/sinat_35907936/article/details/118862614
SPM12读取Nifti文件——查看轴状面、冠状面与矢状面切片
SPM12提供spm_vol(N)来获取Nii文件的头部信息,返回一个带有图像信息的结构体。N可以是单个Nii文件的路径,也可以是包含多个Nii文件路径的数组。
V = spm_vol('brain.nii');
返回的结构体包含一个完整的Nifti-1的对象,和被SPM12简化了的Nifti-1属性,如图1所示,因为T1是三维影像,所以返回的1X1的结构体,如果是fMRI则会返回nX1的结构体。属性中比较重要的是,路径属性fname和维度属性dim。

SPM12用spm_read_vols()来解析头部信息结构体,并返回一个三维或者四维数组Y,就是图像数据体素值本身,另外还有一个3xn的数组是每个体素的坐标。
[Y,XYZmm] = spm_read_vols(V);
size(Y);
轴状位、冠状位与矢状位是医学上人体的三个方位。左右为冠(X),前后为矢(Y),上下为轴(Z)。沿矢状位,切分左右的面为矢状面(sagital,固定X);沿冠状位,切分前后的面为冠状面(coronal,固定Y);切分上下的为轴状面(横断面,axial,固定Z),如图2所示,图源。

有了上面的分析,我们只需要在三维数据中,固定对应的轴,就可以取出对应的二维切面来。代码如下,其间为了最终显示还涉及如维度压缩squeeze,维度交换permute,尺度变换mapminmax等操作。
代码:
nifti_file = spm_vol('MNI152_T1_1mm.nii');
data = spm_read_vols(nifti_file);
% 水平面
Z_66 = squeeze(data(:,:,66));
Z_66 = permute(Z_66,[2,1,3]);
figure(1);
imagesc(mapminmax(Z_66, 0, 255));
set(gca, 'YDir', 'normal');
colormap(gray);
axis off;
% 冠状面
Y_111 = squeeze(data(:,111,:));
Y_111 = permute(Y_111,[2,1]);
figure(2);
imagesc(mapminmax(Y_111, 0, 255));
set(gca, 'YDir', 'normal');
colormap(gray);
axis off;
% 矢状面
X_99 = squeeze(data(99,:,:));
X_99 = permute(X_99,[2,1]);
figure(3);
imagesc(mapminmax(X_99, 0, 255));
set(gca, 'YDir', 'normal');
colormap(gray);
axis off;
结果:

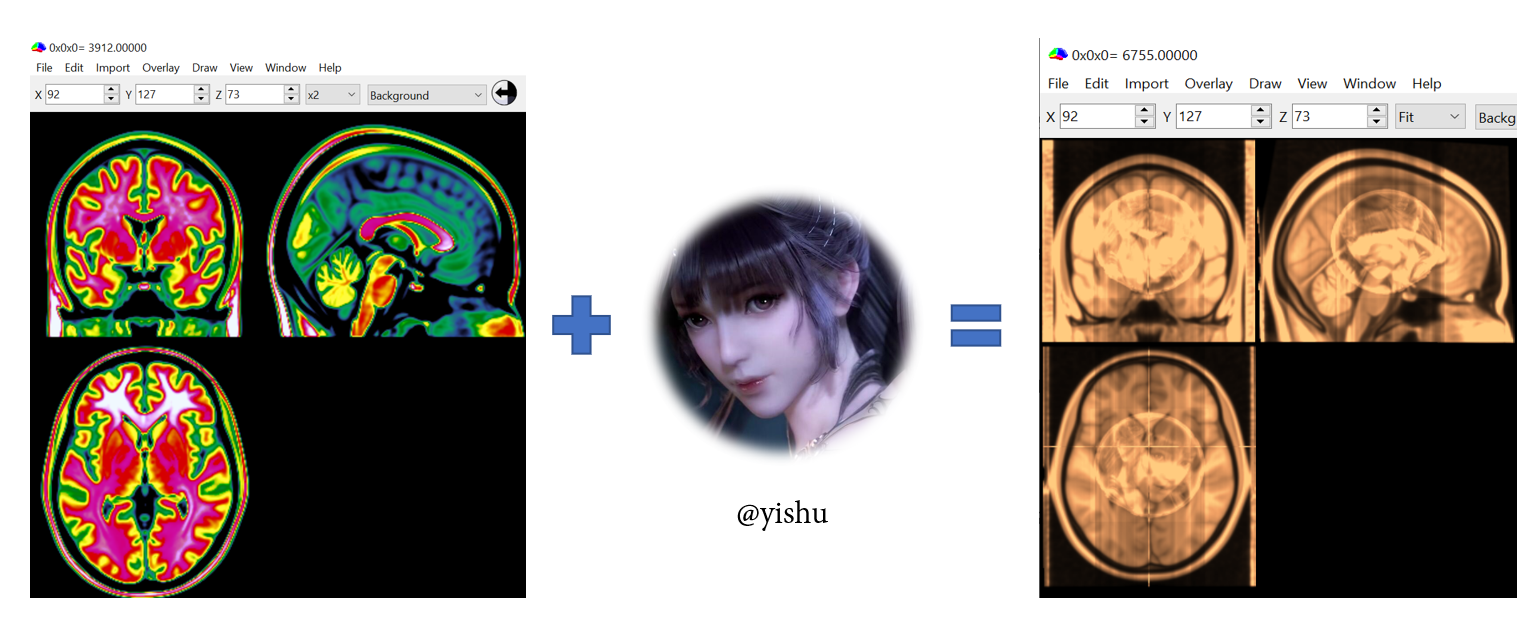
SPM12写入Nifti文件——把小舞放进脑海里
能取出数据,就表明我们有操作数据的能力了。可要要对数据进行什么样的操作呢,或许可以把女神放进“脑海里”。比如笔者就找了张斗罗大陆小舞的照片,并把它嵌入到了三个切面里,使得每次打开nii文件就可以直接看到她。如果用mricron打开MNI152_T1_1mm.nii,会发现默认的X Y Z为92 127 73,估计这是坐标原点的位置,以这三个值作为三个切片固定轴的坐标,取出平面嵌入图片并保存之后,每次用mricron打开就可以直接看到嵌入的图片。
SPM12用spm_write_vol()来保存新的数据,参数是头部信息结构体和数据数组。这里除了动了一些值以外,并没有改变原图像的空间体素信息,所以直接用原来的头即可,只是需要重命名一下,避免覆盖原始文件。
spm_write_vol(V,Y);
V.fname = 'newname.nii';
代码:
% 加载待嵌入100x100的图片
I = imresize(flipud(imread('xiaowu.jpg')), [100,100]);
I = double(rgb2gray(I));
figure(1)
imagesc(I)
colormap(gray);
% 加载nii
nifti_file = spm_vol('MNI152_T1_1mm.nii');
data = spm_read_vols(nifti_file);
% 水平面
Z_73 = squeeze(data(:,:,73));
Z_73_pic = mapminmax(Z_73, 0, 255);
Z_73_pic = permute(Z_73_pic,[2,1]);
Z_73_pic(61:160, 41:140) = 0.5*I + 0.5*Z_73_pic(61:160, 41:140); % 按照比例融合切面与原数据
figure(2);
imagesc(Z_73_pic);
set(gca, 'YDir', 'normal');
colormap(gray);
axis off;
% 冠状面
Y_127 = squeeze(data(:,127,:));
Y_127_pic = mapminmax(Y_127, 0, 255);
Y_127_pic = permute(Y_127_pic,[2,1]);
Y_127_pic(41:140, 41:140) = 0.5*I + 0.5*Y_127_pic(41:140, 41:140);
figure(3);
imagesc(Y_127_pic);
set(gca, 'YDir', 'normal');
colormap(gray);
axis off;
% 矢状面
X_91 = squeeze(data(91,:,:));
X_91_pic = mapminmax(X_91, 0, 255);
X_91_pic = permute(X_91_pic,[2,1]);
X_91_pic(41:140, 61:160) = 0.5*I + 0.5*X_91_pic(41:140, 61:160);
figure(4);
imagesc(mapminmax(X_91_pic, 0, 255));
set(gca, 'YDir', 'normal');
colormap(gray);
axis off;
Z_73_pic = permute(Z_73_pic,[2,1]);
Y_127_pic = permute(Y_127_pic,[2,1]);
X_91_pic = permute(X_91_pic,[2,1]);
% 将融合的数据缩放并覆盖原数据
data(:,:,73) = mapminmax(Z_73_pic, min(min(data(:,:,73))), max(max(data(:,:,73))));
data(:,127,:) = mapminmax(Y_127_pic, min(min(data(:,127,:))), max(max(data(:,127,:))));
tmp(1,:,:)= mapminmax(X_91_pic, min(min(min(data(92,:,:)))), max(max(max(data(92,:,:)))));
data(91,:,:) =tmp;
% 保存nii文件
nifti_file.fname = 'xiaowu.nii';
spm_write_vol(nifti_file,data);
结果:

copyright©意疏:https://blog.csdn.net/sinat_35907936/article/details/118862614
Matlab自带函数读写NIFTI文件2——用6mm半高全宽高斯核平滑脑影像
读取NIFTI信息。可以通过info.ImageSize获取boundingbox大小,通过info.PixelDimensions获取体素尺寸。
info = niftiinfo('brain.nii')
三种方式读取NIFTI图像数据,返回N维数组。
V = niftiread(filename)
V = niftiread(headerfile,imgfile)
V = niftiread(info)
写入NIFTI图像数据
% 使用默认的头部信息,filename新文件名
niftiwrite(V,filename)
% 使用info头部信息
niftiwrite(V,filename,info)
例子:用半高全宽FWHM为6mm的高斯核平滑脑影像,对于高斯分布:FWHM = 2 × sqrt(2ln2) × σ ≈ 2.355 σ ,故6mm半高全宽对应2.55mm的标准差。
V = niftiread('MNI152_T1_1mm.nii');
info = niftiinfo('MNI152_T1_1mm.nii');
info.Description = 'smoothed with 6mm FWHM Gaussian kernel'
SV=imgaussfilt3(V,2.55);
niftiwrite(SV,'sMNI152_T1_1mm.nii',info);
效果

NIfTI_20140122读写NIFTI文件——用6mm半高全宽高斯核平滑脑影像
笔者比较喜欢用NIfTI_20140122来读写NIFTI文件。因为它把文件的头部信息和图像数组同时包含在一个同一个结构体中,用起来比较方便。直接通过file.img得到图像数组,通过file.hdr.dime.dim和file.hdr.dime.pixdim获取boundingbox大小与体素尺寸。
file = load_nii('brain.nii');
save_nii(file, 'newname.nii');

例子:用半高全宽FWHM为6mm的高斯核平滑脑影像
file = load_nii('MNI152_T1_1mm.nii');
file.img = imgaussfilt3(file.img,2.55);
save_nii(file, 'sMNI152_T1_1mm.nii');
Dpabi读写NIFTI文件——NIFTI文件合并
Dpabi的y_ReadAll函数比较强大,不仅可以读取NIFTI文件,还能读取GIFTI格式与普通的mat文件,不仅可以读单个文件,还能同时读取一个文件夹里的文件,并通过增加一维的方式来存放文件夹中每一个NIFTI的数据,这可以用于NIFTI数据的合并。这里只用来读NIFTI文件,通过读取文件夹的方式,并合并数据。
[Data, VoxelSize, FileList, Header] = y_ReadAll(InputName);
y_Write(Data,Header,OutName);
例子:NIFTI文件合并
[Data, VoxelSize, FileList, Header] = y_ReadAll(test);
y_Write(Data,Header,'cMNI152_T1_1mm.nii');
效果:文件夹中两个NIFTI文件,被合并成了一个NIFTI文件。

copyright©意疏:https://blog.csdn.net/sinat_35907936/article/details/118862614
Nibabel读写NIFTI文件3——通过阈值分割图像
Nibabel是python上用于解析神经影像的工具包,除了读写NIFTI外,还有其他非常丰富的接口,具体见官网。nib.load()返回的是一个nibabel图像对象实例,包含头部信息和数据数组,如下图。Nibabel有两种方式来保存NIFTI文件。通过img.get_fdata(),img.header.get_data_shape(),img.header.get_zooms()方法,可以获取数据,boundingbox大小与体素尺寸。
img = nib.load('brain.nii')
% 两种保存方式
nib.save(img, 'newname.nii')
img.to_filename('newname.nii')

例子:通过阈值分割图像。get_fdata()方法取出来的数组是numpy数组,修改之后依旧是numpy数组。如果要使用to_filename或者save保存修改的数组为NIFTI数据,需要用nibabel的NIFTI工具,加上头和仿射矩阵4。
import cv2
import nibabel as nib
img = nib.load('MNI152_T1_1mm.nii')
new_data = img.get_fdata().copy()
new_data[new_data<6000]=0
new_img = nib.Nifti1Image(new_data, img.affine, header=img.header)
new_img.to_filename('tMNI152_T1_1mm.nii')
效果:

Nilearn读写NIFTI文件5——算fMRI平均图像
Nilearn是做脑影像分析非常重要的一个python包,它不仅包括NIFTI的解析,还包括统计和机器学习工具。它通过image模块来读取数据,返回的是一个nibabel图像对象实例,然后用to_filename来保存数据。
from nilearn import image
img = image.load_img('rest.nii')
print(img.shape)
img = image.mean_img(img)
print(img.shape)
img.to_filename('mean_rest.nii')
效果:用image自带的处理方法处理,是携带着头文件一起的,所以处理完成后的数据依旧是NIFTI格式,可以直接保存。

copyright©意疏:https://blog.csdn.net/sinat_35907936/article/details/118862614
参考
https://ww2.mathworks.cn/help/images/ref/niftiwrite.html. ↩︎
https://ww2.mathworks.cn/help/images/ref/niftiread.html?searchHighlight=nifti&s_tid=srchtitle. ↩︎
https://nipy.org/nibabel/nibabel_images.html ↩︎
http://cn.voidcc.com/question/p-ajddryeu-tp.html ↩︎
http://nilearn.github.io/modules/reference.html#module-nilearn.image ↩︎
更多推荐
 已为社区贡献5条内容
已为社区贡献5条内容










所有评论(0)