
简介
该用户还未填写简介
擅长的技术栈
可提供的服务
暂无可提供的服务
我们已经预处理了一个由 400 个大脑组成的重要亚组,分为训练(300)、验证(50)和测试(50)。我们使用几个软件包(FreeSurfer 7.1 和 SAMSEG)计算了包含 40 个皮质和皮质下解剖标签的分割图。我们将融合这些标签图,并在 MGH 神经科学家的帮助下手动验证所有 400 次扫描。

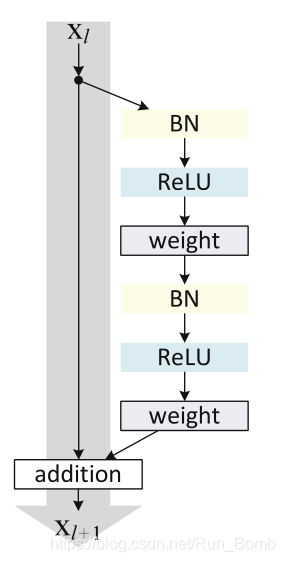
???? 参考文档:《百度飞浆:零基础入门深度学习》假设输入图片尺寸是 H×WH \times WH×W,卷积核(池化窗口)大小为 kh×kwk_h \times k_wkh×kw卷积层: 假设填充为(ph,pw)(p_h, p_w)(ph,pw),步幅为 (sh,sw)(s_h, s_w)(sh,sw),则卷积层输出特征图的大小为:Hout=H+2ph−khsh+1H_{out} =
来自 B 站刘二大人的《PyTorch深度学习实践》P11 的学习笔记GoogLeNet1×1 卷积上一篇我们知道,卷积的个数取决于输入图像的通道数。1×1 卷积能起到特征融合、改变通道数和减少计算量的效果,被称为神经网络中的神经网络例如,我们先通过 1×1 卷积减少了通道的数量,让大的卷积核计算更少的通道数,能大大减少计算量:Inception ModuleInception(盗梦空间) Mod
Deep Learning-Based Concurrent Brain Registration and Tumor SegmentationEstienne, T. et al. “Deep Learning-Based Concurrent Brain Registration and Tumor Segmentation.” Frontiers in Computational Neuro
1.5b 模型,4GB显存就能跑。7b、8b 模型,8GB显存就能跑。14b 模型,12GB显存能跑。32b 模型,24GB显存能跑。提供 Web UI,需要 Ollama 在后台运行。Ollama提供本地模型运行服务。

1.5b 模型,4GB显存就能跑。7b、8b 模型,8GB显存就能跑。14b 模型,12GB显存能跑。32b 模型,24GB显存能跑。提供 Web UI,需要 Ollama 在后台运行。Ollama提供本地模型运行服务。

划词翻译插件设置自定义 ollama 翻译源

Deep Learning-Based Concurrent Brain Registration and Tumor SegmentationEstienne, T. et al. “Deep Learning-Based Concurrent Brain Registration and Tumor Segmentation.” Frontiers in Computational Neuro
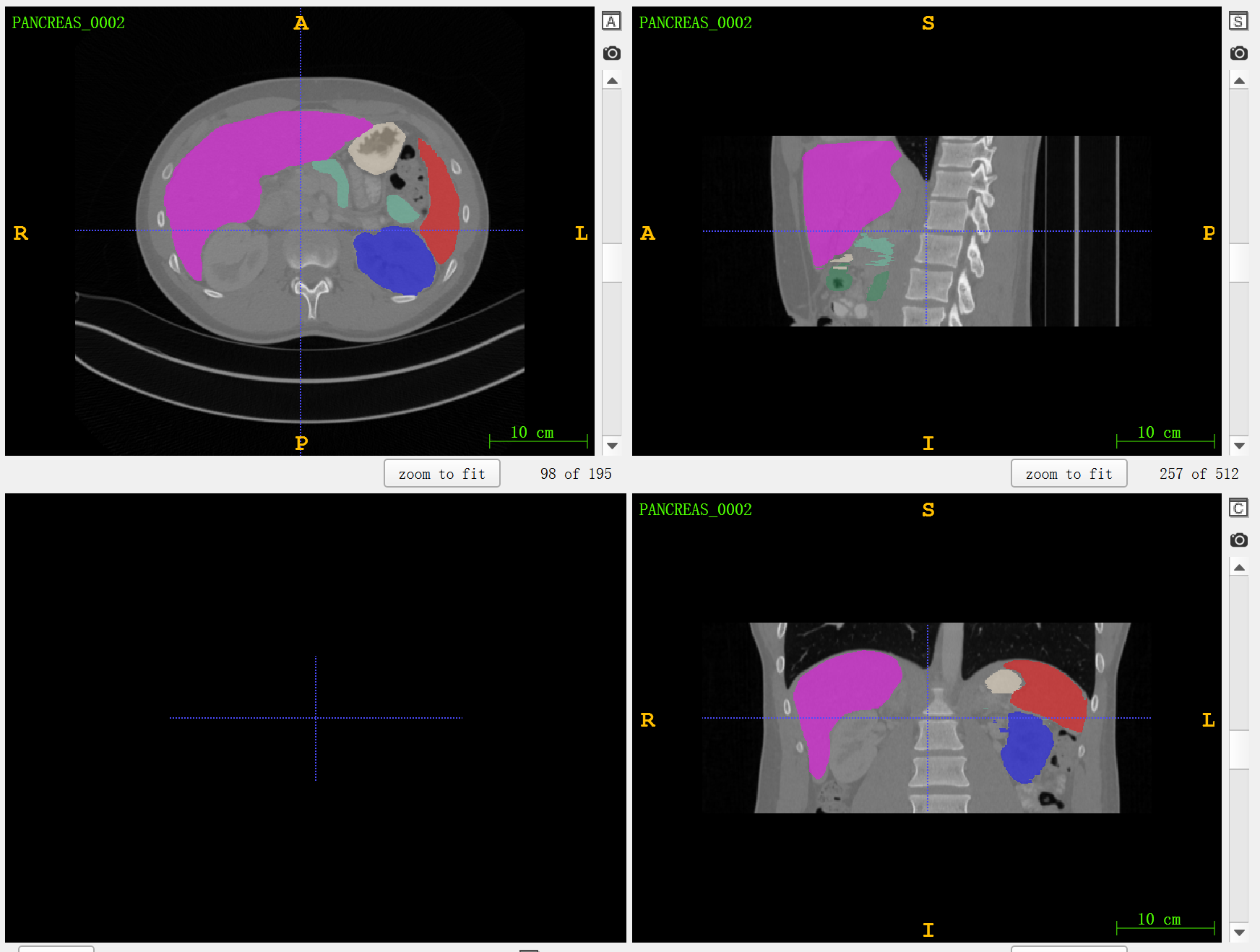
❤️❤️❤️ c3d 是一个免费的、强大到无以复加的 3D 图像处理工具,它尤其在深度学习医学图像研究和应用中能发挥出无与伦比的生产力 —— 我 ????我将以 Pancreas-CT 数据集及其标签为例,简要介绍 c3d 处理 dicom 格式 3D 图像(dicom 转成 nii)、Nifti 格式 3D 图像的常用功能下载安装 c3d 和 ITK-SNAP官网下载地址:http://www.
转载自:【论文笔记】VoxelMorph-无监督医学图像配准模型本文是无监督的医学图像配准模型——VoxelMorph 的论文笔记。变形配准(Deformable registration)策略通常包括两步:第一步是可实现全局对齐的仿射变形,然后是具有自由度更高的更缓慢变形变换。本文主要是关心第二步。一、记号fff 和 mmm 分别表示 fixed image 和 moving image,ϕ\p










