实战:使用yolov3完成肺结节检测(Luna16数据集)及肺实质分割
实战:使用yolov3完成肺结节检测(Luna16数据集)yolov3是一个比较常用的端到端的目标检测深度学习模型,这里加以应用,实现肺结节检测。由于Luna16数据集是三维的,需要对其进行切片操作,转换成yolov3可以处理的二维图片。1. yolov3代码及原理* 代码* 原理2. Luna16数据集* 数据集介绍* 转换成voc数据集格式3. 检测效果预览更新:2019....
实战:使用yolov3完成肺结节检测(Luna16数据集)
yolov3是一个比较常用的端到端的目标检测深度学习模型,这里加以应用,实现肺结节检测。由于Luna16数据集是三维的,需要对其进行切片操作,转换成yolov3可以处理的二维图片。
1. yolov3代码及原理
* 代码
* 原理
2. Luna16数据集
* 数据集介绍
* 转换成voc数据集格式
3. 检测效果预览
更新:2019.9.10
对数据集进行肺实质分割。
更新:2019.9.14
使用分割后的数据进行训练
更新:2019.9.23
修改anchor size后重新训练
更新:2019.12.5
发现一个注释的错误:
a = image_array.transpose(1,2,0)[:,:,20] #transpose是将(z,x,y)的三维矩阵转为(x,y,z)的矩阵应该是transpose是将(z,y,x)的三维矩阵转为(y,x,z)的矩阵
更新:2019.12.11
发现应该坐标换算错误:
#如果图像进行旋转了,那么坐标也要旋转
if isflip :
x_max = 512 - x_min#修改前是 x_min = 512 - x_min 以下类似
y_max = 512 - y_min
x_min = 512 - x_max
y_min = 512 - y_max
x = 512 - x
y = 512 - y补充 2020.11.19
博主临近毕业,可能有消息无法及时回复,见谅,有问题请留言。
有很多csdner向我要数据集和代码,关于代码核心部分已经在博客中讲解(已上传csdn,存在些许bug),如果要提高自己能力,建议看懂,自己上手。如果是想要快速上手,测试相关数据集,我已经上传百度云VOC数据集,密码:zc0p。
赠人玫瑰,手留余香,如果我的博文/数据集/代码对你有所帮助,麻烦点个赞~~
补充:关于为什么分享数据集size不是512*512的,这里我对原始CT是进行了归一化的,即1mm*1mm*1mm,所以尺寸不一样。
1. yolov3代码及原理
1.1代码
基于c的:
https://github.com/AlexeyAB/darknet
基于pytorch的:
https://github.com/eriklindernoren/PyTorch-YOLOv3
1.2原理
可查看:
Pytorch | yolov3原理及代码详解(一)
Pytorch | yolov3原理及代码详解(一)_NotFound1911的博客-CSDN博客_pytorch和yolo的关系
Pytorch | yolov3原理及代码详解(二)
Pytorch | yolov3原理及代码详解(二)_NotFound1911的博客-CSDN博客_yolov3pytorch代码详解
Pytorch | yolov3原理及代码详解(三)
Pytorch | yolov3原理及代码详解(三)_NotFound1911的博客-CSDN博客_yolov3pytorch代码详解
Pytorch | yolov3原理及代码详解(四)
Pytorch | yolov3原理及代码详解(四)_NotFound1911的博客-CSDN博客
2.Luna16数据集
2.1 Luna16数据集介绍
LUNA16数据集介绍_pursuit_zhangyu的博客-CSDN博客_luna16
Luna16数据集_一一|一一一亅的博客-CSDN博客_luna16数据集
上面说的很全,这里只进行补充:
2.1.1.图像的翻转
.mhd文件中有CT图像的属性,其中有一条是:
TransformMatrix = 1 0 0 0 1 0 0 0 1- TransformMatric:图像矩阵是否翻转的标志。(在现实中CT扫描的时候有的人是正卧,有的人是仰卧的,所以会导致图像会出现翻转的情况。)
- TransformMatrix = 1 0 0 0 1 0 0 0 1是正卧的,这里举一个反卧的例子:TransformMatrix = -1 0 0 0 -1 0 0 0 1。当图像反卧的时候我们把图像的x,y坐标进行倒序调整,使其正卧。如下面代码所示(加载CT图像):
def load_itk_image(filename):
with open(filename) as f:
contents = f.readlines()
line = [k for k in contents if k.startswith('TransformMatrix')][0]
offset = [k for k in contents if k.startswith('Offset')][0]
EleSpacing = [k for k in contents if k.startswith('ElementSpacing')][0]
# 把值进行分割
offArr = np.array(offset.split(' = ')[1].split(' ')).astype('float')
eleArr = np.array(EleSpacing.split(' = ')[1].split(' ')).astype('float')
CT = CTImage(offArr[0],offArr[1],offArr[2],eleArr[0],eleArr[1],eleArr[2])
transform = np.array(line.split(' = ')[1].split(' ')).astype('float')
transform = np.round(transform)#round() 方法返回浮点数x的四舍五入值
if np.any(transform != np.array([1, 0, 0, 0, 1, 0, 0, 0, 1])):#判断是否相等
isflip = True
else:
isflip = False
itkimage = sitk.ReadImage(filename)
numpyimage = sitk.GetArrayFromImage(itkimage)
if(isflip == True):
numpyimage = numpyimage[:,::-1,::-1] #::-1 倒序
return (numpyimage, CT, isflip)因此,如果图像要翻转,那么坐标也要进行处理(512是CT的宽和高)(2019.12.11进行修正)(2019.12.25进行修正):
#如果图像进行旋转了,那么坐标也要旋转
if isflip :
x_max1 = 512 - x_min
y_max1 = 512 - y_min
x_min1 = 512 - x_max
y_min1 = 512 - y_max
x_max = x_max1
y_max = y_max1
x_min = x_min1
y_min = y_min1
x = 512 - x
y = 512 - y2.1.2图像的预处理
正如上面博客所说的,CT图像的像素值是 [-1000,+ 400],所以我们进行了预处理,对像素值进行截断处理。同时由于CT图像是单通道图片,如果直接截断保存单张图片,是不行的,必须要先转换成RGB格式,在进行保存,否则保存下来就是一张全黑的图片。
def truncate_hu(image_array):
image_array[image_array > 400] = 400
image_array[image_array <-1000] = -1000
def normalazation(image_array):
max = image_array.max()
min = image_array.min()
#归一化
image_array = (image_array - min)/(max - min)*255
#image_array = image_array.astype(int)#整型
image_array = np.round(image_array)
return image_array #这个图的x,y坐标是反的,在生成标注的时候需要注意。
case_path = 'data/subset3/1.3.6.1.4.1.14519.5.2.1.6279.6001.123697637451437522065941162930.mhd'
numpyimage, _, _ = load_itk_image(case_path)
truncate_hu(numpyimage)
image_array = normalazation(numpyimage)
#3维图像切割
a = image_array.transpose(1,2,0)[:,:,20] #transpose是将(z,y,x)的三维矩阵转为(y,x,z)的矩阵
#转换成RGB类型并保存
im = Image.fromarray(a)
im = im.convert("RGB")
im.save("error/test1.png")2.2 Luna16数据处理成VOC格式
2.2.1 遍历Luna16的标注文件
Luna16的所有标注均在annotations.csv文件中。这个标注的含义上述博客有讲解,这里遍历标注文件并转换成相应的x,y,w,h标注时需要注意两点:
1.同一个CT图可能具有多个肺结节。

所以在遍历文件的时候,一定要把一个CT图中所有的肺结节遍历完,才能遍历下一站CT图。这里提供一个遍历文件的代码:
#查询文件 返回路径 -1表示无
def search(path=".", name="",fileDir = []):
for item in os.listdir(path):
item_path = os.path.join(path, item)
if os.path.isdir(item_path):
search(item_path, name)
elif os.path.isfile(item_path):
if name in item:
fileDir.append(item_path)
#print("fileDir:",fileDir)
return fileDir2.坐标换算
上述博客中也提到的了,要完成世界坐标到图像坐标的换算。
#世界坐标转换到图像中的坐标
def worldToVoxelCoord(worldCoord, offset, EleSpacing):
stretchedVoxelCoord = np.absolute(worldCoord - offset)
voxelCoord = stretchedVoxelCoord / EleSpacing
return voxelCoord肺结节的x,y,z的换算不难理解,补充的是w和h的换算。w是在x轴方向上,h是在y轴方向上,换算方式类似,但注意w和h是长度单位,所有不会有偏置(坐标原点)。
w = np.round(r/CT.x_ElementSpacing).astype(int)
h = np.round(r/CT.y_ElementSpacing).astype(int)这里的CT是我建的一个类,目的是方便CT各项数据的传递。
#定义CT图像类保存 中心点
class CTImage(object):
"""docstring for Hotel"""
def __init__(self, x_offset, y_offset, z_offset, x_ElementSpacing, y_ElementSpacing, z_ElementSpacing):
self.x_offset = x_offset
self.y_offset = y_offset
self.z_offset = z_offset
self.x_ElementSpacing = x_ElementSpacing
self.y_ElementSpacing = y_ElementSpacing
self.z_ElementSpacing = z_ElementSpacing2.2.2 生成.xml文件
VOC数据集格式介绍:
Pascal Voc数据集详细分析_持久决心的博客-CSDN博客_pascal voc
VOC具有很多属性,但是我们这里在做肺结节检测的时候,有些属性是不需要的,我们需要的属性只有物体的名词以及坐标等。下面就是生成的一个.xml标签。
<annotation>
<folder>VOC</folder>
<filename>0003.png</filename>
<size>
<width>512</width>
<height>512</height>
<depth>3</depth>
</size>
<object>
<name>nodule</name>
<bndbox>
<xmin>381</xmin>
<ymin>266</ymin>
<xmax>389</xmax>
<ymax>389</ymax>
</bndbox>
</object>
</annotation>
生成.xml的函数为(Luna16的CT图切割之后都是3*512*512的):
def beatau(e,level=0):
if len(e)>0:
e.text='\n'+'\t'*(level+1)
for child in e:
beatau(child,level+1)
child.tail=child.tail[:-1]
e.tail='\n' + '\t'*level
def ToXml(name, x, y, w, h):
root = Element('annotation')#根节点
erow1 = Element('folder')#节点1
erow1.text= "VOC"
erow2 = Element('filename')#节点2
erow2.text= str(name)
erow3 = Element('size')#节点3
erow31 = Element('width')
erow31.text = "512"
erow32 = Element('height')
erow32.text = "512"
erow33 = Element('depth')
erow33.text = "3"
erow3.append(erow31)
erow3.append(erow32)
erow3.append(erow33)
erow4 = Element('object')
erow41 = Element('name')
erow41.text = 'nodule'
erow42 = Element('bndbox')
erow4.append(erow41)
erow4.append(erow42)
erow421 = Element('xmin')
erow421.text = str(x - np.round(w/2).astype(int))
erow422 = Element('ymin')
erow422.text = str(y - np.round(h/2).astype(int))
erow423 = Element('xmax')
erow423.text = str(x + np.round(w/2).astype(int))
erow424 = Element('ymax')
erow424.text = str(y + np.round(h/2).astype(int))
erow42.append(erow421)
erow42.append(erow422)
erow42.append(erow423)
erow42.append(erow424)
root.append(erow1)
root.append(erow2)
root.append(erow3)
root.append(erow4)
beatau(root)
return ElementTree(root)2.2.3 生成训练集、测试集、验证集
Luna16一共有1186个肺结节,我使用其中70%为训练集,15%为测试集,15%为验证。首先要生成train.txt, test.txt, val.txt。
import random
import numpy as np
getTrain = []
getTest = []
getVal = []
arrTrain = []
arrTest = []
arrVal = []
arr = np.arange(1,1186)
#训练集
num = 0
for i in np.random.permutation(arr):
if num >829:
break
arrTrain.append(i)
num = num + 1
getTrain=np.array(arrTrain)
getTrain.sort()
f=open('train.txt','w')
for i in getTrain:
f.write('{:04d}\n'.format(i))
f.close()
#测试集
num = 0
for i in np.random.permutation(arr):
if num >178:
break
arrTest.append(i)
num = num + 1
getTest=np.array(arrTest)
getTest.sort()
f=open('test.txt','w')
for i in getTest:
f.write('{:04d}\n'.format(i))
f.close()
#验证集
num = 0
for i in np.random.permutation(arr):
if num >178:
break
arrVal.append(i)
num = num + 1
getVal=np.array(arrVal)
getVal.sort()
f=open('val.txt','w')
for i in getVal:
f.write('{:04d}\n'.format(i))
f.close()
生成之后.txt就有对应的图片ID。


下一步就有yolov3自带的voc_label.py进行解析,生成相应的label。为了使用,下列代码的difficult默认为0了。
import xml.etree.ElementTree as ET
import pickle
import os
from os import listdir, getcwd
from os.path import join
sets=[('', 'train'), ('', 'val'), ('', 'test')]
classes = ["nodule"]
def convert(size, box):
dw = 1./(size[0])
dh = 1./(size[1])
x = (box[0] + box[1])/2.0 - 1
y = (box[2] + box[3])/2.0 - 1
w = box[1] - box[0]
h = box[3] - box[2]
x = x*dw
w = w*dw
y = y*dh
h = h*dh
return (x,y,w,h)
def convert_annotation(year, image_id):
in_file = open('E:/LUNA/VOCdevkit/VOC%s/Annotations/%s.xml'%(year, image_id))
out_file = open('E:/LUNA/VOCdevkit/VOC%s/labels/%s.txt'%(year, image_id), 'w')
tree=ET.parse(in_file)
root = tree.getroot()
size = root.find('size')
w = int(size.find('width').text)
h = int(size.find('height').text)
for obj in root.iter('object'):
difficult = 0
cls = obj.find('name').text
if cls not in classes or int(difficult)==1:
continue
cls_id = classes.index(cls)
xmlbox = obj.find('bndbox')
b = (float(xmlbox.find('xmin').text), float(xmlbox.find('xmax').text), float(xmlbox.find('ymin').text), float(xmlbox.find('ymax').text))
bb = convert((w,h), b)
out_file.write(str(cls_id) + " " + " ".join([str(a) for a in bb]) + '\n')
wd = getcwd()
for year, image_set in sets:
if not os.path.exists('VOCdevkit/VOC%s/labels/'%(year)):
os.makedirs('VOCdevkit/VOC%s/labels/'%(year))
image_ids = open('VOCdevkit/VOC%s/ImageSets/Main/%s.txt'%(year, image_set)).read().strip().split()
list_file = open('%s_%s.txt'%(year, image_set), 'w')
for image_id in image_ids:
list_file.write('%s/VOCdevkit/VOC%s/JPEGImages/%s.png\n'%(wd, year, image_id))
convert_annotation(year, image_id)
list_file.close()
os.system("cat 2007_train.txt 2007_val.txt 2012_train.txt 2012_val.txt > train.txt")
os.system("cat 2007_train.txt 2007_val.txt 2007_test.txt 2012_train.txt 2012_val.txt > train.all.txt")
这一步完成之后可以看到根目录下有以下文件:

打开后,里面包含的是路径:

对应训练文件的Labels也出现了:


2.2.4 修改anchor size
anchor size是需要根据自己的数据集进行修改的,在这里我没有修改,想要修改的同学可以参考以下链接:
YOLO-v3模型参数anchor设置_m_buddy的博客-CSDN博客_anchor设置
YOLOv3使用笔记——Kmeans聚类计算anchor boxes_Gotta-C的博客-CSDN博客_yolov3聚类
3. 检测效果预览
3.1 训练
在检测之前,需要对生成的VOC格式的Luna16进行训练。在进行这部之前,一定检查第二部分生成的:.xml, .jpgs, .txt是否存在。下一步则是要改yolov3的网络结构。

每一个yolo层的class修改为1(这里只检测肺结节),filters修改为18(3*(1+5))。同时.data和.names均进行修改。

待.cfg,.data, .names文件修改完毕后,则可以开始训练。训练过程(训练中,待更新,这里使用的是C版本的yolov3):

3.2 训练结果显示
还在训练中,训练完了更新。
训练了大概3天,迭代了48000次,loss值约0.03(GTX1080)。
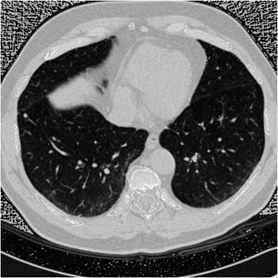
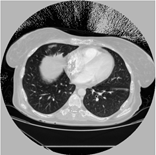
原图:
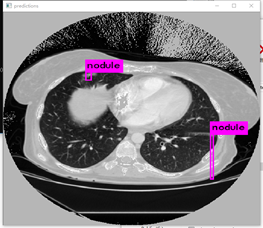
预测:

标注图(根据标注生成):

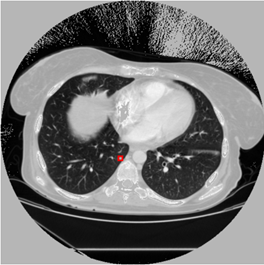
可以发现yolov3检测肺结节还是有点吃力的,存在较多的漏检、误检。原因可能是:1.迭代次数还不够,模型没有完全收敛。2.需要对数据集进行聚类分析,修改anchors的尺寸和比例。
3.3 存在的问题
1. 数据不平衡问题。
在本次训练的时候,只使用了正例(有肺结节)的例子,其实还有很多类似肺结节但不是肺结节的例子。

比如上图是根据标注生成的图片,我们看到肺结节很小,但其实图像左下方还有很多类似肺结节的结点,但却不是肺结节。这是因为我只使用正例(是真实肺结节)的标注。

class=1表示有肺结节。但如果把全部类似肺结节进行考虑的话,其实应该分两步做。第一步:检测出类似肺结节区域。第二部:从类似肺结节区域中得到真实肺结节。像这种正负样布不均衡问题,应该可以考虑focal loss优化。
2.数据结构信息不完整
这点主要体现在CT图像从3维转换到2维,损失了结构信息。Luna16数据集一共112GB,经过处理成二维之后,只有247MB。下图均是根据标注生成的图片(labelImage)。我们发现第一张图只有右下角是肺结节,但左上角不是。第二张图左上角是肺结节,但右下角不是肺结节,而两张图却及其类似。这是因为CT图像是三维的,我是按Z轴方向进行切割,也是说标注肺结节的Z轴是不同的,但下面两张图的Z轴坐标却非常的相近,所以会造成这个情况。我认为应该处理的方法,给每个结节赋予一个概率值,当大于某个概率值才会显示。


以上,简单的把yolov3用于实际数据集(Luna16)处理。
把Luna转换成VOC格式的脚本已上传:
https://download.csdn.net/download/qq_24739717/11422858
里面包含了两个脚本,使用到的所有函数已在第二部分讲解。
这两个脚本不是最新的。
Luna16数据集转VOC数据集&肺实质分割&生成,Mat.zip_luna16分割-深度学习代码类资源-CSDN下载
更新:2019.9.10
对数据集进行肺实质分割。

1.标准化数据,查看数值分布:

2.找出肺部附近的平均像素值,对洗掉的图像进行重新正规化。使用Kmeans分离前景(不透明组织)和背景(透明组织,即肺)。这样做只在图像的中心,尽量避免非组织部分的图像。
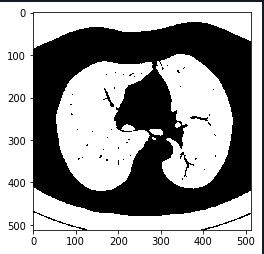
3.使用腐蚀和膨胀。可参考https://www.jianshu.com/p/05ef50ac89ac。腐蚀操作对去除某些区域的颗粒状很有帮助;然后使用大的膨胀去吞噬肺部区域的血管,,并且去除黑色噪声,特别是不透明射线造成的黑色肺部区域。

4.提取肺部mask。选取CT图像的中间部分,是为了把四周区域(床板)给剔除,接下来计算图像的连通域,获取较大的连通域为肺部mask。对该mask再做一次大的膨胀来填充和取出肺部mask。这个时候边缘可能会不整齐,可以使用腐蚀来优化边缘,但同时有可能会造成肺部空洞的风险(因为前面的膨胀操作并不一定能完全填充空洞,会由于腐蚀的作用再次放大)。

参考:https://github.com/booz-allen-hamilton/DSB3Tutorial
更新:2019.9.14
使用分割后的数据进行训练
训练了大概四天,确实比没有分割前有较大提升。使用肺实质分割之后,图片背景更干净了,对检测效果有很大的帮助。不过我忘了使用kMeans对数据集聚类,修改anchor size了,不然IOU应该还有更大的提升。
下图左边是预测,右边是根据标注生成的ground truth。





更新:2019.9.23
修改anchor size后重新训练
对数据集进行kmean是聚类,修改anchor size,这里需要主要几点:
1.kmean聚类在yolo中是选择了9个聚类中心,但是由于肺结节数据集的特殊性,其聚类中心基本上不可能为9个,使用9个聚类中心进行聚类会出现nan或者一直运行的情况。
2.数据集的bounding boxes占整幅图片的比例很小,当归一化到0~1之间之后,数值大部分只有0.01x,会加大运算量,甚至无法收敛,这里我先把其尺寸全部放到10倍,在聚类完毕之后再除以10。
3.根据标注生成的标注图片,不能拿发现肺结节的bounding boxes基本上为1:1,同时考虑到yolov3使用了3个特征尺度进行预测,所这里选择聚类中心为3。
多次聚类求得比较好的anchor size(使用3个聚类中心有时仍然可能无法收敛或者出现nan):
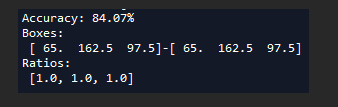
这个之84.07%已经很高了,比voc 67%左右高出不少。
当然也测试了一下其他聚类中心(1个或者2个):
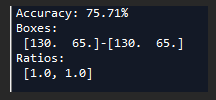
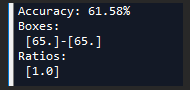
但都没有使用3个聚类中心的好。
训练:

检测效果:


更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容









所有评论(0)