顶刊(Nature)中公开分享了数据分析Code和绘图代码 | Condense-seq
原文:Native nucleosomes intrinsically encode genome organization principles文章链接:https://www.nature.com/articles/s41586-025-08971-7#code-availability期刊:NatureIF:50.5发表时间:2025年5月7日Python。
原文链接:顶刊(Nature)中公开分享了数据分析Code和绘图代码 | Condense-seq
前言
- 原文:Native nucleosomes intrinsically encode genome organization principles
- 文章链接:https://www.nature.com/articles/s41586-025-08971-7#code-availability
- 期刊:Nature
- IF:50.5
- 发表时间:2025年5月7日

文章使用分析数据和绘图语言:Python
原文链接:顶刊(Nature)中公开分享了数据分析Code和绘图代码 | Condense-seq
摘要
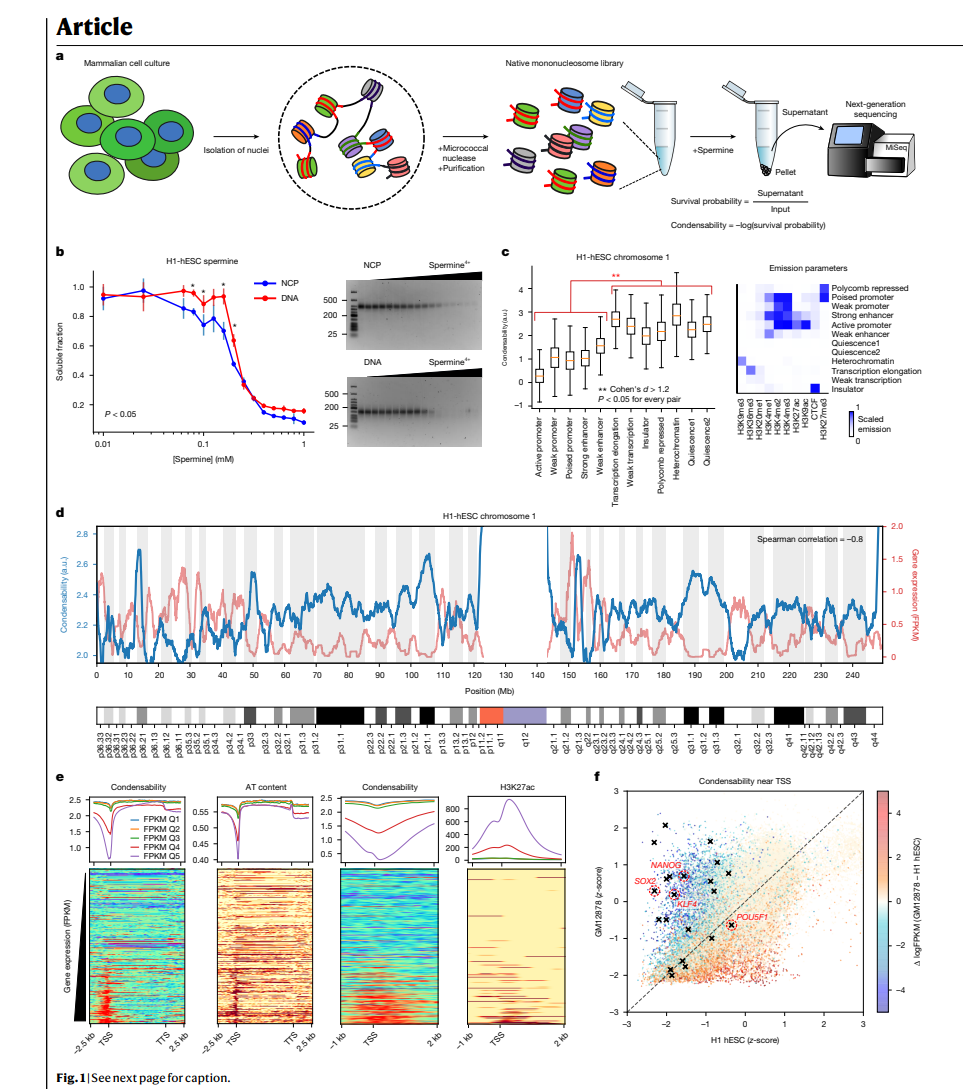
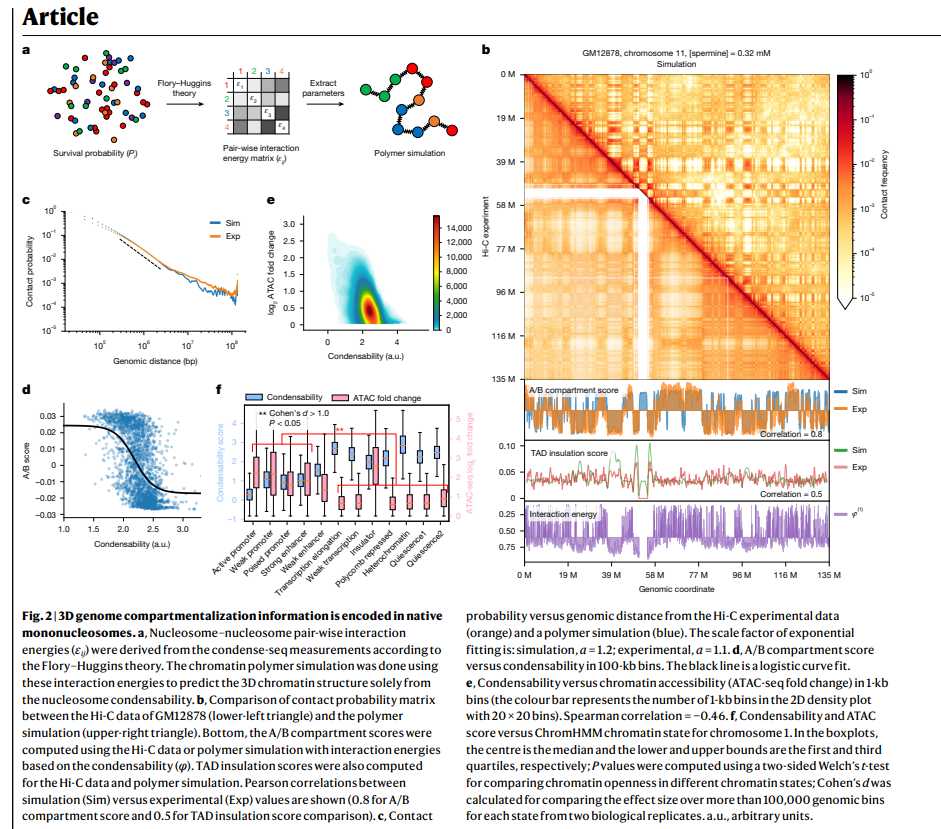
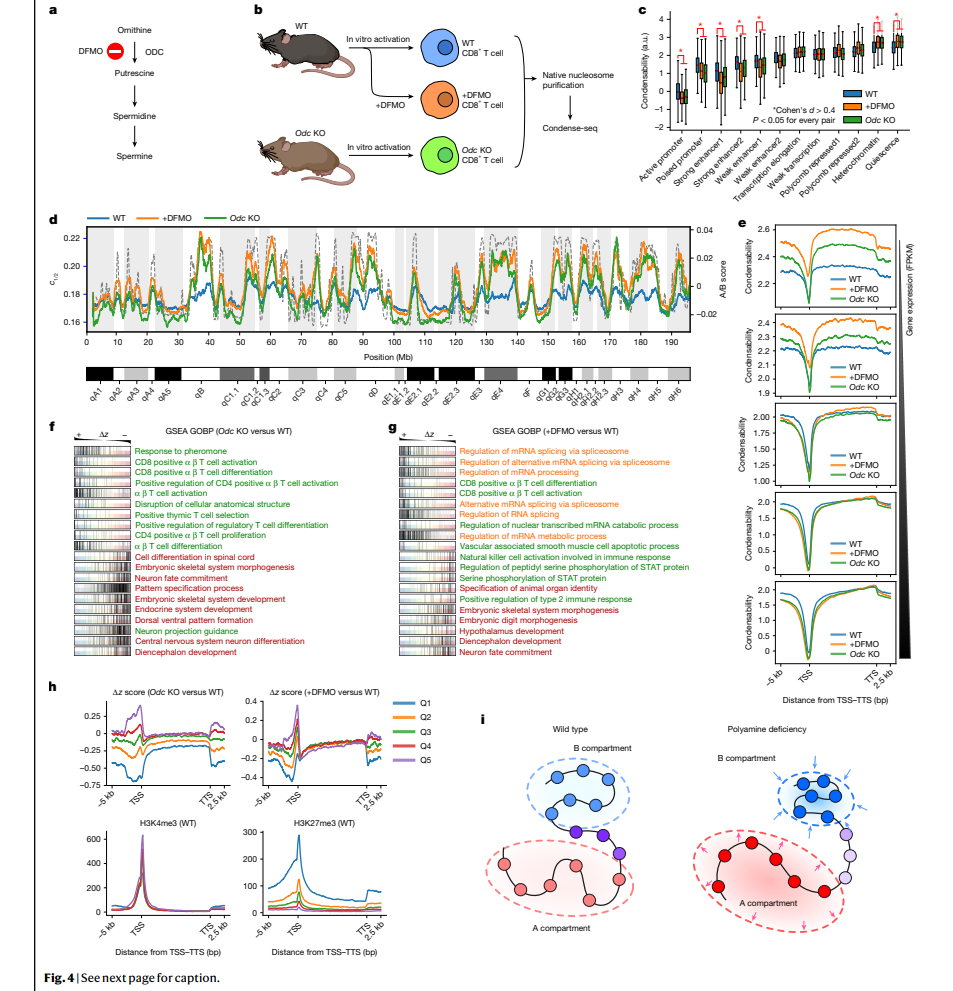
真核生物基因组被包裹在由 147 个碱基对组成的核小体中,围绕着组蛋白核心,并分别组织成真染色质和异染色质,分别对应于 A 和 B 区室 1,2。在这里,我们研究了单个核小体是否包含足够的信息来将 3D 基因组组织成区室,例如,在其生物物理特性方面。我们纯化了天然单核小体,使其具有高度的单分散性,并使用生理浓度的聚胺来确定它们的凝聚力。已知分配到 A 区室的染色体区域具有低凝聚力,而那些用于 B 区室的染色体区域具有高凝聚力。以凝聚力作为唯一输入的染色质聚合物模拟,在没有任何反式因子的情况下,再现了 A/B 区室。凝聚力也与基因表达强烈反相关,特别是在启动子附近,并且以细胞类型依赖的方式。因此,单核小体具有与基因开启或关闭相关的生物物理特性。与遗传和表观遗传特征的比较表明,核小体的凝聚性是一种突现特性,为投射高维细胞染色质状态提供了一个自然轴。使用各种凝聚剂或组蛋白修饰和突变进行分析表明,编码在核小体中的基因组组织原理在本质上主要是静电性的。小鼠 T 细胞中的聚胺耗竭,由于敲除或抑制鸟嘌呤脱羧酶而导致高极化的凝聚性,表明当细胞无法依赖聚胺将核小体的生物物理特性转化为三维基因组组织时,它们会强化凝聚性对比,这可能解释了在聚胺缺乏时观察到的功能障碍。
在公众号后台回复关键词:20250612,即可获得对应的Code。
About Condense-seq
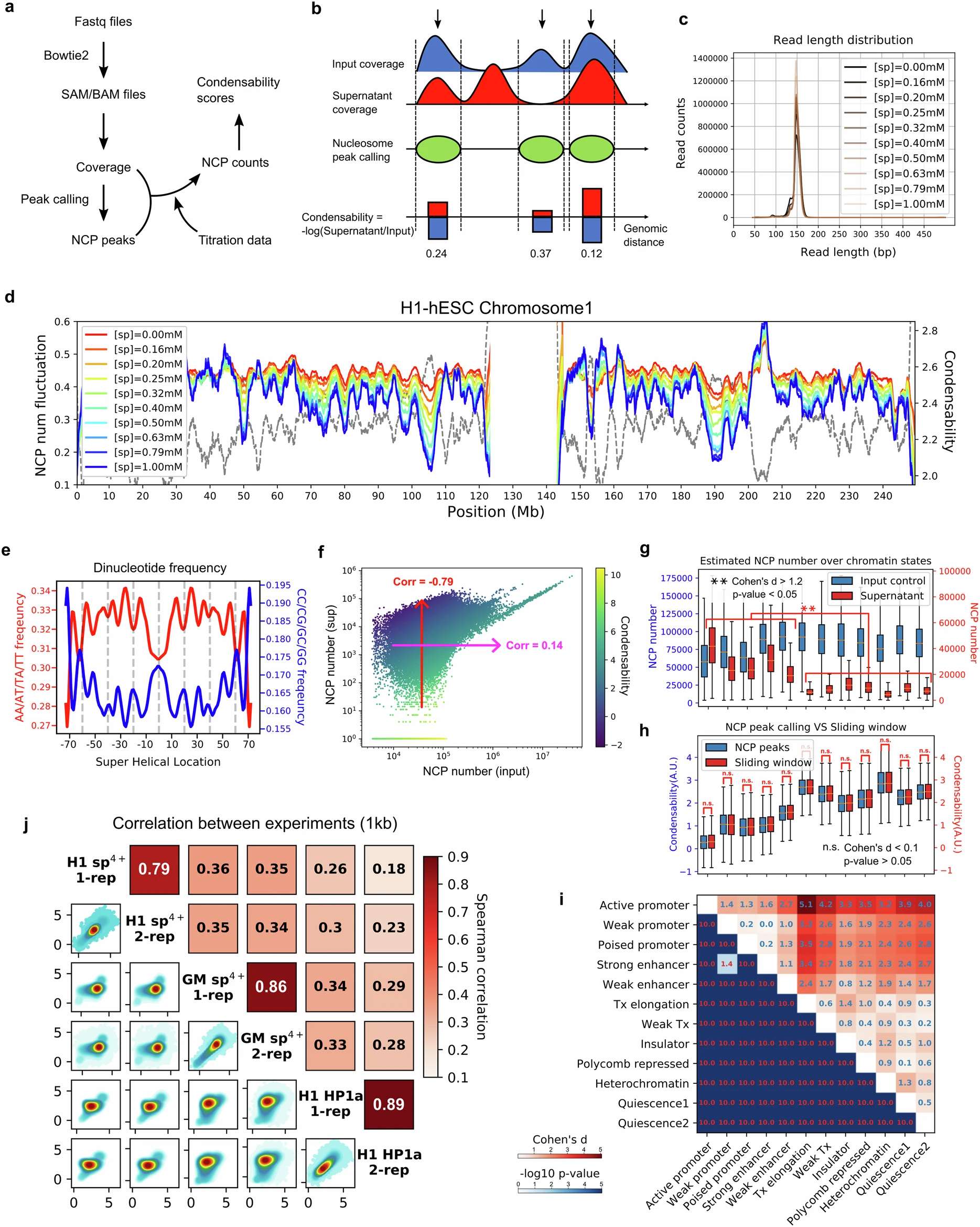
Condense-seq是一种高通量技术,旨在直接测量天然核小体在全基因组范围内的生物物理特性,具有单核小体分辨率。在这种方法中,天然单核小体从细胞中纯化,并使用各种冷凝剂在体外冷凝,包括聚胺、多聚化和异染色质蛋白。通过测序冷凝液的上清液相并与输入进行比较,可以确定单个基因组核小体的冷凝倾向或 “冷凝性”。我们的研究结果表明,天然核小体的冷凝性阐明了驱动真核基因组组织的生物物理原理。
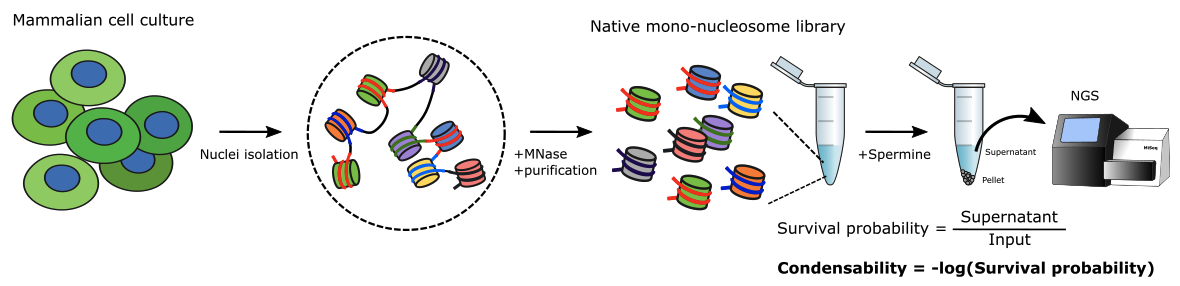
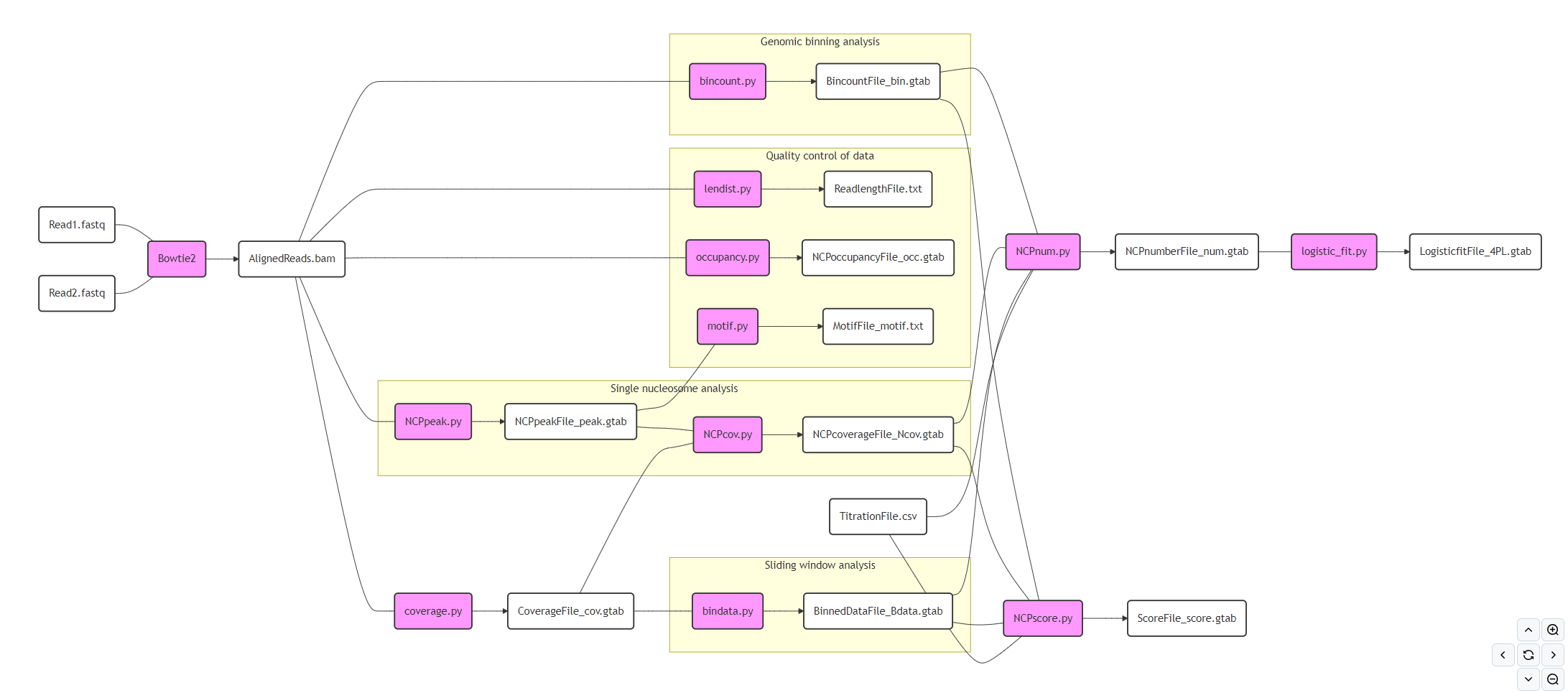
网页:https://github.com/spark159/condense-seq/tree/master
作者公开分享了对应的Python代码,供大家学习。
网页:https://zenodo.org/records/15036149
文章图形

若我们的教程对你有所帮助,请点赞+收藏+转发,大家的支持是我们更新的动力!!
2024已离你我而去,2025加油!!
往期部分文章
1. 最全WGCNA教程(替换数据即可出全部结果与图形)
推荐大家购买最新的教程,若是已经购买以前WGNCA教程的同学,可以在对应教程留言,即可获得最新的教程。(注:此教程也仅基于自己理解,不仅局限于此,难免有不恰当地方,请结合自己需求,进行改动。)
2. 精美图形绘制教程
3. 转录组分析教程
4. 转录组下游分析
BioinfoR生信筆記 ,注于分享生物信息学相关知识和R语言绘图教程。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容










所有评论(0)